-
Články
Top novinky
Reklama- Vzdělávání
- Časopisy
Top články
Nové číslo
- Témata
Top novinky
Reklama- Kongresy
- Videa
- Podcasty
Nové podcasty
Reklama- Kariéra
Doporučené pozice
Reklama- Praxe
Top novinky
ReklamaSekvenování nové generace – nástroj vědy či rutinní patologie?
Next generation sequencing – a science tool or routine pathology?
Molecular assays for translocation detection in different tumors have gradually been incorporated into routine diagnostics. However, conventional methods such as fluorescence in situ hybridization (FISH) and reverse transcriptase-PCR come with several drawbacks. Next-generation sequencing (NGS) can provide in-depth detection of numerous gene alterations. The anchored multiplex PCR assay proved to be a fast and easy-to-analyze approach for routine diagnostics laboratories. Next-generation sequencing-based anchored multiplex PCR technique (Archer FusionPlex Panels) is beneficial in both diagnosis for patient care and in identification of a novel fusion breakpoint in tumors.
NGS is useful in identifying targetable molecular changes (point mutations, fusion genes, etc.) in tumors that can serve as a rationale for inclusion of patients with advanced disease in ongoing clinical trials and allow for better risk stratification.
Keywords:
next generation sequencing (NGS) – somatic mutations – Fusion genes – targeted therapy
Autoři: Lenka Krsková 1; Markéta Kalinová 1; Tereza Němečková 1; Barbora Šípalová 1; Marcela Mrhalová 1; Miroslav Koblížek 1; Jan Balko 1; Jaromír Háček 1; Daniela Nováková-Kodetová 1; Aleš Vícha 2; Petr Brož 2,3; Michal Zápotocký 2; Edita Kabíčková 2; Josef Zámečník 1
Působiště autorů: Ústav patologie a molekulární medicíny, 2. LF UK a FN Motol, Praha 1; Klinika dětské hematologie a onkologie, 2. LF UK a FN Motol, Praha 2; Bioxsys s. r. o., Ústí nad Labem 3
Vyšlo v časopise: Čes.-slov. Patol., 57, 2021, No. 3, p. 136-143
Kategorie: Přehledový článek
Souhrn
Molekulární metody pro detekci translokací byly postupně začleněny do rutinní diagnostiky nádorových onemocnění. Konvenční metody, jako je fluorescenční in situ hybridizace (FISH) a reverzně transkriptázová-PCR, však mají i některé nevýhody. Sekvenování nové generace (NGS) může poskytnout citlivou detekci alterací mnoha genů.
RNA NGS na principu Anchored multiplex PCR se ukázalo jako rychlý a snadno analyzovatelný přístup pro laboratoře rutinní diagnostiky. Archer FusionPlex panely jsou přínosné jak v diagnostice nádorů, tak v identifikaci nových fúzních genů.
NGS je užitečný nástroj při identifikaci cílitelných molekulárních změn (bodové mutace, fúzní geny atd.). U pacientů s pokročilým onemocněním může NGS napomoci k zařazení těchto pacientů k léčbě na základě stanovení rizikových markerů (risk udapted therapy) a/nebo při průkazu léčebného cíle k cílené léčbě.
Klíčová slova:
sekvenování nové generace (NGS) – somatické mutace – fúzní geny – cílená terapie
Sekvenování nové generace (NGS next-generation sequencing) nebo také masivně paralelní sekvenování (MPS) je metodika, která přináší obrovský rozvoj v oblasti personalizované medicíny. NGS je na rozdíl od klasické Sangerovy sekvenační metody schopno detekovat přidávání bází jednu po druhé a zároveň sekvenovat tisíce až miliony rozdílných molekul DNA či cDNA (komplementární DNA převedená z RNA) najednou během jednoho sekvenačního běhu.
Metoda NGS je již nedílnou součástí řady pracovišť patologie. Jde především o univerzitní zařízení a prediktivní laboratoře, kde se stala rutinní metodou vyšetřování somatických změn v nádorových buňkách. Počátek využití somatického NGS v rámci terapeuticko-indikační patologie je spjat se začátkem testování somatických mutací v genech BRCA1 a BRCA2 díky objevu cílené terapie PARP inhibitory u pacientek s ovariálním karcinomem. Pokud bychom geny BRCA1/2 testovali klasickou Sangerovou sekvenační metodou, trvalo by nám to při počtu více než 20 exonů každého z genů minimálně čtvrt roku. Mutační analýza v rámci indikace k terapii však musí být rychlá, přesná a musí splňovat mezinárodní kritéria.
NGS technologie však nenachází využití jen v rámci prediktivního testování, ale napomáhá i v diagnostice onemocnění, určení prognózy, k detekci minimální reziduální/diseminované nemoci (MRN/MDN) (1), či umožňuje definovat jednotlivé subklony ať už nádorových buněk nebo buněk imunitního systému, případně nádorového mikroprostředí. Při testování somatických mutací je vždy důležitá citlivost metody. U technologie NGS dosahujeme požadované citlivosti pomocí vysokého počtu čtení.
Molekulární patologie využívá primárně vzorky nádorových tkání, v menší míře se vyšetřuje i tzv. tekutá biopsie k detekci volné nádorové DNA (ctDNA), případně vzorky kostní dřeně (KD)/ periferní krve (PK) k monitoraci MRN/MDN.
Tekutá biopsie znamená odběr PK u pacientů se solidními nádory, ve které analyzujeme přítomnost ctDNA v krevní plazmě. Mechanizmus uvolňování ctDNA do krve je spjatý s apoptózou a nekrózou nádorových buněk a zčásti jde i o aktivní sekreci. Vyšetření v rámci identifikace molekulárních změn nádoru se standardně provádí z tkáně histologicky verifikovaného primárního nádoru a/nebo z metastáz. V některých případech však není tato tkáň dostupná. Vzhledem k tomu, že odběr PK je nenáročný, s minimální zátěží pro pacienta a lze ho provádět opakovaně, má tato metoda velký klinický význam. Metodika detekce ctDNA v plazmě pacientů se solidními nádory má význam pro diagnostiku a monitoraci nádorového onemocnění, predikci léčebné odpovědi, časnou diagnózu relapsu nebo vznikající rezistence na léčbu (příkladem je detekce rezistentní mutace T790M v genu EGFR), i identifikaci nových vhodných terapeutických cílů (2). Nicméně frakce ctDNA kolísá v závislosti na typu nádoru a stádiu onemocnění, přičemž změny prokázané v ctDNA se nemusí vždy zcela shodovat se změnami prokázanými v nádoru. Vyšetření ctDNA ale nenahrazuje bioptické vyšetření tkáně (3,4).
Princip metodiky NGS je, že molekuly nukleových kyselin jsou nejprve fragmentovány (nejčastěji enzymaticky) na relativně krátké části a na jejich konce je připojen adaptér (případně „molekulární barkód“), což je velmi krátká molekula DNA o známém sekvenčním složení. Slouží k následnému navázání sekvenovaného úseku nukleové kyseliny na pevný povrch. Přidávají se ještě tzv. indexy (krátké sekvenčně známé úseky), které rozlišují jednotlivé vzorky sekvenovaných pacientů mezi sebou (vzorky pacientů jsou v konečných krocích přípravy knihovny smíchány do jedné zkumavky). Takto upravená nukleová kyselina se nazývá sekvenační knihovna. V případě nejrozšířenější Illumina technologie dochází k uchycení molekul nukleové kyseliny na povrch pomocí adaptéru, každý řetězec DNA či cDNA je namnožen a vzniká klastr identických molekul koncentrovaných v jednom místě, což umožní zesílení výsledného signálu při sekvenování daného úseku nukleové kyseliny. Sekvenační proces poté probíhá dle použité platformy.
Využívají se různé NGS sekvenátory (např. Illumina: MiniSeq, MiSeq, NextSeq, HiSeq, NovaSeq), které umožňují např. sekvenaci celého genomu, exomu, transkriptomu, případně amplikonu v několika dnech (5). V případě detekce somatických mutací využíváme nejčastěji cíleného („target enrichement“) sekvenování, které umožňuje získat informaci o přesně definovaných genech či jejich částech. Pro somatické NGS testování je doporučováno minimální pokrytí 300 čtení („coverage“) jednotlivých částí nukleových kyselin (genů). Se zvyšujícím se množstvím pokrytí každé části genů zvyšujeme možnost záchytu případných subklonů. Další variantou NGS je např. analýza metylace DNA daného genomu („methylom“).
Analýza NGS dat se přenáší do oblasti bioinformatiky, protože ve výsledku získáváme obrovské množství dat (sekvencí), které je potřeba statisticky zpracovat, zarovnat k referenčnímu genomu a následně správně interpretovat nalezené varianty. Lidský genom obsahuje 3,2x109 bází, kdy v případě analýzy celého genomu při pokrytí 30x (tzn., že každá část DNA je čtena minimálně 30x), získáváme pro jednoho pacienta 30 krát 3,2x109 dat.
K sekundární a terciální analýze NGS dat jsou k dispozici různé databáze (NCBI, Blast, Igblast, Cosmic, Ensembl, ClinVar, Poly - Phen, Sift, Varsome, NextGENe, QCI Interpret atd.), které lze využít pro analýzu získaných sekvencí a nalezených změn v DNA (RNA/cDNA). Tyto databáze se neustále aktualizují. V případě analýzy celých genomů je proces počítačového skládání výsledných sekvencí (sekvenační technologie umožňuje sekvenaci DNA o délce několik stovek bází) velmi náročný.
Molekulární patologie vyšetřuje vzorky nádorové tkáně, proto je pro následnou analýzu nezbytně nutné znát kvalitu vyšetřované tkáně (množství nádorových buněk, tumor infiltrujících lymfocytů apod.). Nevýhodou detekce somatických mutací nádorů je hlavně heterogenita nádorových buněk a identifikace změn (variant), které nejsou dosud v on-line literatuře a databázích popsány. V potaz je nutné brát i z jakého materiálu probíhalo NGS vyšetření. DNA izolovaná z tkání fixovaných ve formolu a zalitých do parafinu bývá nekvalitní a může obsahovat fixační artefakty (hlavně tranzice C >T). Analýza NGS dat je složitý a zdlouhavý proces, který vyžaduje zkušeného bioinformatika.
NGS v diagnostice fúzních genů
S příchodem technologie NGS se otevřela nová éra detekce fúzních genů a jejich chimérických transkriptů.
Metody jako jsou RT-PCR nebo fluorescenční in situ hybridizace (FISH), jsou vždy cílené pouze na konkrétní fúzní gen, ale pomocí NGS můžeme naráz otestovat velké množství známých fúzních genů nebo najít fúzní geny zcela nové, které mohou být zapojeny do patogeneze určitých skupin nádorů a následně tak napomoci vyčlenit nové podskupiny nádorů definovaných určitým fúzním genem (např. Ewing-like sarkomy s fúzním genem CIC-DUX4 nebo BCOR-CCNB3) či skupinou fúzních genů (přestavby genu PLAG1 u lipoblastomů s fúzními geny HAS2-PLAG1, RAB2A-PLAG1, COL1A2-PLAG1 apod., nebo přestavby genu BRAF v patogenezi pilocytárních astrocytomů s fúzními geny KIAA1549-BRAF, GNAI-BRAF, BCAS1-BRAF, PRKAR2B-BRAF, apod.) (6,7).
Velmi rozšířená aplikace NGS je právě sekvenování RNA (cDNA), které se využívá pro kvantitativní i kvalitativní analýzu. Vyizolovná RNA je pomocí reverzní transkripce přepsána do cDNA a ta je následně sekvenována. Výhodou NGS na podkladě RNA je především možnost měření intenzity exprese jednotlivých genů, identifikace různých variant fúzních genů a detekce sestřihových variant genů (8). Metoda RNA sekvenování (RNAseq) je obecně velmi citlivá na kvalitu vyizolované nukleové kyseliny, takže fixace nádorové tkáně ve formolu a následné zpracování materiálu v podobě parafínových bloků často znehodnotí vstupní nukleovou kyselinu ve vzorku do té míry, že znemožní analýzu pomocí NGS. Pro analýzu potenciálních fúzních genů je pak možnou alternativou analýza pomocí cíleného (targeted) sekvenování, při kterém je amplifikována a sekvenována pouze RNA (cDNA) z přesně definovaných oblastí (9). V případě detekce fúzních genů lze využít kity pro detekci a lokalizaci fúzních partnerů řádově desítek genů zapojených do nádorové transformace. Menší počet analyzovaných genů umožňuje sekvenovat vzorky do velké hloubky a pracovat i s méně kvalitní vstupní RNA než je tomu v případě analýzy celého transkriptomu. V naší laboratoři s výhodou využíváme Archer FusionPlex panely, pomocí kterých jsme schopni analyzovat i vzorky často suboptimální kvality, jak tomu u vzorků izolovaných z parafínových bloků (FFPE ) občas bývá, a provádět tak i retrospektivní analýzy vybraných typů nádorů. Podstatou těchto kitů je amplifikace PCR produktu, která začíná v některém z exonů genů, které jsou zahrnuty v panelu, pomocí genově specifického primeru. Po iniciální elongaci prvního vlákna je přiligován/připojen linker, který je následně využit k amplifikaci PCR produktu. Reakce je multiplexová a např. v případě Archer FusionPlex Sarcoma kitu zahrnuje 127 exonů 26 genů zapojených v patogenezi sarkomů (10). Následně je výsledná cDNA knihovna sekvenována a analyzována komerčním programem. Ten dle zarovnávání k referenčnímu genomu umožní detekovat jak známé, tak neznámé fúze. Přidanou hodnotou těchto kitů je i možnost detekce SNV/Indel změn vybraných genů. Celá příprava knihovny a analýza trvá zhruba 5 dní. Zatímco konvenční PCR metody vyžadují znalost obou sekvencí ohraničující oblast našeho zájmu, „anchored“ PCR Archer kitu umožňuje detekci fúzního partnera i v případě, že známe nebo předpokládáme pouze jednoho z fúzních partnerů.
Jelikož se v panelu Archer FusionPlex Lung kitu vyskytují geny, které jsou zapojeny nejen do patogeneze nádorů plic, ale i např. do patogeneze nádorů CNS - low-grade gliomů (LGG) (např. BRAF, FGFR1-3, ALK, ROS1, NTRK1-3, apod.) a ALK pozitivního zánětlivého myofibroblastického tumoru (IMT ALK+) (fúze genu ALK) s výhodou jsme tento panel využili k detekci fúzních genů u LGG i IMT ALK+. U ependymomů a dalších nádorů CNS jsme pak využili FusionPlexOncology Research kit, který má širší záběr pokrytých genů (celkem 74 genů v panelu). Celkem jsme zatím vyšetřili 59 pacientů s nádory CNS. Fúzní gen jsme detekovali u 33 (56 %) z nich, u 3 vzorků byl materiál tak degradován, že neumožnil získání výsledku, u 9 vzorků jsme nalezli bodové patogenní mutace a u 2 pacientů onkogenní variantu genu BRAF. Z detekovaných 33 fúzních genů bylo 6 nových, dosud nepopsaných fúzních genů (obr. 1). Pomocí RNAseq se nám podařilo odhalit vzácné a nové fúze genů FGFR2/3, NTRK1/2/3, RAF1 a dále i ROS1 a ALK. U 25 pacientů s pediatrickým LGG byla zahájena léčba BRAF, MEK nebo NTRK inhibitorem v závislosti na typu alterace.
Obr. 1. Nově diagnostikovaný fúzní gen PAG-BRAF u dětské pacientky s low-grade gliomem. 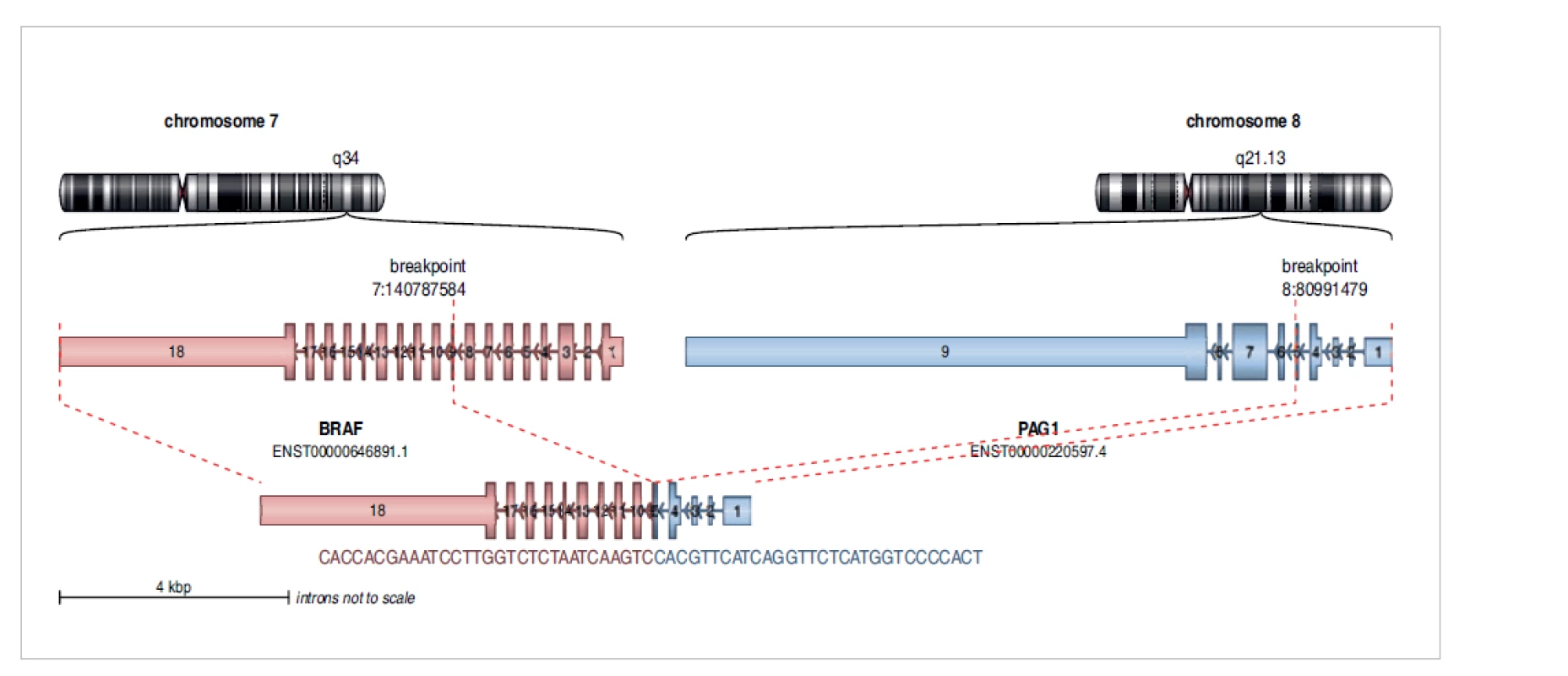
Dále jsme pomocí Archer FusionPlex Lung nebo Archer FusionPlex Sarcoma kitů vyšetřili 36 pacientů se sarkomy a detekovali 19 (53 %) fúzních genů, z nichž 3 dosud nebyly publikované. V rámci Archer FusionPlex Lymphoma kitu jsme zatím vyšetřili 14 pacientů s různými typy lymfomů, nejčastěji ALK pozitivní ALCL a nalezli jsme jak popsané (např. MYH9-ALK, ATIC-ALK), tak dosud nepopsané změny (CAPRIN-ALK).
Narůst využití technik k detekci fúzních genů na podkladě NGS nám umožňuje odhalit nové genové fúze a vylepšit tak naše chápání patogeneze celé řady nádorů. Nejenom, že nám NGS umožňuje potvrdit morfologickou diagnózu, ale poskytuje nám i informace důležité k personalizované terapii a k identifikaci účinných molekulárních biomarkerů.
V následujícím textu uvádíme ve vybraných kazuistikách, komplexní přístup k diagnostice s využitím RNA sekvenování nové generace.
Kazuistika 1 – sklerotizující epiteloidní fibrosarkom
K histologickému a molekulárnímu vyšetření jsme dostali v roce 2016 biopsii nádoru malé pánve od 5leté dívenky. V odebraném materiálu byla zachycena fibrózně změněná tkáň, která byla málo vaskularizovaná, místy byly v hyalinně změněném vazivu drobné areaktivní nekrózy. V okraji takto změněné tkáně se nacházelo výrazněji buněčné ložisko z větších polygonálních buněk s většími jádry, místy s jadérkem. Některé buňky byly větší s nepravidelnými jádry. Mezi buňkami byla patrná produkce kolagenu (obr. 2). Nádor byl mitoticky neaktivní.
Obr. 2. HE 5leté pacientky se sklerotizujícím epiteloidním fibrosarkomem. 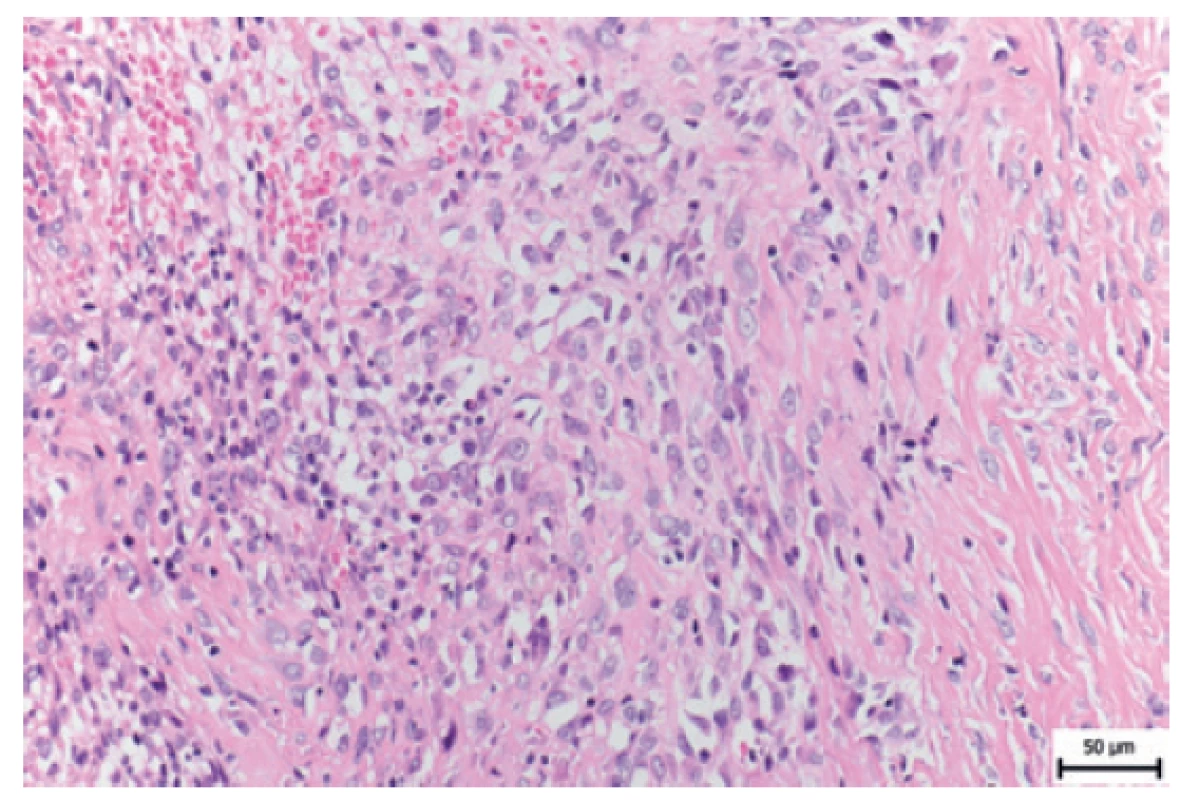
Pomocí imunohistochemického vyšetření byla prokázána pozitivita nádorových buněk v průkazu vimentinu a INI1. EMA, CD57 a S100 protein byly pouze ložiskově pozitivní; negativní byly nádorové buňky v průkazu D2-40, desminu, myogeninu i MyoD1, sarkomerického aktinu, cytokeratinů, GFAP. Markery CD31 i CD34 byly pozitivní pouze v cévách. Proliferační marker Ki-67 byl pozitivní ložiskově asi v 10 % buněk buněčné oblasti.
Pro suspekci ze sklerotizujícího epiteloidního fibrosarkomu bylo provedeno molekulární vyšetření, které pomocí RT-PCR neprokázalo fúzní geny FUS-CREB3L1 ani FUS-CREB3L2, které by svědčily pro toto onemocnění.
Nádor byl na základě výše uvedených vyšetření uzavřen jako pomalu rostoucí low-grade fibroblastická léze s potenciálně lokální agresivitou.
Nález byl dále konzultován na zahraničním pracovišti s obdobným závěrem.
V roce 2020 při progresi nádoru malé pánve bylo indikováno vyšetření pomocí Sekvenování nové generace s použitím Archer FusionPlex Sarcoma kitu. Pomocí tohoto vyšetření byl prokázán fúzní gen EWSR1-CREB3L1 (obr. 3a), který je vzácně popisován u diagnózy sklerotizujícího epiteloidního fibrosarkomu (SEF). Nález byl potvrzen pomocí RT-PCR a Sangerovy sekvenace (obr. 3b). Byla prokázána fúze exonu 11 genu EWSR1 s exonem 6 genu CREB3L1. Paralelně s vyšetřením pomocí NGS bylo provedeno vyšetření pomocí metylační SNP array (obr. 3c), které na základě metylačního profilu zařadilo jednoznačně nádor jako sklerotizující epiteloidní fibrosarkom.
Obr. 3. A: Diagnostikovaný fúzní gen EWSR1-CREB3L1 u dětské pacientky se sklerotizujícím epiteloidním fibrosarkomem. Analýza pomocí Arriba software https://github.com/suhrig/arriba. B: Sekvenační analýza fúzního produktu EWSR1-CREB3L1 pomocí Sangerovy sekvenace. C: Metylační analýza pomocí metylační SNParray. 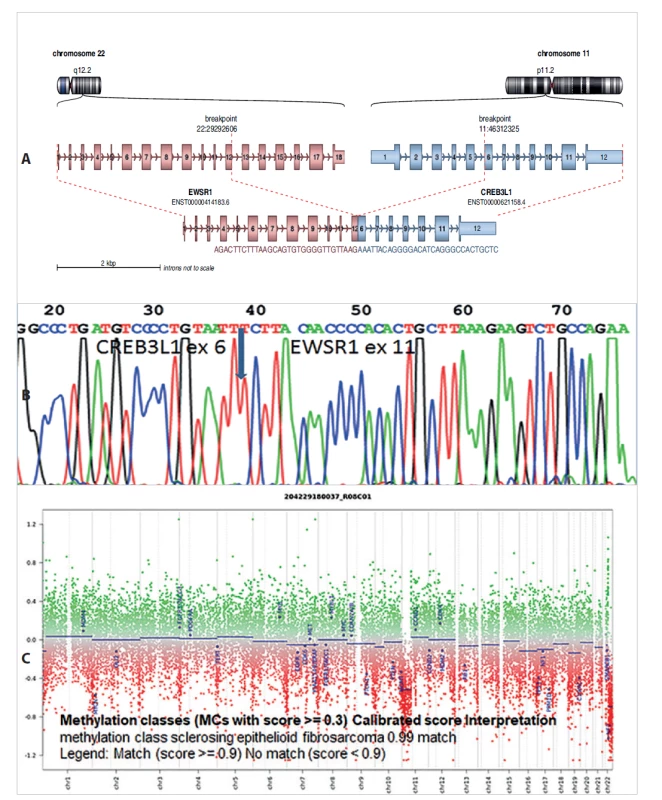
Na základě výše zmíněných vyšetření byl nádor jednoznačně zařazen jako SEF, který je v takto raném věku naprosto raritní.
Kazuistika 2 – vřetenobuněčný sarkom
K histologickému a molekulárnímu vyšetření jsme dostali biopsii od 10letého chlapce s nádorem břišní dutiny velikosti 190 mm x 140 mm x 65 mm (obr. 4a). V době diagnózy byla detekována solitární metastáza v pravé plíci.
Nádor byl tvořen vřetenitými buňkami, které byly fascikulárně uspořádané. Ložiskově byly buňky naznačeně rozetovitě uspořádané a tvořily částečně kalcifikovanou mezibuněčnou hmotu (obr. 4b). Struktury osteoidu nebyly přítomné.
Obr. 4. A: Magnetická rezonance 10letého chlapce s rozsáhlým sarkomem jaterní krajiny. B: HE 10letého pacienta s vřetenobuněčným sarkomem. 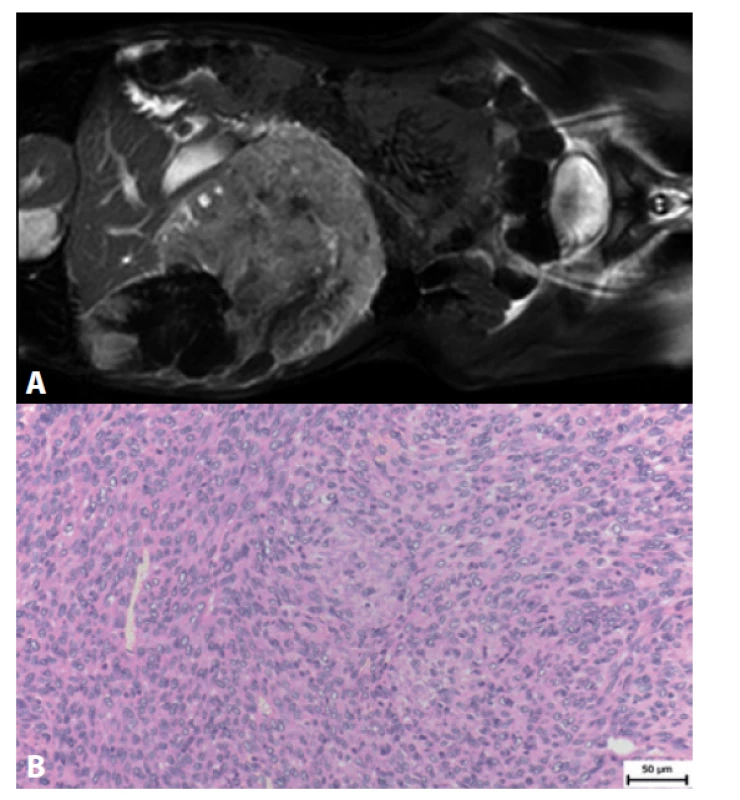
Pomocí imunohistochemického vyšetření byla prokázána pozitivita nádorových buněk v průkazu hadkosvalového i sarkomerického aktinu, vimentinu a CD99; negativní byly nádorové buňky v průkazu desminu, myogeninu i MyoD1, EMA, ALK, BCL2, GFAP, SOX10, CD57 i STAT6. Marker CD34 byl pozitivní pouze v cévách.
Pomocí metody fluorescenční in situ hybridizace nebyl prokázán zlom v oblasti genu EWSR1 a pomocí RT-PCR nebyl prokázán fúzní gen CIC/DUX4 ani BCOR/CCNB3, které by svědčily pro diagnózu tzv. „Ewing-like sarkomu“. Vzorek nádoru byl dále vyšetřen pomocí SNP array, kde bylo vzneseno podezření na možnou přítomnost fúzního genu s genem BRAF. Na základě tohoto vyšetření byl nádor vyšetřen pomocí RNA sekvenování nové generace s použitím Archer FusionPlex Lung panelu, který je schopný detekovat přestavby v genu BRAF. Pomocí NGS metody byl prokázán dosud nepopsaný fúzní gen MTAP/BRAF (obr. 5a). Na základě tohoto výsledku byly navrženy primery pro PCR a pomocí metody RT-PCR a přímého sekvenování byla potvrzena fúze exonu 7 genu MTAP s exonem 9 genu BRAF (obr. 5b).
Obr. 5. A: Nově diagnostikovaný fúzní gen MTAP-BRAF u dětského pacienta s vřetenobuněčným sarkomem. Analýza pomocí Arriba software https://github.com/suhrig/arriba. B: Sekvenační analýza fúzního produktu MTAP-BRAF pomocí Sangerovy sekvence. 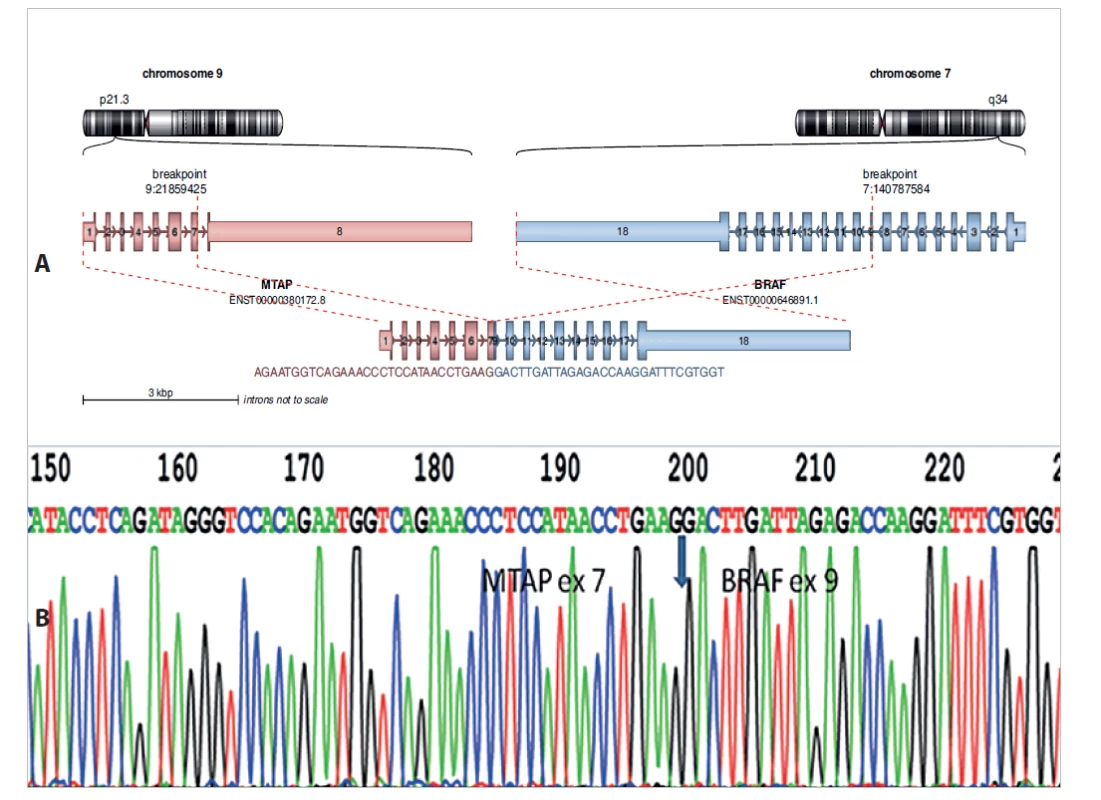
Na základě těchto vyšetření byl nádor uzavřen jako vřetenobuněčný nádor s MTAP/BRAF fúzním genem. Pro progresi onemocnění je v současné době pacient na základě průkazu BRAF fúzního genu léčen cílenou terapií pomocí MEK inhibitoru.
Kazuistika 3 – Ewingův sarkom
Další kazuistikou je případ 26letého pacienta s osteolytickým nádorem žebra. K histologickému vyšetření jsme obdrželi konzultační biopsii s jizevnatou fibrotizovanou tukovou tkání s úseky kosterní svaloviny, infiltrovanou v solidních čepech uspořádaným nádorem z kulatých až oválných a epiteloidních buněk s mírně atypickými kulatými až oválnými jádry a amfofilní cytoplazmou (obr. 6a). Ojediněle byla patrná mitotická aktivita.
Pomocí imunohistochemického vyšetření byla prokázána pozitivita nádorových buněk v průkazu cytokeratinu AE1/3 i cytokeratinu KL1 (obr. 6b), CD99, FLI-1, BCL2, CD56 a ložiskovou pozitivitou se synaptofyzinem a SATB2. Proliferační antigen Ki - 67 byl pozitivní cca v 15 % nádorových buněk. Negativní byly nádorové buňky v průkazu s napsinem A, p63, kalretininem, CK7, CD138, S100, CD45, NKX2.2 a CDX2.
Obr. 6. A: HE 26letého pacienta s Ewingovým sarkomem. B: Imunohistochemický průkaz cytokeratinu-KL1. 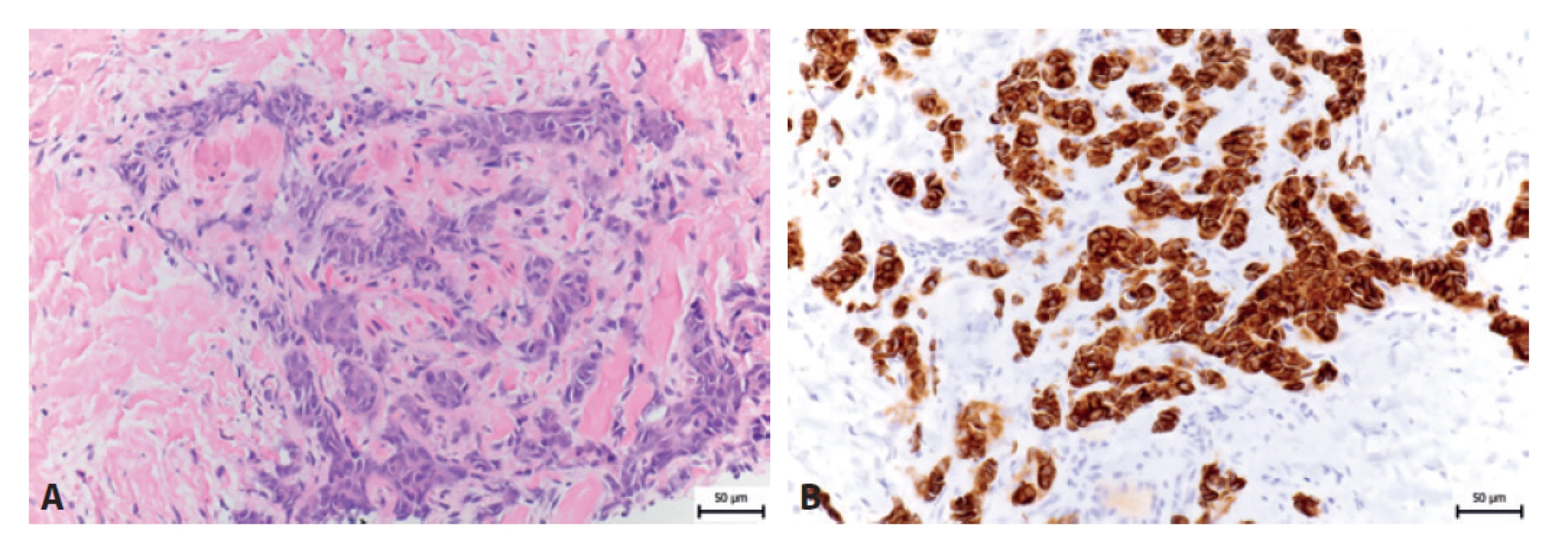
Nádor byl pomocí metody fluorescenční in situ hybridizace a pomocí RT-PCR vyšetřen na zlom genu SS18 a fúzní gen SS18-SSX s negativním výsledkem. Pomocí metody FISH byl prokázán zlom v oblasti genu EWSR1. Pro existenci velkého počtu fúzních partnerů genu EWSR1 byl vzorek dále vyšetřen pomocí metody NGS s použitím Archer FusionPlex Sarcoma kitu s průkazem fúzního genu EWSR1/ERG (obr. 7). Tento fúzní gen byl následně potvrzen i pomocí metody RT-PCR.
Obr. 7. Diagnostikovaný fúzní gen EWSR1-ERG u dospělého pacienta s diagnózou Ewingova sarkomu. Analýza pomocí Arriba software. 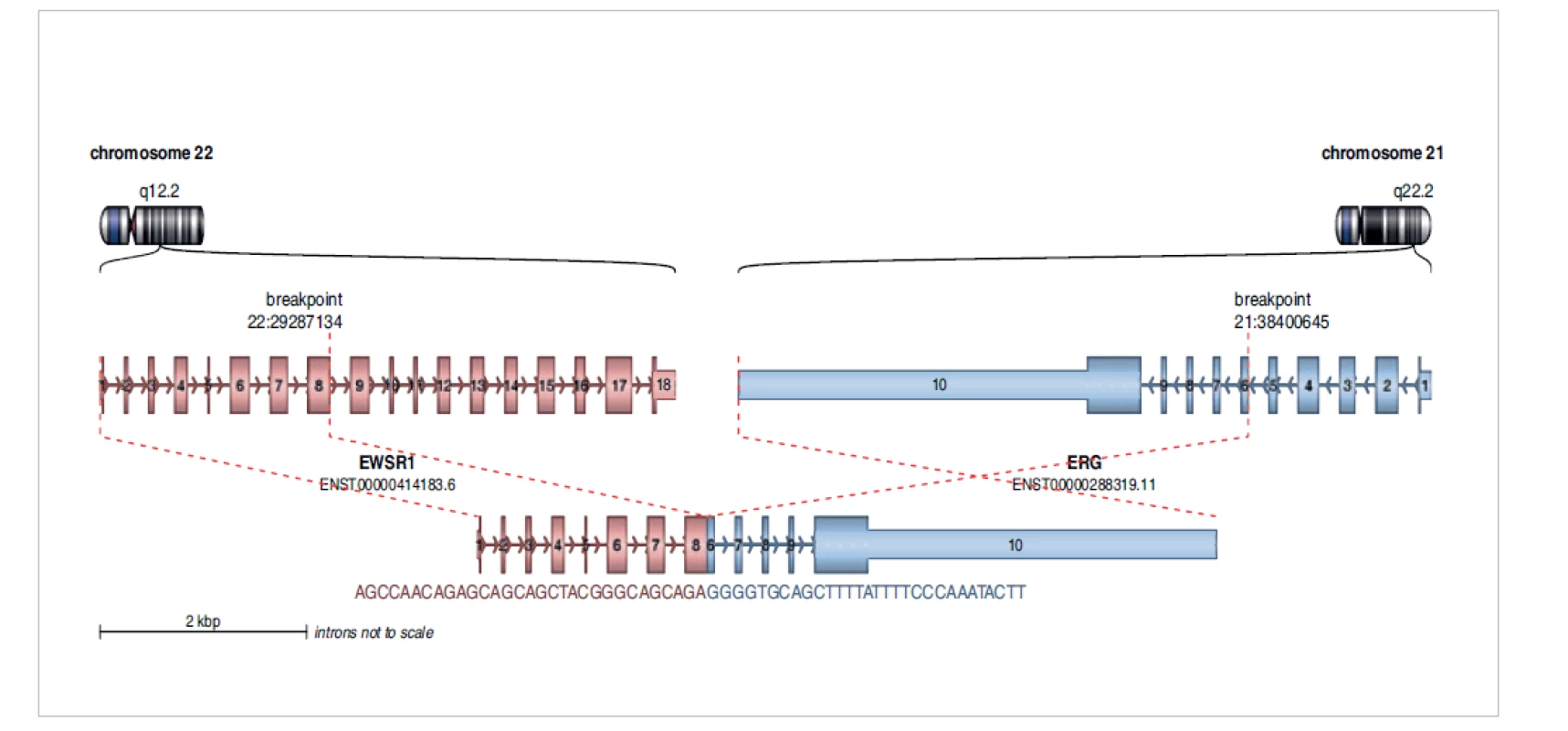
Kazuistika 4 – low-grade gliom
K histologickému i molekulárnímu vyšetření jsme dostali biopsii 8leté dívky s expanzí vycházející z oblasti optického chiasmatu s propagací suprachiasmaticky.
Z morfologického vyšetření byla zastižena proliferace z protáhlých gliových buněk (pozitivní při imunohistochemickém průkazu GFAP), spíše hypocelulární nebo středně buněčná. Nádorové buňky byly bez atypií a bez výraznější mitotické aktivity (obr. 8). Ojediněle byly detekovány hyalinní globule či náznaky Rosenthalových fibril. Tyto úseky přecházely do minoritnějších buněčnějších oblastí tvořených pravidelnými okrouhlými buňkami s kulatými jádry se zrnitým chromatinem, které byly negativní při imunohistochemickém průkazu GFAP, ale vykazovaly slabou pozitivitu při průkazu synaptofyzinu a NSE. Negativní byl průkaz širokospektrých cytokeratinů a EMA. Marker CD56 byl pozitivní v obou komponentách, IDH1 i p53 byly v obou komponentách negativní. Průkaz INI1 byl v obou komponentách zachovalý. Proliferační aktivita stanovená pomocí markeru Ki-67 byla nízká (do 2 % buněk).
Obr. 8. HE 8-leté dívky s low-grade gliomem. 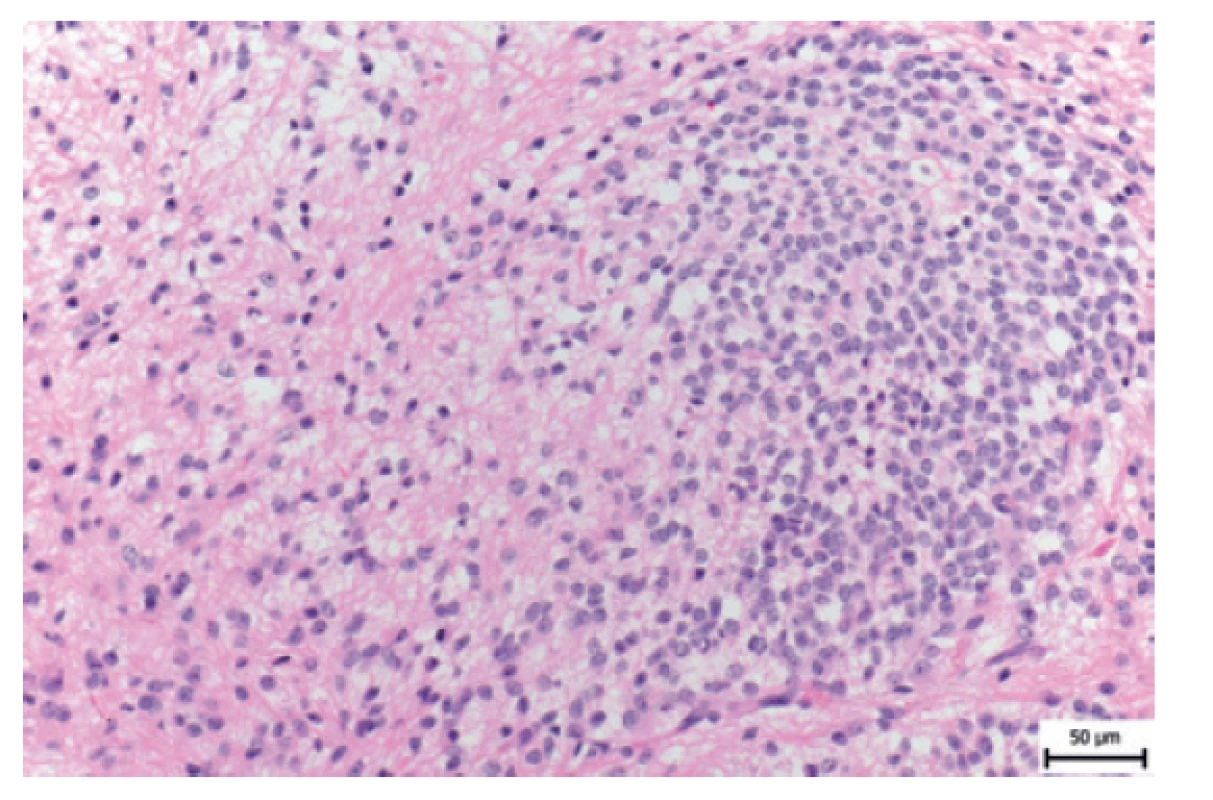
Ve vyšetřovaném materiálu dominoval obraz low-grade gliomu. Ložiskově byl zastižen přechod do oblasti s neurocytární diferenciací. Proto byl nádor interpretován jako nádor ze skupiny glioneuronálních nádorů, grade I/II.
Vzorek nádoru byl dále vyšetřen pomocí SNP array, kde bylo vzneseno podezření na možnou přítomnost fúzního genu s genem NTRK3. Z důvodu suspektní přestavby genu NTRK3 byl vzorek vyšetřen pomocí RNA sekvenování nové generace za použití Archer FusionPlex Sarcoma kitu s nálezem dosud nepopsaného fúzního genu BCR1/NTRK3 (obr. 9a). Na základě výsledku byly designovány primery pro PCR a pomocí metody RT-PCR a následné sekvenace byla prokázána fúze exonu 1 genu BCR1 s exonem 14 genu NTRK3 (obr. 9b). Na základě průkazu terapeuticky postihnutelného genu je pacientka dva roky úspěšně léčena pomocí cílené terapie pomocí Larotrectinibu, po kterém došlo ke stabilizaci onemocnění a výraznému zlepšení zraku.
Obr. 9. A: Nově diagnostikovaný fúzní gen BCR-NTRK3 u dětské pacientky s low-grade gliomem. Analýza pomocí Arriba software https://github. com/suhrig/arriba. B: Sekvenační analýza fúzního produktu BCR-NTRK3 pomocí Sangerovy sekvenace. 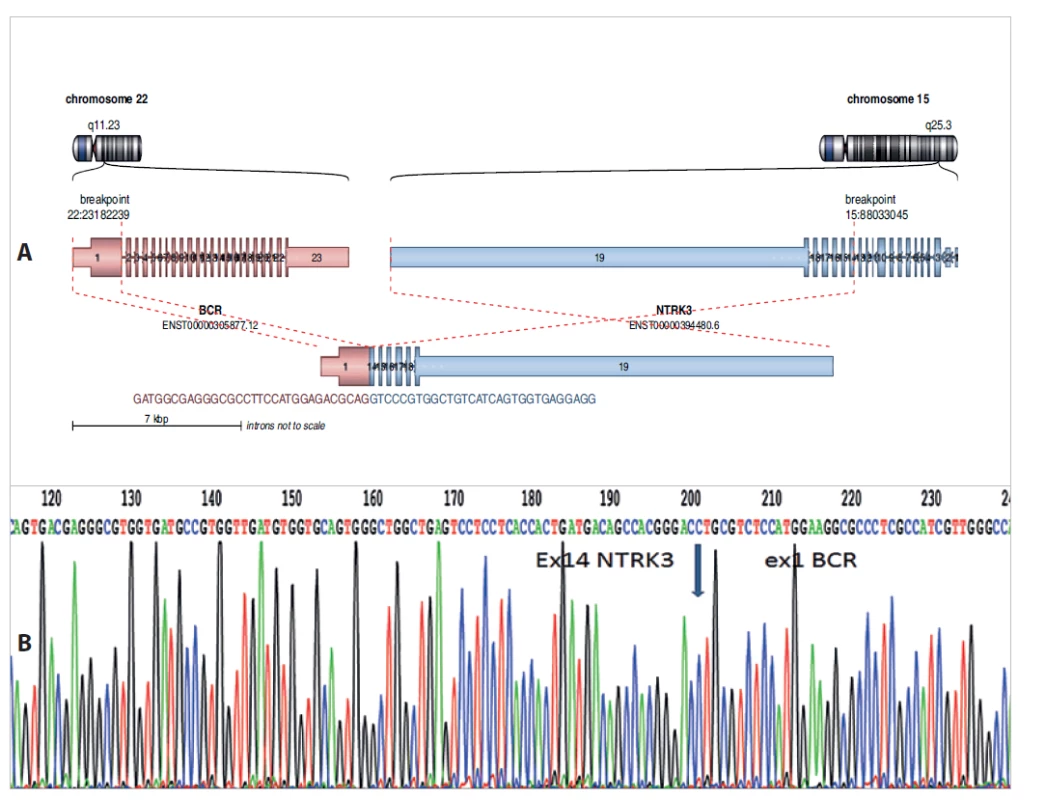
Obr. 10. MRI pacientky s LGG s prokázaným fúzním genem BCR1-NTRK3. Na MRI mozku se zaměřením na zrakovou dráhu byl nález solidní patologické expanze v oblasti optického chiasmatu s propagací suprachiasmaticky, ventrálně do oblasti baze frontálního laloku vlevo a dorzálně do oblasti mediální části temporálního laloku a insuly vpravo. 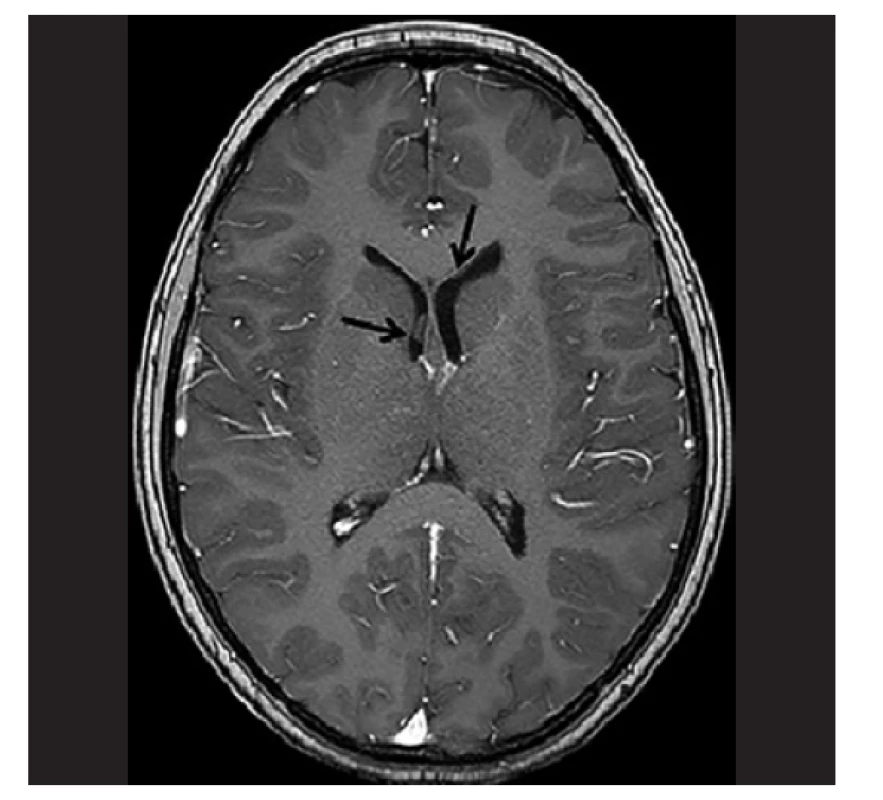
PODĚKOVÁNÍ
Diagnostika a výzkum byly podpořeny z Výzkumného záměru Ministerstva zdravotnictví ČR 00064203, grantem Primus/19/ MED/06 a Nadací 1000 statečných.
PROHLÁŠENÍ
Autor práce prohlašuje, že v souvislosti s tématem, vznikem a publikací tohoto článku není ve střetu zájmů a vznik ani publikace článku nebyly podpořeny žádnou farmaceutickou firmou. Toto prohlášení se týká i všech spoluautorů.
Adresa pro korespondenci:
RNDr. Lenka Krsková, Ph.D.
Ústav patologie a molekulární medicíny 2. LF UK a FN Motol
V Úvalu 84, Praha 5, 15006
tel: 224435634
Zdroje
1. Sánchez R, Ayala R, Martínez-López J. Minimal residual disease monitoring with nextgeneration sequencing methodologies in hematological malignancies. Int J Mol Sci 2019; 20(11): 2832.
2. Helman E, Nguyen M, Karlovich CA, et al. Cell-Free DNA Next-Generation Sequencing Prediction of Response and Resistance to Third-Generation EGFR Inhibitor. Clin Lung Cancer 2018; 19(6): 518-530.
3. Bettegowda C, Sausen M, Leary RJ, et al. Detection of circulating tumor DNA in earlyand late-stage human malignancies. Sci Transl Med 2014; 6(224): 224ra24.
4. Chen M, Zhao H. Next-generation sequencing in liquid biopsy: cancer screening and early detection. Hum Genomics 2019; 13(1): 34.
5. Froňková E. Next-generation sequencing. Cesk Patol 2013; 49(3):129-32.
6. Alvarez-Breckenridge C, Miller JJ, Nayyar N, et al. Clinical and radiographic response following targeting of BCAN-NTRK1 fusion in glioneuronal tumor. NPJ Precis Oncol 2017; 1(1): 5.
7. Schreck KC, Grossman SA, Pratilas CA. BRAF mutations and the utility of RAF and MEK inhibitors in primary brain tumors. Cancers (Basel) 2019; 11(9): 1262.
8. Gullapalli RR, Lyons-Weiler M, Petrosko P, Dhir R, Becich MJ, LaFramboise WA. Clinical Integration of Next-Generation Sequencing Technology. Clin Lab Med 2012; 32(4): 585 - 599.
9. Heyer EE, Deveson IW, Wooi D, et al. Diagnosis of fusion genes using targeted RNA sequencing. Nat Commun 2019; 10(1): 1388.
10. Lam SW, Cleton-Jansen AM, Cleven AHG, et al. Molecular Analysis of Gene Fusions in Bone and Soft Tissue Tumors by Anchored Multiplex PCR–Based Targeted Next-Generation Sequencing. J Mol Diagn 2018; 20(5): 653 - 663.
Štítky
Patologie Soudní lékařství Toxikologie
Článek Next-Generation PathologyČlánek 'PATOLOGIE MĚKKÝCH TKÁNÍČlánek 'PATOLOGIE ORL OBLASTIČlánek 'UROPATOLOGIEČlánek 'GYNEKOPATOLOGIEČlánek 'NEFROPATOLOGIEČlánek 'HEPATOPATOLOGIEČlánek 'KARDIOPATOLOGIEČlánek 'HEMATOPATOLOGIEČlánek 'PATOLOGIE MĚKKÝCH TKÁNÍČlánek 'PATOLOGIE GITČlánek 'CYTODIAGNOSTIKAČlánek 'LABORATORNÍ METODYČlánek 'PULMOPATOLOGIEČlánek Sekvenování nové generace a molekulární onkologický multidisciplinární tým z pohledu onkologa
Článek vyšel v časopiseČesko-slovenská patologie

2021 Číslo 3-
Všechny články tohoto čísla
- Next-Generation Pathology
- Cesta k cíli vede přes spolupráci
- 'PATOLOGIE MĚKKÝCH TKÁNÍ
- 'PATOLOGIE ORL OBLASTI
- 'UROPATOLOGIE
- 'GYNEKOPATOLOGIE
- 'NEFROPATOLOGIE
- 'HEPATOPATOLOGIE
- 'KARDIOPATOLOGIE
- 'HEMATOPATOLOGIE
- 'PATOLOGIE MĚKKÝCH TKÁNÍ
- 'PATOLOGIE GIT
- 'CYTODIAGNOSTIKA
- 'LABORATORNÍ METODY
- Sekvenování nové generace – nástroj vědy či rutinní patologie?
- 'PULMOPATOLOGIE
- Sekvenování nové generace a molekulární onkologický multidisciplinární tým z pohledu onkologa
- Možnosti molekulárního testování somatických aberací v nádorové tkáni metodou NGS v rutinní praxi - aktuální situace v České republice
- Sekvenování nové generace a jeho aplikace v diagnostice neuromuskulárních onemocnění
- Využití metylačního profilu v diagnostice a prognostice nádorových onemocnění CNS
- Přehled problematiky hodnocení tumor infiltrujících lymfocytů u karcinomu prsu v rutinní diagnostické praxi
- Nediagnostikovaná kryptokoková meningoencefalitída s rozsiahlymi hypoxickoischemickými zmenami mozgu charakteru tretieho stupňa difúzneho axonálneho poškodenia
- Obrovskobuněčná myokarditida u mladé ženy odhalená při pitvě: kazuistika
- Doporučení pro testování PD-L1: metodika testování a reportování výsledků Verze_1 (17. 5. 2021)
- Molekulární testování u karcinomu endometria (společné doporučení ČOS, onkogynekologické sekce ČGPS, SROBF a SČP ČLS JEP)
- Česko-slovenská patologie
- Archiv čísel
- Aktuální číslo
- Informace o časopisu
Nejčtenější v tomto čísle- Doporučení pro testování PD-L1: metodika testování a reportování výsledků Verze_1 (17. 5. 2021)
- Sekvenování nové generace – nástroj vědy či rutinní patologie?
- Molekulární testování u karcinomu endometria (společné doporučení ČOS, onkogynekologické sekce ČGPS, SROBF a SČP ČLS JEP)
- Využití metylačního profilu v diagnostice a prognostice nádorových onemocnění CNS
Kurzy
Zvyšte si kvalifikaci online z pohodlí domova
Revma Focus: Spondyloartritidy
nový kurz
Autoři: prof. MUDr. Vladimír Palička, CSc., Dr.h.c., doc. MUDr. Václav Vyskočil, Ph.D., MUDr. Petr Kasalický, CSc., MUDr. Jan Rosa, Ing. Pavel Havlík, Ing. Jan Adam, Hana Hejnová, DiS., Jana Křenková
Autoři: MUDr. Irena Krčmová, CSc.
Všechny kurzyPřihlášení#ADS_BOTTOM_SCRIPTS#Zapomenuté hesloZadejte e-mailovou adresu, se kterou jste vytvářel(a) účet, budou Vám na ni zaslány informace k nastavení nového hesla.
- Vzdělávání



