-
Články
Top novinky
Reklama- Vzdělávání
- Časopisy
Top články
Nové číslo
- Témata
Top novinky
Reklama- Kongresy
- Videa
- Podcasty
Nové podcasty
Reklama- Kariéra
Doporučené pozice
Reklama- Praxe
Top novinky
ReklamaMultiple-locus variable number tandem repeat analýza souboru kmenů Bordetella pertussis v ČR z období 1967–2015: rozšíření varianty adaptované na proočkovanou populaci
Multiple-locus variable number tandem repeat analysis of Bordetella pertussis strains collected in the Czech Republic in 1967–2015: spread of a variant adapted to the population with a high vaccination coverage
Aim:
To perform multiple-locus variable number tandem repeat analysis (MLVA) of B. pertussis strains from the collection of the National Reference Laboratory for Diphtheria and Pertussis (NRL/DIPE), National Institute of Public Health (NIPH), Prague. The study strains were isolated from clinical specimens collected mostly in the Czech Republic over a nearly 50-year period from 1967 to 2015 (June). The isolates from three periods characterized by different vaccination strategies and trends in pertussis are compared for genetic diversity and distribution of MLVA types (MT). Based on the results obtained, the suitability for use of MLVA in the analysis of epidemic outbreaks of B. pertussis in the Czech Republic is considered.Material and methods:
DNA samples extracted from B. pertussis strains included in the present study were examined by MLVA using the standard protocol. Data were processed by means of the eBURST algorithm and the calculation of the Simpson diversity index (DI) was used for the statistical analysis. Data were analyzed as a whole and also separately for strains from the three periods: 1967–1980, 1990–2007, and 2008–2015 (June).Results:
Fourteen different MT were detected in the study strains, with three of them not being reported before. The most common MTs were MT27 and MT29. MT29 was predominant in 1967–1980 while MT27 was the most prevalent in 1990–2007 and 2008–2015 (June). The DI was the lowest (0.49) in 2008–2015 (June), and comparably higher DIs were calculated for the two previous periods (i.e. 0.667 for 1967–1980 and 0.654 for 1990–2007).Conclusion:
MLVA revealed a decrease in genetic diversity and shifts in MT distribution of B. pertussis strains isolated from clinical specimens in the Czech Republic from 1967 to 2015 (June). These shifts in the Czech Republic can be characterized as a progressive increase in global MTs at the expense of the locally unique ones. The most common MT, similarly to other geographical areas with long-term high vaccination coverage, is MT27. The results of MLVA of 136 B. pertussis strains can provide a background for using this method in molecular epidemiological analysis of smaller groups of strains.KEYWORDS:
Bordetella pertusis – MLVA – population – diversity – adaptation
Autoři: D. Lžičařová 1,2; J. Zavadilová 1; M. Musílek 1
; Z. Jandová 1; P. Křížová 1
; K. Fabiánová 1,3
Působiště autorů: Centrum epidemiologie a mikrobiologie, Státní zdravotní ústav, Praha 1; Ústav lékařské mikrobiologie 2. LF UK a FN Motol 2; Ústav epidemiologie, 3. LF UK, Praha 3
Vyšlo v časopise: Epidemiol. Mikrobiol. Imunol. 65, 2016, č. 2, s. 102-111
Kategorie: Původní práce
Souhrn
Cíl práce:
Cílem práce je Multiple-locus variable number tandem repeat analýza (MLVA) souboru kmenů B. pertussis ze sbírky Národní referenční laboratoře pro pertusi a difterii (NRL/DIPE) Státního zdravotního ústavu (SZÚ) Praha. Jedná se o kmeny získané ze vzorků klinického materiálu převážně na území ČR v téměř padesátiletém období 1967–2015 (červen). Je porovnávána genetická diverzita a zastoupení detekovaných MLVA typů (MT) ve třech obdobích lišících se vakcinační strategií a trendem výskytu pertuse. Na podkladě získaných výsledků je posouzena vhodnost použití metody MLVA pro analýzu epidemických výskytů B. pertussis v ČR.Materiál a metodiky:
Vzorky DNA extrahované z kmenů B. pertussis studovaného souboru byly analyzovány metodou MLVA podle standardizovaného postupu. Data byla zpracována pomocí algoritmu eBURST, pro statistické zpracování byl zvolen výpočet Simpsonova indexu diverzity (DI). Data byla zpracována pro celý soubor a též odděleně pro skupiny kmenů z tří období: 1967–1980, 1990–2007 a 2008–2015 (červen).Výsledky:
V souboru bylo detekováno 14 různých MT, z toho 3 dosud nezjištěné. Nejčetnější byly v celém souboru MT27 a MT29. MT29 převládal v období 1967–1980, MT27 v obdobích 1990–2007 a 2008–2015 (červen). Hodnota DI byla nejnižší (0,49) pro období 2008–2015 (červen), pro dvě předchozí období byly hodnoty vyšší a vzájemně srovnatelné (0,667 pro období 1967–1980 a 0,654 pro období 1990–2007).Závěr:
MLVA analýzou souboru kmenů B. pertussis izolovaných z klinického materiálu na území ČR v období 1967–2015 (červen) byl prokázán pokles genetické diverzity ve studované populaci a změny v zastoupení a četnosti jednotlivých MT. Tyto změny lze charakterizovat jako postupný nárůst výskytu celosvětově rozšířených MT v České republice na úkor MT regionálně unikátních. Převažujícím MT, stejně jako ve většině geografických oblastí s dlouhodobě proočkovanou populací, je MT27. Výsledky MLVA analýzy souboru 136 kmenů B. pertussis mohou být podkladem pro účelné využití metody k molekulárně epidemiologickému zpracování souborů menšího rozsahu.KLÍČOVÁ SLOVA:
Bordetella pertusis – MLVA – populace – diverzita – adaptaceÚVOD
Od 90. let 20. století je v ČR zaznamenáván vzestup nemocnosti pertusí [1]. Výskyt onemocnění narůstá i přes trvale vysoké procento proočkovanosti populace [2]. V roce 1958 bylo na území ČR zahájeno plošné očkování celobuněčnou pertusovou vakcínou. Následoval strmý pokles nemocnosti. Nízký výskyt pertuse trval až do počátku 90. let, od roku 1993 byl pozorován vzestup nemocnosti [1]. V roce 2002 bylo zahájeno očkování acelulární vakcínou, do roku 2006 se očkovalo souběžně celobuněčnou (většinová populace) a acelulární vakcínou (z indikace pediatra nebo za úhradu). Od roku 2007 se pro plošné očkování používá výhradně acelulární vakcína. Vzestupný trend výskytu onemocnění pertusí trvá od roku 1993 do současné doby [1, 2, 3]. Do 90. let byla většina případů zaznamenána u dětí mladších tří let. Od roku 1993 je nejvyšší nárůst pozorován ve skupině 10–14 let [2]. Reakcí na tento trend bylo zahájení přeočkovávání všech 10–11letých v roce 2009. Následoval posun maximálního výskytu pertuse do věkové kategorie 15–19letých [3]. V roce 2014 byla pozorována dosud nejvyšší nemocnost od roku 1963 [3]. Podobný vývoj epidemiologické situace v dlouhodobě vysoce proočkovaných populacích je pozorován celosvětově [4, 5, 6, 7, 8].
Periodické kolísání výskytu pertuse ve 2–4letých intervalech, pozorované i po poklesu nemocnosti [1], značí, že B. pertussis koluje v populaci i přes dlouhodobě vysokou proočkovanost [9, 10].
V literatuře se uvádí více možných příčin vzestupu nemocnosti:
- vyvanutí imunity [11, 12, 13],
- rozdílnost v typu imunitní odpovědi na acelulární vakcínu a na přirozenou infekci [14, 15],
- nižší účinnost vakcín oproti předpokladu [16],
- adaptace B. pertussis na imunizovanou populaci [7, 9, 13, 17, 18, 19],
- uplatnění asymptomatických nosičů v přenosu onemocnění [9] i další změny v procesu transmise [20, 21],
- lepší povědomí o výskytu onemocnění a nové možnosti diagnostiky [22].
V postvakcinační éře se mění nejen epidemiologická situace, ale i klinický průběh pertuse. Je možné se setkat jak s těžkým až fatálním průběhem onemocnění u neočkovaných dětí [23], s klasickým průběhem ve 3 stadiích u dospívajících a dospělých [24], tak i s atypickým průběhem pod obrazem dlouhotrvajícího kašle nereagujícího na obvyklou antitusickou terapii [24, 25]. Pravděpodobný je i výskyt subklinických forem a asymptomatického nosičství [9].
Analýzou genomu B. pertussis a nejblíže příbuzných druhů Bordetella bronchiseptica a Bordetella parapertussis bylo zjištěno, že populace B. pertussis je geneticky vysoce monomorfní a evoluční vývoj této populace je klonální. Tento typ bakteriální populace je charakterizován praktickou absencí horizontálního přenosu genů. V průběhu adaptace na lidského hostitele byl genom B. pertussis výrazně redukován, další geny byly inaktivovány. Proces redukce genomu B. pertussis pokračuje. Ztráta či neúčinná exprese genů kódujících povrchové struktury může hrát roli v adaptaci B. pertussis ve smyslu úniku před obranyschopností očkovaného hostitele. V populacích s různými strategiemi vakcinace dochází ke ztrátě shodných oblastí genomu B. pertussis. Plasticita genomu B. pertussis je podmíněna několika mechanismy. Jsou to především změny způsobené přítomností a aktivitou inzerčních sekvencí (IS) a mutace. Získání genů jiných bakteriálních druhů nebylo u B. pertussis prokázáno. Genom B. pertussis obsahuje ve srovnání s genomem B. bronchiseptica i B. parapertussis výrazně vyšší počet IS, převládá IS 481. IS se podílejí na ztrátě nebo inaktivaci genů mechanismem delece, homologní rekombinace, inverze, transpozice, vmezeřením do strukturálních nebo řídících oblastí genomu. Tímto mechanismem mohou být inaktivovány i geny kódující významné faktory patogenity, pertaktin a pertusový toxin. Mutace se na vývoji variability genomu B. pertussis podílejí ve velké míře, především substituce (Single nucleotide polymorphisms, SNP), méně často i inzerce a delece. Z hlediska využití pro účely typizace jsou podstatné zejména mutace v genech kódujících povrchové antigenní struktury a faktory patogenity a mutace v tandemových repeticích. V oblastech repetic se uplatňují, byť v menší míře, také rekombinace. Nápadně vyšší procento mutací v genech kódujících povrchové struktury a faktory patogenity svědčí o selekčním tlaku mechanismů specifické imunity v proočkované populaci [26, 27, 28, 29, 30, 31, 32, 33, 34, 35].
Detekce alelické proměnlivosti v oblastech kódujících vybrané antigeny je podstatou analýzy Multi Antigen Sequence Typing (MAST) [36, 37, 38]. Výsledky sekvenační analýzy MAST ukazují, že v zemích s dlouhodobě vysokou proočkovaností populace narůstá výskyt kmenů B. pertussis nesoucích antigenní determinanty odlišné od antigenních determinant původních kmenů použitých pro výrobu vakcín [18, 19, 33, 35, 38, 39]. Změny některých antigenních determinant jsou spojeny s vyšší virulencí kmenů [18, 19, 33, 40, 41, 42, 43]. V ČR byla u 70 izolátů B. pertussis z let1967–2010 ze sbírky NRL/DIPE provedena sekvenační analýza oblastí kódujících 3 zásadní antigenní determinanty, ptxA, gen pro podjednotku S1 pertusového toxinu, prnA, oblast 1 genu pro pertaktin, fim3, gen pro fimbrie typu 3 a ptxP, promotor řídící expresi genu pro pertusový toxin. Výsledky ukazují na změny ve všech analyzovaných oblastech genomu [38].
Variabilita v oblasti tandemových repetic vzniká nejčastěji na podkladě tzv. slipped strand mispairing, kdy v průběhu replikace DNA dochází k inzerci nebo deleci jedné nebo více jednotek v replice. Výsledkem je odlišný počet repetic v templátu a v replice. Tyto mutace lze považovat za náhodné, ve fenotypu se mohou a nemusí projevit, závisí to mj. na počtu bází v repetitivní jednotce [44]. Přesná funkce genů obsahujících tyto repetice často není známa, stejně jako jejich přesný vztah k evolučnímu vývoji a fylogenetickým vazbám v rámci druhu [44]. Méně častým mechanismem změn v oblasti repetic je homologní rekombinace. U B. pertussis byla popsána jako jeden z mechanismů vedoucích k polymorfismu v oblasti genu tcfA. Výsledkem homologní rekombinace mezi dvěma repeticemi je delece celého genu [45].
V genomu B. pertussis (typový kmen Tohama I) bylo nalezeno 13 míst tandemových repetic s různou mírou variability. Z tohoto počtu bylo 5 repetitivních sekvencí dostatečně polymorfních. Zároveň se tyto sekvence vyskytovaly v genomu většiny kmenů v populaci B. pertussis, byly tedy využitelné pro MLVA typizaci [46]. Dvě VNTR oblasti zvolené pro MLVA, VNTR1 a VNTR4 jsou umístěny v genech kódujících membránové struktury, zatímco oblasti VNTR3, VNTR5 a VNTR6 jsou umístěny v pseudogenech. Jedna z oblastí (VNTR3) byla u části studovaných izolátů duplikována [46].
Metoda MLVA je určena pro molekulární analýzu bakteriálních populací jak v rámci místně a časově ohraničených epidemických výskytů [47, 48, 49, 50, 51], tak i pro analýzu souborů bakteriálních kmenů pocházejících z větších územních celků a z různých období [52, 53, 54, 55, 56]. Její velkou předností je možnost snadného porovnávání výsledků získaných na různých pracovištích [52, 53].
Databáze dosud popsaných MT B. pertussis je dostupná na webových stránkách Multiple-Locus Variable number tandem repeat Analysis [57], stejně jako standardizovaný MLVA protokol. Na základě získaných MLVA profilů a typů lze porovnávat i izoláty analyzované na různých pracovištích, případně určit jejich vzájemnou příbuznost, to vše za předpokladu, že použitá metoda je dostatečně validovaná a standardizovaná [58]. Na podkladě získaných dat je možné stanovit a porovnávat genetickou diverzitu bakteriálních populací, detekovat klonální expanzi a jiné změny ve studované populaci [46]. V ČR dosud nebyla k dispozici metoda vhodná k molekulární typizaci klinických izolátů B. pertussis. V NRL/DIPE se prováděla sérotypizace B. pertussis pomocí monoklonálních protilátek proti fimbriím typu 2 a 3, vybrané kmeny byly analyzovány několika metodami v rámci evropské surveillance B. pertussis [53].
Cílem této práce byla MLVA analýza souboru 136 kmenů B. pertussis ze sbírky NRL/DIPE SZÚ Praha. Kmeny byly izolovány z klinického materiálu v období 1967–2015. Celý soubor, kromě pěti kmenů izolovaných na území dnešní Slovenské republiky (SR), pochází z území ČR. Byla porovnána genetická diverzita a zastoupení detekovaných MT ve třech obdobích lišících se vakcinační strategií a trendem výskytu pertuse v populaci žijící na území ČR. Výsledky analýzy velkého souboru kmenů by zároveň měly být podkladem pro posouzení vhodnosti použití metody MLVA pro analýzu možného epidemického výskytu B. pertussis v ČR.
MATERIÁL A METODIKY
Soubor kmenů B. pertussis
Analyzováno bylo celkem 136 kmenů B. pertussis ze sbírky NRL/DIPE. Kmeny izolované z klinického materiálu na území ČR (131 kmen) a SR (5 kmenů) byly zaslány do NRL/DIPE v průběhu téměř padesátiletého období – roky 1967–2015 (červen).
Období 1967–2015 (červen), ze kterého pocházejí všechny studované kmeny B. pertussis, bylo pro účely studie rozděleno na tři kratší období s ohledem na trend výskytu onemocnění pertusí na území ČR a na používanou očkovací látku:
- 1967–1980, období očkování celobuněčnou vakcínou československé výroby a klesajícího trendu výskytu pertuse;
- 1990–2007, období souběžného používání zahraničních vakcín, celobuněčné i acelulární vakcíny a počátku vzestupného trendu výskytu pertuse;
- 2008–2015 (červen), období očkování zahraniční acelulární vakcínou a pokračujícího vzestupného trendu výskytu pertuse.
Kultivace a identifikace kmenů
Kmeny B. pertussis byly kultivovány na Charcoal agaru (Oxoid) a na Bordet-Gengou agaru v normální atmosféře při teplotě 35 ± 1 °C po dobu 96 hodin. Druhová identifikace byla ověřena sklíčkovou aglutinací (Bordetella pertussis diagnostické antisérum, Remel LTD, USA).
Extrakce DNA
Extrakce DNA byla provedena pomocí soupravy QIAamp DNA Mini Kit (Qiagen, SRN) podle protokolu pro extrakci DNA gramnegativních bakterií doporučeného výrobcem.
VNTR PCR
PCR amplifikace úseků DNA obsahujících analyzované tandemové repetice byla provedena podle publikovaného protokolu [46, 57] v termocykleru Veriti (Applied Biosystems), USA. Oproti původnímu protokolu byla u VNTR6 snížena teplota hybridizace primerů na 62 °C. Po změně reakčních podmínek bylo dosaženo optimální kvality produktů amplifikace, nutné pro jejich další zpracování.
Analýza produktů
Příprava PCR produktů k analýze v genetickém analyzátoru 3130xl (Applied Biosystems, USA), byla provedena podle protokolu dostupného na webových stránkách MLVA [57]. Při testování protokolu v konkrétních podmínkách laboratoře bylo zjištěno, že pro dosažení uspokojivých výsledků je třeba změnit ředění standardu a některých produktů amplifikace. LIZ500 Marker byl použit v ředění 1 : 300, výsledné ředění produktů PCR bylo pro VNTR1 a VNTR6 1 : 200, pro VNTR3, VNTR4 a VNTR5 1 : 500.
Vzorky byly vždy analyzovány bezprostředně po přípravě, bez skladování.
Určení alel na základě délky amplikonů
Zjištění počtu VNTR repetic u jednotlivých kmenů bylo provedeno ve výstupních souborech analyzátoru softwarem Gene Marker (SoftGenetics, USA). Délky úseků jednotlivých repetic a jejich počet v izolátech byl pro každý nově zjištěný MT ověřen sekvenací analyzátorem 3130xl [46].
Hodnoty délek VNTR repetic byly porovnány s hodnotami uvedenými pro jednotlivé VNTR v seznamu na [57].
Přiřazení MLVA typů
Pro každý testovaný kmen bylo provedeno 5 VNTR reakcí a po následné analýze produktů a určení alel byl stanoven MLVA profil. MLVA profil je tvořen kombinací 6 přirozených čísel označujících počet repetic u jednotlivých VNTR alel. Každému MLVA profilu bylo přiřazeno přirozené číslo, označující příslušný MT. Známé MT byly přiřazeny pomocí porovnání profilů získaných testováním s databází známých MT [57]. Kmeny, jejichž MLVA profily neodpovídaly žádnému z dosud známých MT, byly pro ověření počtu repetic testovány sekvenací [46] a odeslány do Rijksinstituut voor Volksgezondheid en Milieu (RIVM, nizozemská instituce odpovídající funkcí a zaměřením přibližně našemu SZÚ) k ověření, přiřazení MT a zařazení do databáze MT.
Zpracování dat
Výstupy z provedené MLVA analýzy byly zpracovány pomocí algoritmu eBURST (based upon related sequence types) [http://eburst.mlst.net]. Výsledkem je uspořádání souboru získaných MT do seskupení podle charakteristik stanovených metodou MLVA. Skupina kmenů (group) byla pro účely této práce nadefinována pro minimální počet pěti shodných z celkem šesti alel. Seskupení vytvořené algoritmem eBURST odpovídá v tomto případě klonálnímu (MLVA) komplexu, definovanému v práci Spratta et al. jako skupina navzájem blízce příbuzných izolátů v rámci studované bakteriální populace, u nichž lze předpokládat, že pocházejí ze společného výchozího kmene [59].
Statistické zpracování
Pro každé ze stanovených období byl vypočten Simpsonův index diverzity (DI) v modifikaci podle Huntera a Gastona [60], 95% interval spolehlivosti (CI) byl vypočten podle Grundmanna [61]. Byl použit Online tool for quantitative assessment of classification agreement [62].
VÝSLEDKY
136 studovaných kmenů B. pertussis z celého sledovaného období 1967–2015 (červen) bylo přiřazeno ke 14 různým MT, z toho u 10 kmenů byl stanoven a přiřazen některý ze 3 nových, dosud nepopsaných MT (MT308, MT309 a MT310). Tyto nové typy byly po ověření zařazeny do databáze MT. MT141, MT255, MT309, MT310, MT75 a MT44 byly nalezeny každý pouze u jednoho kmene. Dva nejčastější MT byly MT27 a MT29. Nebyl zaznamenán žádný MT, který by se vyskytoval ve všech třech sledovaných obdobích. DI pro celý soubor měl hodnotu 0,697 a CI (95%) byl 0,623-0,771 – tabulka 1.
Tab. 1. Přehled výsledků MLVA analýzy souboru kmenů B. pertussis, ČR, 1967–2015 (červen) Table 1. MLVA analysis of <i>B. pertussis</i> strain collection, Czech Republic, 1967–2015 (June) 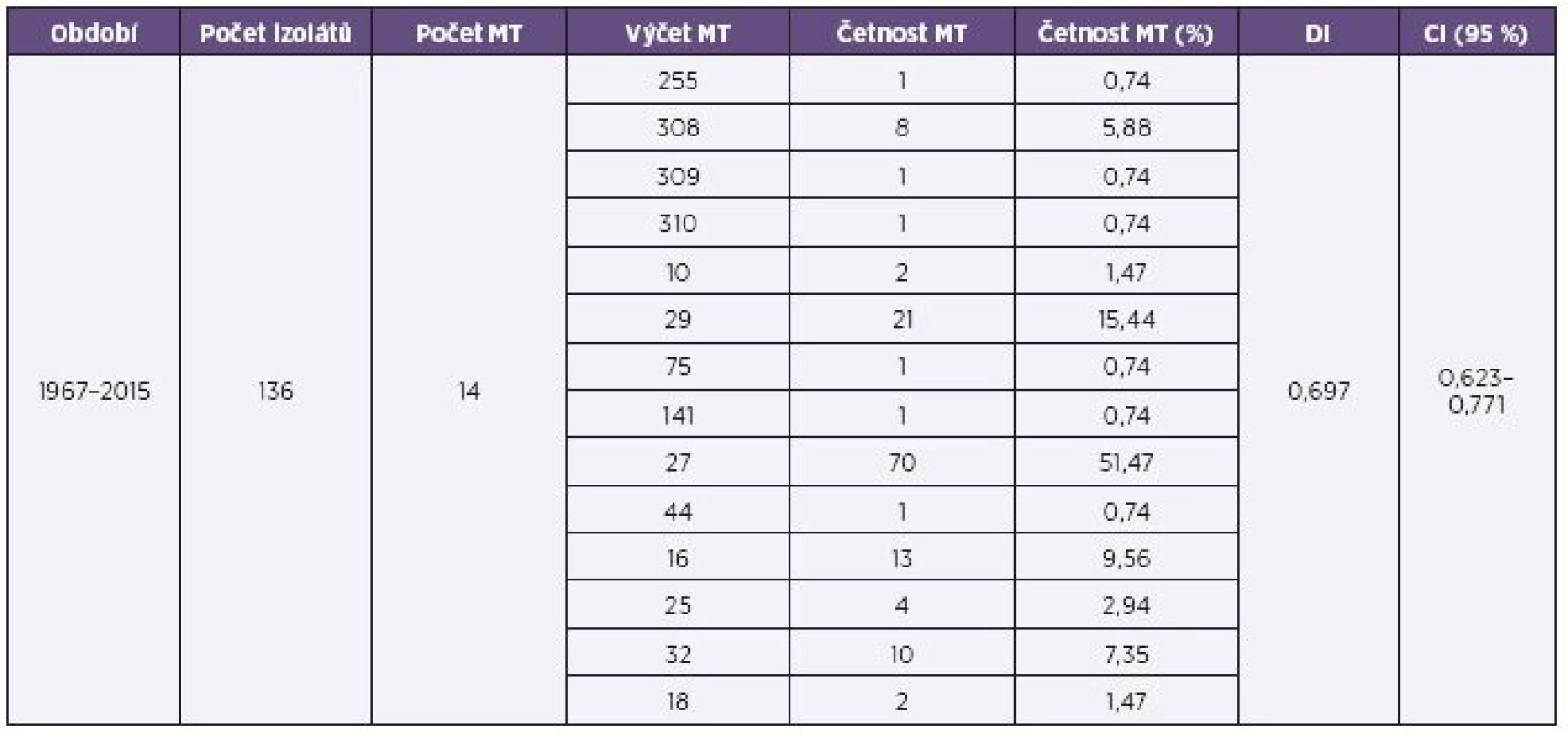
Přehled MT zastoupených ve třech srovnávaných obdobích shrnuje tabulka 2.
Tab. 2. Přehled výsledků MLVA analýzy souboru kmenů <i>B. pertussis,</i> ČR, 1967–1980, 1990–2007, 2008–2015 (červen) Table 2. MLVA analysis of <i>B. pertusis</i> strain collection, Czech Republic, 1967–1980, 1990–2007, 2008–2015 (June) 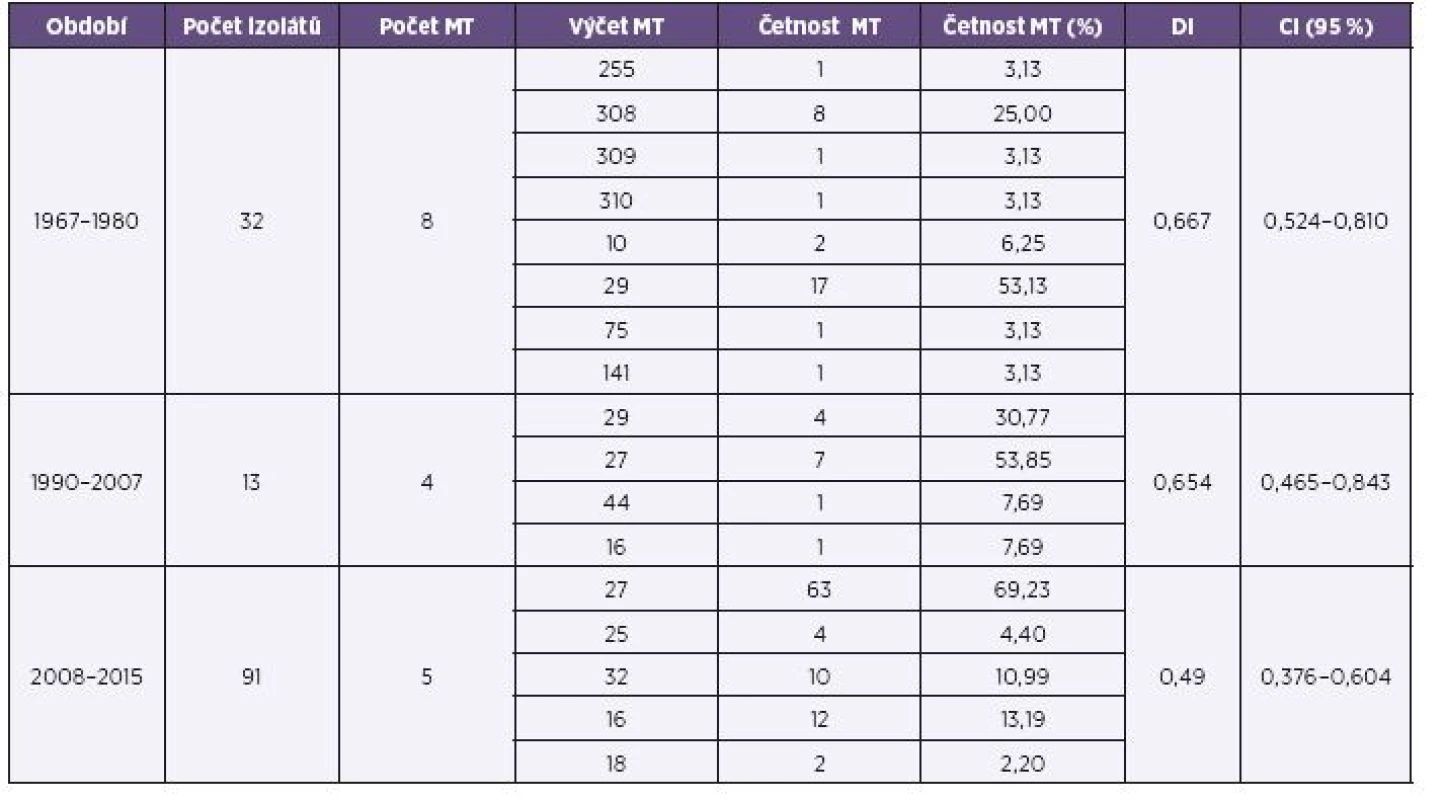
Z období plošného očkování celobuněčnou vakcínou a dlouhodobého trendu poklesu nemocnosti pertusí (1967–1980) bylo analyzováno 32 kmenů B. pertussis. Tyto kmeny byly přiřazeny k 8 MT (MT141, MT255, MT308, MT309, MT310, MT10, MT75, MT29). Kromě MT29 se ostatní zjištěné MT vyskytovaly pouze v souboru z tohoto období. 10 kmenů bylo přiřazeno k dosud nepopsaným MT. MT308 byl v souboru zastoupen 8 kmeny (25 %), ostatní nově přiřazené MT (MT309, MT310) byly zastoupeny po jednom kmeni. MT29 v tomto období jednoznačně převažoval, byl zastoupen 17 kmeny (53 %) a jako jediný se vyskytoval i v následujícím období. DI pro období 1967–1980 měl hodnotu 0,667, CI (95%) byl 0,524–0,810.
Z období souběžného očkování celobuněčnou a acelulární vakcínou a počátku vzestupu nemocnosti (1990–2007) bylo do studie zařazeno 13 kmenů B. pertussis patřících k 4 různým MT. MT44 byl zastoupen jediným kmenem a vyskytl se výhradně v tomto období. MT29 (převažující v období předcházejícím) byl zastoupen 5 kmeny (35 %) a po roce 1993 již nebyl jeho výskyt zaznamenán. MT16 byl v souboru z tohoto období zastoupen jedním kmenem z roku 2007 a jeho výskyt byl vyšší v následujícím období. Převládajícím typem byl MT27, zastoupený 7 kmeny (50 %), jeho výskyt byl rovněž vyšší v následujícím období. DI pro období 1990–2007 měl hodnotu 0,654, CI (95%) 0,465–0,843.
Z období plošného očkování acelulární vakcínou a pokračujícího trendu vzestupu nemocnosti pertusí (2008–2015) bylo analyzováno 91 kmenů B. pertussis. Kmeny byly přiřazeny k 5 různým MT, z toho MT27 a MT16 se již vyskytovaly v období předcházejícím, MT18, MT25 a MT32 se v předchozích dvou obdobích nevyskytly. Převažujícím typem byl stejně jako v předchozím období MT27, jeho poměrné zastoupení bylo v tomto období ještě vyšší (69 %). Také poměrný výskyt MT16 byl oproti předchozímu období vyšší (13 %). MT32 byl zastoupen 10 kmeny (11 %), MT25 4 kmeny (4,4 %) a MT18 2 kmeny (2,2 %). Výskyt MT25 byl poprvé zaznamenán u kmene z roku 2014. DI měl pro období 2008–2015 hodnotu 0,490, CI (95%) 0,376–0,604.
Analýza dat algoritmem eBURST [59] podle nadefinovaných parametrů ukázala, že MT z celého analyzovaného souboru kmenů tvoří jednu skupinu odpovídající MLVA komplexu a jeden singleton – MT75. V diagramu eBURST pro celý soubor testovaných kmenů je za ancestrální typ označen MT27 – obrázek 1. Tento MT byl současně zastoupen nejvyšším počtem kmenů. MT29 (druhý nejčastější MT) a MT141 byly pak označeny jako „subgroup founders“, ancestrální MT v rámci podskupiny. MT75 se od všech ostatních MT lišil ve více než dvou alelách, a proto jej lze označit za typ ostatním nepříbuzný. Tento MT není zobrazen v diagramech. U kmene B. pertussis, který je jediným zástupcem MT75 v souboru, byly rovněž zjištěny dosud neznámé typy antigenních determinant ptxP a prnA [63].
Obr. 1. Diagram eBURST, MLVA analýza kmenů B. pertussis, ČR, 1967–2015 (červen) Figure 1. eBURST diagram, MLVA analysis of B. pertussis strain collection, Czech Republic, 1967–2015 (June) 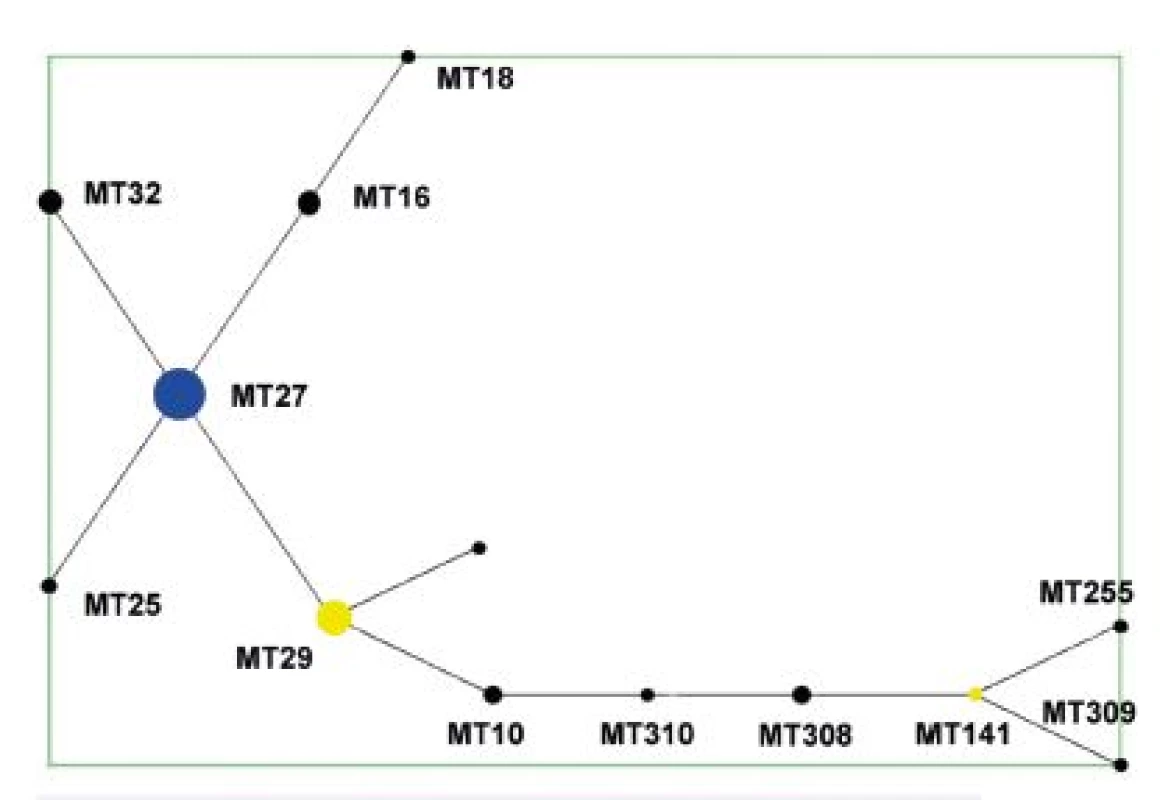
V diagramu není zobrazen singleton MT 75. Modrá barva označuje ancestrální MT (primary founder), žlutá barva označuje ancestrální MT v rámci podskupiny (subgroup founders). MT 75, the singleton, is not shown. Primary founders featured in blue, subgroup founders featured in yellow. Z diagramu eBURST pro soubor kmenů z období 1967–1980 je patrné, že ancestrálním MT zde byl MT141, zatímco MT29, zastoupený nejvyšším počtem kmenů, má ve skupině pouze 1 tzv. Single Locus Variant (SLV), MT10 – obrázek 2.
Obr. 2. Diagram eBURST, MLVA analýza kmenů <i>B. pertussis,</i> ČR, 1967–1980 Figure 2. eBURST diagram, MLVA analysis of <i>B. pertussis</i> strain collection, Czech Republic, 1967–1980 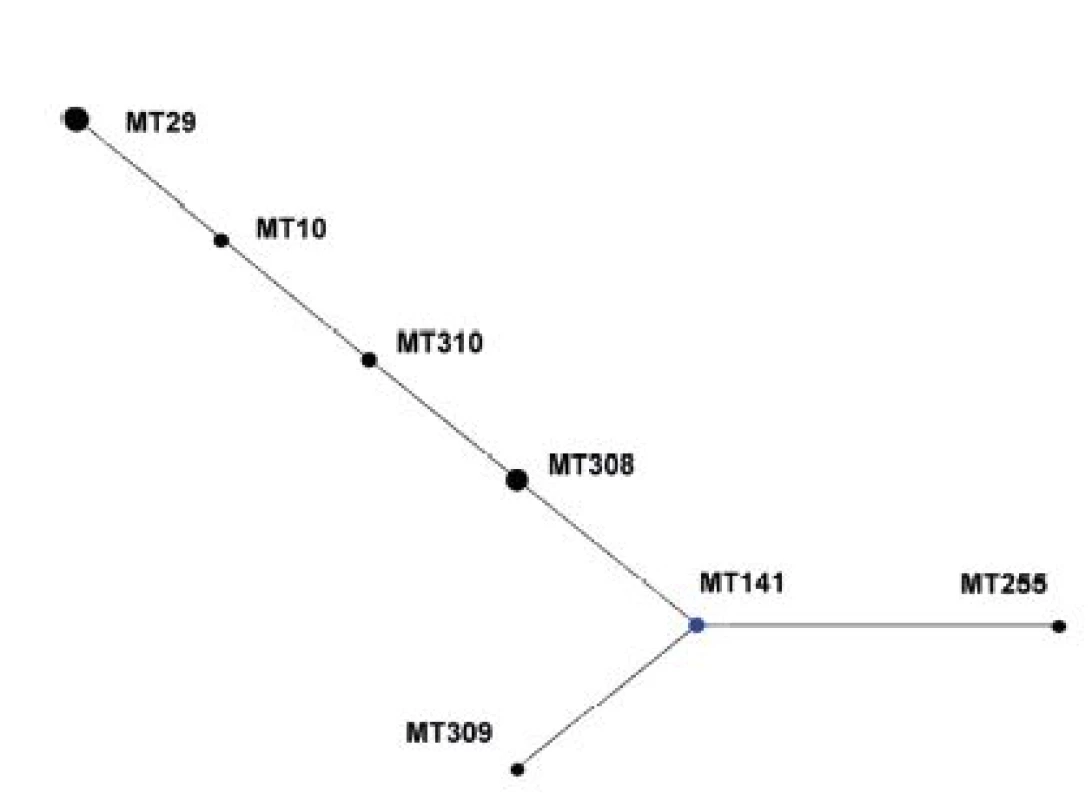
eBURST analýza souboru kmenů z období 1990–2007 nevyhodnotila žádný z MT jako ancestrální. MT27 a MT29 mají shodně po 2 SLV i Double Locus Variants (DLV) – obrázek 3.
Obr. 3. Diagram eBURST, MLVA analýza kmenů <i>B. pertussis,</i> ČR, 1990–2007 Figure 3. eBURST diagram, MLVA analysis of <i>B. pertussis</i> strain collection, Czech Republic, 1990–2007 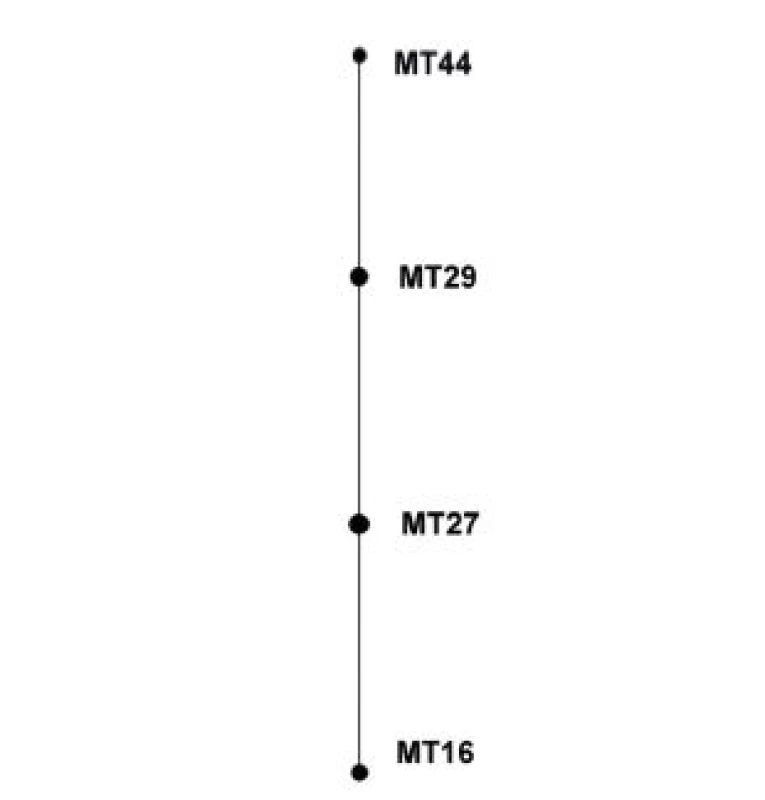
V souboru kmenů z období 2008–2015 (červen) byl jako ancestrální vyhodnocen MT27, který je současně zastoupen nejvyšším počtem kmenů a který byl rovněž vyhodnocen jako ancestrální MT pro celý soubor kmenů – obrázek 4.
Obr. 4. Diagram eBURST, MLVA analýza kmenů <i>B. pertussis,</i> ČR, 2008–2015 (červen) Figure 4. eBURST diagram, MLVA analysis of <i>B. pertussis</i> strain collection, Czech Republic, 2008–2015 (June) 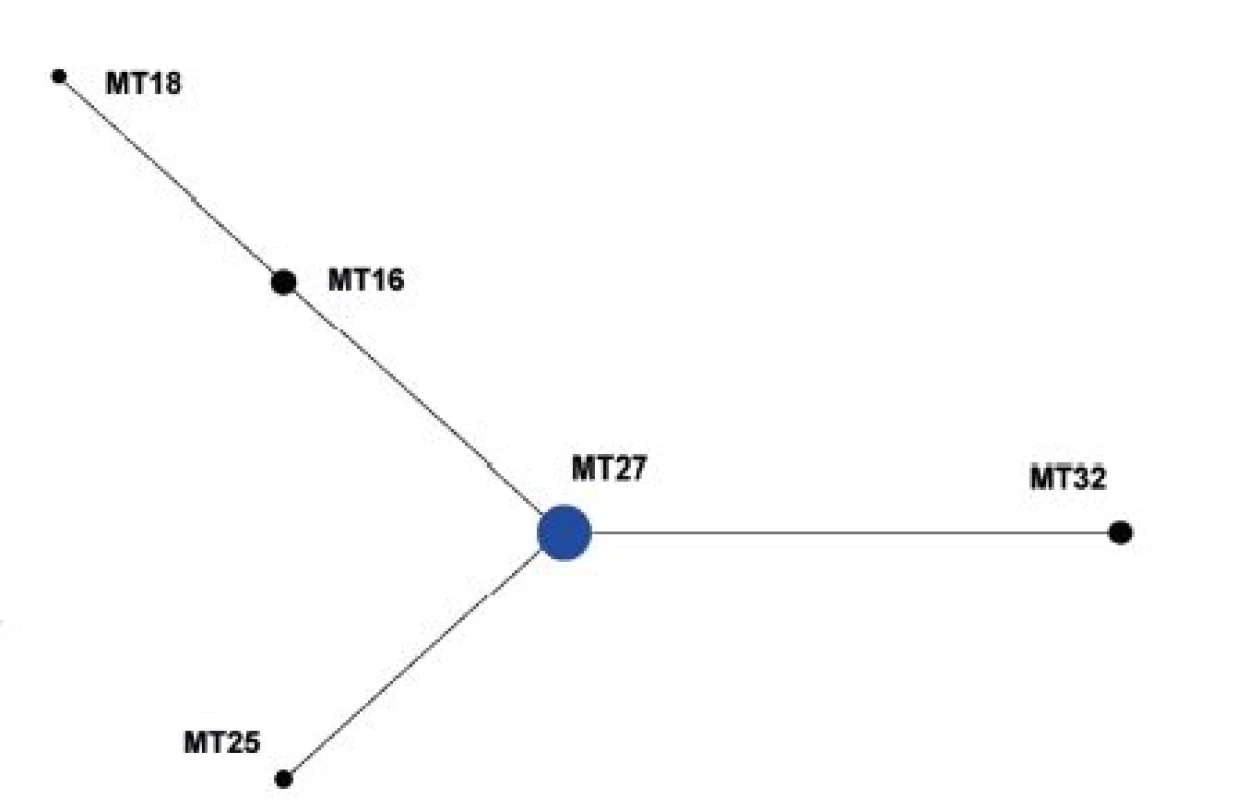
DISKUSE
Zpracování výsledků MLVA analýzy souboru 136 kmenů B. pertussis izolovaných z klinického materiálu na území ČR (a v případě pěti kmenů i dnešní SR) v období 1967–2015 ukázalo pokles genetické diverzity v tomto souboru v čase uplynulém od zahájení plošné vakcinace a zároveň změny v zastoupení MLVA typů.
Pět kmenů, u kterých byl dohledán původ na území dnešní Slovenské republiky, bylo v souboru ponecháno. Všechny tyto kmeny pocházejí z let 1968–1971, a spadají tedy do prvního sledovaného období. V té době byla na území ČSSR uplatňována jednotná vakcinační strategie, lze tedy předpokládat i selekční tlak obdobných mechanismů získané imunity.
DI je výrazně nižší v souboru kmenů z období 2007–2015, tedy z období vymezeného počátkem celoplošného očkování acelulární pertusovou vakcínou v ČR, oproti oběma předcházejícím obdobím. DI pro období 1967–1980 a 1990–2007 se významně neliší. Z těchto období byly pro analýzu k dispozici výrazně nižší počty kmenů než z recentní doby. Kmeny z doby před zahájením vakcinace (tedy před rokem 1958), z raného postvakcinačního období (1959–1966) a z období 1981–1989 k dispozici nebyly. V jiných zemích s dlouhodobě proočkovanou populací (USA, Velká Británie) byl zaznamenán pokles DI po zahájení vakcinace oproti období předcházejícímu [54], a rovněž vzestup genetické diverzity populace B. pertussis v období poklesu proočkovanosti [54, 55]. Na území ČR nebyl ve sledovaném období významný pokles proočkovanosti zaznamenán [1,2]. Z analýzy souboru kmenů z území ČR je patrný pouze pokles DI v období po zahájení plošného očkování acelulární vakcínou, který je ve shodě s výsledky obdobných studií ve světě [35, 54, 55].
V celém souboru (tvořícím 1 MLVA komplex a 1 singleton) četností výrazně převažují dva MT: MT27 a MT29. Vysoká prevalence těchto typů je patrná i na globální úrovni, pokud jde o země s dlouhodobě proočkovanou populací [47, 53, 54, 55, 56, 64]. Výjimkou jsou země s odlišnou vakcinační strategií, v Evropě především Polsko [53]. MT29 je nejčetněji zastoupen v prvním sledovaném období, po roce 1993 se již nevyskytuje. MT27 se naopak objevuje v období 1990–2007, kdy již převažuje, v následujícím období je pak jeho převaha ještě výraznější. Také tento vývoj je globálním trendem [47, 53, 54, 55]. V Polsku byl zaznamenán opačný vývoj, k dispozici však byly pouze kmeny izolované do roku 2006 [53].
V prvním období (1967–1980) byl zaznamenán výskyt 3 dosud nepopsaných MT, z toho MT308 je druhým nejčetněji zastoupeným MT. Regionálně unikátní MT byly popsány i v obdobných studiích z jiných zemí, v obdobích prevakcinačních a raně postvakcinačních [54, 55]. Nalezení těchto typů jen na jednom území může být zapříčiněno celkově vyšší heterogenitou a regionální růzností populací B. pertussis před vakcinací a krátce po jejím zahájení. Jiným možným vysvětlením je nemožnost porovnávat reprezentativní výběry kmenů z těchto období pro nízký počet dochovaných viabilních kmenů. MT29 a MT10 naopak vykazují globální výskyt. MT10 převažoval v prevakcinační éře ve Velké Británii [54], USA [55] a dalších zemích s dlouhodobě proočkovanou populací [56]. Mezi lety 1989–2007 byl tento typ zaznamenán mezi japonskými klinickými izoláty [56]. MT29, typ lišící se od MT10 pouze ve VNTR1, je ve světě dominujícím typem především v raném postvakcinačním období [54, 55]. V souboru kmenů z 12 evropských zemí je tento typ ještě zastoupen v období 1998–2001, poté již jeho výskyt nebyl zaznamenán, s výjimkou Polska. Příslušnost k tomuto MT korelovala u studovaných izolátů s přítomností ptxP1 alely promotoru genu pro pertusový toxin [47, 65]. Stejně tomu bylo i v souboru českých izolátů [63]. Kmen patřící k MT75 byl izolován v roce 1970. Jedná se o jediný kmen v souboru, který není součástí MLVA komplexu a jeví se jako nepříbuzný ostatním kmenům souboru. Nejde však o unikátní MT, tento typ se vyskytl již v rozmezí let 1920–1950 v USA a v roce 1993 u 1 izolátu z Francie [56].
V druhém období (1990–2007) již převažuje MT27. V roce 2007 je izolován první kmen patřící k MT16. Z tohoto období byl k dispozici nízký počet kmenů, což odpovídá tehdejšímu nízkému výskytu onemocnění [2].
Z třetího období bylo analyzováno 91 kmenů, oproti dvěma předcházejícím obdobím výrazně vyšší počet. Téměř 70 % těchto kmenů náleží k MT27.
Kmeny patřící k MT27 jsou vybaveny ptxP3 alelou promotoru genu pro pertusový toxin [47, 51, 66]. Kmeny nesoucí tuto alelu jsou v současnosti rozšířeny globálně a převažují ve většině zemí s dlouhodobě vakcinovanou populací [65]. Alela ptxP3 byla poprvé zachycena v Nizozemsku v roce 1988 [47], nárůst počtu kmenů nesoucích tuto alelu byl tamtéž spojen s nárůstem počtu hlášených onemocnění pertusí a s vyšším počtem hospitalizací a úmrtí [65]. Při porovnání s kmeny nesoucími alelu ptxP1 byla u ptxP3-kmenů prokázána vyšší produkce pertusového toxinu [65]. U myší byla potvrzena u ptxP3-kmenů B. pertussis lepší schopnost kolonizovat sliznici trachey a plicní parenchym [67]. Byly prokázány odlišnosti v regulaci exprese genů u ptxP1 - a ptxP3-kmenů [19,67]. Pro kmeny nesoucí ptxP3 je charakteristická zvýšená exprese mnoha faktorů virulence, což vede především k výraznějšímu potlačení imunitní odpovědi hostitele a usnadnění přenosu mezi dospělými jedinci, kteří se již setkali s antigenními strukturami B. pertussis, buď prostřednictvím vakcíny, nebo nákazou [13,19,27]. Z faktorů virulence se nejvýznamněji uplatňuje pertusový toxin, zodpovědný zejména za intoxikaci alveolárních makrofágů, potlačení mechanismů slizniční imunity a celulární i protilátkové odpovědi, Vag8, autotransportní protein odpovědný za rezistenci ke komplementu, a efektory transportního systému typu III (T3SS). K efektorům T3SS se řadí BteA, cytotoxin působící smrt epiteliálních buněk, a BopN, modulátor celulární odpovědi [19]. Analýzou SNP mutací bylo prokázáno, že ptxP1 - a ptxP3-kmeny patří k různým evolučním liniím [47]. Srovnávací genomickou hybridizací bylo prokázáno, že ptxP3 - kmeny vesměs neobsahují oblast genomu čítající 18 genů, tzv. RD-10 (Region of Difference), zatímco v genomu ptxP1-kmenů je tato oblast přítomna [47].
Celkově lze změny ve výskytu a zastoupení jednotlivých MT v české populaci B. pertussis od zahájení vakcinace charakterizovat jako posun od rozmanitější populace s významným zastoupením regionálně unikátních typů k populaci uniformnější, tvořené z velké části MT s globálním výskytem, s výraznou dominancí MT27. Tento vývoj je možné pozorovat v oblastech s dlouhodobě proočkovanou populací celosvětově [53, 54, 55, 56]. MT16 a MT27 jsou významně zastoupeny v celoevropské populaci B. pertussis. V USA a Velké Británii se od konce 90. let vyskytují MT27, MT16, MT18, MT32, MT25 [54,55]. Ve Velké Británii jejich poměrné zastoupení s časem narůstá [54]. V Japonsku se MT16, MT32, MT27 vyskytovaly v období 2002 a 2004, v dalších letech narůstá poměrné zastoupení MT27, ostatní zmíněné MT již v pozdějších obdobích nebyly zachyceny [51]. MT70, typ s významným poměrným zastoupením v Austrálii [56] a Polsku [53] a v letech 1998–2001 také ve Velké Británii [54], nebyl v souboru českých kmenů zachycen, stejně tak MT64, významně zastoupený v Austrálii [56] a MT186, častý v Japonsku [51].
Z výsledků MLVA analýzy po zpracování algoritmem eBURST nelze automaticky usuzovat na evoluční stáří a příbuznost kmenů patřících k jednotlivým MT. Jedná se o hypotetický model, který se může v různé míře lišit od reálných poměrů a vztahů evoluční příbuznosti ve studované populaci. Příčiny této možné nepřesnosti najdeme v každém z kroků, které vedou k výsledné grafické a číselné podobě získaných dat.
Prvním krokem je získání souboru bakteriálních kmenů pro testování. Tento soubor by ideálně měl být reprezentativním výběrem z celé populace daného bakteriálního druhu ve studovaném období a geografické oblasti. Studovaný soubor kmenů B. pertussis z tohoto pohledu ideální není, z období před rokem 2008 byl k dispozici nižší počet kmenů. Přesto však bylo v populaci B. pertussis z území ČR možné sledovat obdobné změny jako v populacích z jiných zemí s dlouhodobě proočkovanou populací.
Dalším krokem je samotná molekulární analýza, aplikace konkrétní metodiky cílící na specifické variabilní oblasti genomu. Variabilita zvolených oblastí je ovlivňována i mechanismy, které mohou vést jak ke shodným parametrům u nepříbuzných kmenů, tak i k opačné situaci. Mutace ve VNTR oblastech, vznikající mechanismem slipped strand mispairing, lze obecně považovat za náhodné [44]. Genom B. pertussis je vysoce monomorfní, přesto byl při srovnání MLVA analýzy s SNP analýzou prokázán konvergentní vývoj ve VNTR oblastech [68]. K tomuto vývoji dochází mechanismem homologní rekombinace nebo díky reverzibilitě procesu slipped strand mispairing [30, 32, 68]. Rekombinace obecně častěji vedou k poklesu diverzity populace než k jejímu vzestupu [69]. V případě doplnění MLVA analýzy analýzou SNP je možné nalézt evolučně nepříbuzné izoláty náležející k témuž MT [68].
Výhodou metody MLVA je snadná proveditelnost, dobrá úroveň rozlišení [46, 68, 70,] a především možnost snadného porovnání výsledků z různých pracovišť pomocí databáze všech dosud detekovaných MT [57]. Schouls et al. [46] prokázali, že změny v diverzitě populace detekované metodou MLVA předcházejí změny v diverzitě zachycené na podkladě sekvenační analýzy genů kódujících antigenní struktury (MAST). Jde tedy o metodu vhodnou i k molekulárně epidemiologické analýze, zejména větších souborů kmenů získaných v kratším časovém období [49, 50, 51, 66, 71]. MLVA umožňuje detekci změn diverzity v souboru oproti běžné populaci, signalizující například možnou klonální expanzi [46]. Metodou MLVA je možné analyzovat i vzorky DNA B. pertussis izolované přímo z materiálu z dýchacích cest, bez nutnosti kultivace [72]. Jak celosvětově, tak i v ČR je vlivem lepší dostupnosti molekulárních technik i pro rutinní diagnostiku stále obtížnější získávat kmeny B. pertussis pro podrobnější analýzu [3, 73].
Pro úspěšné zavedení typizační metody určené k rutinní epidemiologické analýze zvýšeného výskytu sledovaného onemocnění a k monitorování epidemiologického vývoje na určitém územním celku je nutné nejprve provést populačně genetickou analýzu dostupného souboru historických i současných klinických izolátů původce z daného území [58]. Takovým podkladem pro analýzu případného epidemického výskytu B. pertussis na území ČR mohou být výsledky této práce. Výrazná dominance jediného MT v souboru recentních izolátů je však pro takové využití limitující a lze předpokládat, že bude vhodné kombinovat MLVA typizaci s dalšími metodami [51]. V ČR v současné době připadá v úvahu již zavedená metoda sekvenační typizace MAST [38].
ZÁVĚR
MLVA analýzou souboru kmenů B. pertussis izolovaných z klinického materiálu na území ČR v období 1967–2015 byl prokázán pokles genetické diverzity ve studované populaci a změny v zastoupení a četnosti jednotlivých MT. Tyto změny lze charakterizovat jako postupný nárůst výskytu globálně rozšířených MT na úkor MT regionálně unikátních a postupné rozšíření a výrazně převažujícího MT27. Tento typ převažuje ve většině geografických oblastí, kde je populace dlouhodobě proočkovaná proti pertusi. Výsledky analýzy mohou být využívány i jako podklad pro aplikaci MLVA v epidemiologické analýze souborů menšího rozsahu. Pro analýzu epidemických výskytů B. pertussis v ČR je vhodné kombinovat metodu MLVA s další metodou molekulární typizace.
Poděkování
Práce byla podpořena výzkumným grantem NT/14058-3 Interní grantové agentury (IGA) MZ ČR.
Autoři děkují všem kolegům, kteří odesílají kmeny B. pertussis do NRL/DIPE, za vynikající spolupráci.
Děkujeme paní Haně Klemové a paní Janě Šimlové nejen za vzorné zpracování materiálu a technickou podporu celého projektu.
Do redakce došlo dne 30. 11. 2015.
Adresa pro korespondenci:
MUDr. Kateřina Fabiánová
Centrum epidemiologie a mikrobiologie
Státní zdravotní ústav
Šrobárova 48
110 00 Praha 10
e-mail: kfabianova@szu.cz
Zdroje
1. Kříž B, Fabiánová K, Maixnerová M, et al. Pertusse – navracející se infekce? Epidemiol Mikrobiol Imunol, 2007;56(2):51–65.
2. Fabiánová K, Beneš Č, Kříž B. Stálý nárůst incidence pertuse v České republice od 90. let. Epidemiol Mikrobiol Imunol, 2010;59(1):25–33.
3. Fabiánová K, Zavadilová J, Šebestová H, et al. Syndrom dávivého kašle. Pertuse a parapertuse v České republice v roce 2014 – rozbor epidemiologické situace. Zprávy CEM, 2015;24(5):172–176.
4. Tan T, Dalby T, Forsyth K, et al. Pertussis Across the Globe: Recent Epidemiologic Trends From 2000 to 2013. Pediatr Infect Dis J, 2015;34(9):e222–232.
5. Rohani P, Drake JM. The decline and resurgence of pertussis in the US. Epidemics, 2011;3(3–4):183–188.
6. Celentano LP, Massari M, Paramatti D, et al. Resurgence of pertussis in Europe. Pediatr Infect Dis J, 2005;24(9):761–765.
7. Clark T. Changing pertussis epidemiology: Everything old is new again. J Infect Dis, 2014 : 20(9):978–981.
8. de Melker HE, Schellekens JF, Neppelenbroek SE, et al. Reemergence of pertussis in the highly vaccinated population of the Netherlands: observations on surveillance data. Emerg Infect Dis, 2000;6(4):348–357.
9. Althouse BM, Scarpino SV. Asymptomatic transmission and the resurgence of Bordetella pertussis. BMC Med, 2015;13 : 146.
10. Bouchez V, Guiso N. Bordetella pertussis, B. parapertussis, vaccines and cycles of whooping cough. Pathog Dis, 2015;73(7). pii: ftv055.
11. Tartof SY, Lewis M, Kenyon C, et al. Waning immunity to pertussis following 5 doses of DTaP. Pediatrics, 2013;131(4):e1047–1052.
12. Lavine J, Bjornstad O, Freiesleben de Blasio B, et al. Short-lived immunity against pertussis, age-specific routes of transmission, and the utility of a teenage booster vaccine. Vaccine, 2012;30(3):544–551.
13. Mooi FR, Van der Maas NA, De Melker HE. Pertussis resurgence: waning immunity and pathogen adaptation – two sides of the same coin. Epidemiol Infect, 2014;142(4):685–694.
14. Smits K, Pottier G, Smet J, et al. Different T cell memory in preadolescents after whole-cell or acellular pertussis vaccination. Vaccine, 2013;32(1):111–118.
15. Edwards KM, Berbers GAM. Immune responses to pertussis vaccines and disease. J Infect Dis, 2014;209(S1):S10–15.
16. Cherry JD. Why do pertussis vaccines fail? Pediatrics, 2012;129(5):968–970.
17. Guiso N. Bordetella pertussis: why is it still circulating? J Infect, 2014;Suppl 1:S119–124.
18. Mooi FR, van Loo IHM, van Gent M, et al. Bordetella pertussis strain with increased toxin production associated with pertussis resurgence. Emerg Infect Dis, 2009;15(8):1206–1213.
19. de Gouw D, Hermans PW, Bootsma HJ, et al. Differentially expressed genes in Bordetella pertussis strains belonging to a lineage which recently spread globally. PLoS One, 2014;9(1):e84523.
20. Aguas R, Goncalves G, Gomes MG. Pertussis: increasing disease as a consequence of reducing transmission. Lancet Infect Dis, 2006;6(2):112–117.
21. Rohani P, Zhong X, King AA. Contact network structure explains the changing epidemiology of pertussis. Science, 2010;330(6006):982–985.
22. Jenkinson D. Increase in pertussis may be due to increased recognition and diagnosis. BMJ, 2012;345:e5463.
23. Paddock CJ, Sander GN, Cherry JD, et al. Pathology and pathogenesis of fatal Bordetella pertussis infection in infants. Clin Infect Dis, 2008;47 : 328–338.
24. Mattoo S, Cherry JD. Molecular Pathogenesis, Epidemiology, and Clinical Manifestations of Respiratory Infections Due to Bordetella pertussis and Other Bordetella Subspecies. Clin Microbiol Rev, 2005;18(2):326–382.
25. Cherry JD. Adult pertussis in the pre - and post-vaccine eras; lifelong vaccine-induced immunity? Expert Rev Vaccines, 2014;13(9):1073–1080.
26. Parkhill J, Sebaihia M, Preston A, et al. Comparative analysis of the genome sequences of Bordetella pertussis, Bordetella parapertussis and Bordetella bronchiseptica. Nature Genet, 2003;35(1):32–40.
27. Mooi FR. Bordetella pertussis and vaccination: The persistence of a genetically monomorphic pathogen. Infect Genet Evol, 2010;10(1):36–49.
28. Bouchez V, Caro V, Levillain E, et al. Genomic content of Bordetella pertussis clinical isolates circulating in areas of intensive children vaccination. PLoS ONE, 2008;3(6):e2437.
29. Brinig MM, Cummings CA, Sanden GN, et al. Significant gene order and expression differences in Bordetella pertussis despite limited gene content variation. J Bacteriol, 2006;188(7):2375–2382.
30. Spratt BG, Maiden MCJ. Bacterial population genetics, evolution and epidemiology. Philos Trans R Soc Lond B Biol Sci, 1999;354(1384):701–710.
31. Diavatopoulos DA, Cummings CA, Schouls LM, et al. Bordetella pertussis, the causative agent of whooping cough, evolved from a distinct, human-associated lineage of B. bronchiseptica. PLoS Pathog, 2005;1(4):e45.
32. King AJ, van Gorkom T, van der Heide HG, et al. Changes in the genomic content of circulating Bordetella pertussis strains isolated from the Netherlands, Sweden, Japan and Australia: adaptive evolution or drift? BMC Genomics, 2010;11 : 64.
33. Kallonen T, He Q. Bordetella pertussis strain variation and evolution postvaccination. Expert Rev Vaccines, 2009;8(7):863–875.
34. Bouchez V, Brun D, Cantinelli T, et al. First report and detailed characterization of B. pertussis isolates not expressing pertussis toxin or pertactin. Vaccine, 2009;27(43):6034–6041.
35. Bart MJ, Harris SR, Advani A, et al. Global population structure and evolution of Bordetella pertussis and their relationship with vaccination. mBio, 2014;5(2):e01074–14.
36. Packard ER, Parton R, Coote JG, et al. Sequence variation and conservation in virulence-related genes of Bordetella pertussis isolates from the UK. J Med Microbiol, 2004;53(Pt5):355–365.
37. Van Loo IHM, Heuvelman KJ, King AJ, et al. Multilocus sequence typing of Bordetella pertussis based on surface protein genes. J Clin Microbiol, 2002;40(6):1994–2001.
38. Zavadilová J, Lžičařová D, Musílek M, et al. Antigenní variabilita kmenů Bordetella pertussis izolovaných v letech 1967–2010 v České republice – možné vysvětlení vzestupu nemocnosti pertusí? Epidemiol Mikrobiol Imunol, 2015;64(3):130–138.
39. Mooi FR, van Loo IH, King AJ. Adaptation of Bordetella pertussis to vaccination: a cause for its reemergence? Emerg Infect Dis, 2001;7(3 Suppl):526–528.
40. Mooi FR, van Oirschot H, Heuvelman K, et al. Polymorphism in the Bordetella pertussis virulence factors P.69/Pertactin and Pertussis toxin in the Netherlands: temporal trends and evidence for vaccine-driven evolution. Infect Immun, 1998;66(2):670–675.
41. Mooi FR, He Q, van Oirschot H, et al. Variation in the Bordetella pertussis virulence factors pertussis toxin and pertactin in vaccine strains and clinical isolates in Finland. Infect Immun, 1999;67(6):3133–3134.
42. Lam C, Octavia S, Ricafort L, et al. Rapid increase in pertactin-deficient Bordetella pertussis isolates, Australia. Emerg Infect Dis, 2014;20(4):626–633.
43. Pawloski LC, Queenan AM, Cassiday PK, et al. Prevalence and molecular characterization of pertactin-deficient Bordetella pertussis in the United states. Clin Vacc Immunol, 2014;21(2):119–125.
44. van Belkum A, Scherer S, van Alphen L, et al. Short-sequence DNA repeats in prokaryotic genomes. Mic Mol Biol Rev, 1998;62(2):275–293.
45. van Gent M, Pierard D, Lauwers S, et al. Characterization of Bordetella pertussis clinical isolates that do not express the tracheal colonization factor. FEMS, 2007;51(1):149–154.
46. Schouls LM, van der Heide HGJ, Vauterin L, et al. Multiple-locus variable-number tandem repeat analysis of Dutch Bordetella pertussis strains reveals rapid genetic changes with clonal expansion during the late 1990s. J Bacteriol, 2004;186(16):5496–5505.
47. King AJ, van Gorkom T, Pennings JLA, et al. Comparative genomic profiling of Dutch clinical Bordetella pertussis isolates using DNA microarrays: identificaton of genes absent from epidemic strains. BMC Genomics, 2008;9 : 311.
48. Karami N, Helldal H, Welinder-Olsson C, et al. Sub-typing of extended-spectrum-β-lactamase-producing isolates from a nosocomial outbreak: application of 10-loci generic Escherichia coli multi-locus variable number tandem repeat analysis. PLoS ONE, 2013;8(12):e83030.
49. Bowden KE, Williams MM, Cassiday PK, et al. Molecular epidemiology of the pertussis epidemic in Washington state in 2012. J Clin Microbiol, 2014;52(10):3549–3557.
50. Galit SRL, Otsuka N, Furuse Y, et al. Molecular epidemiology of Bordetella pertussis in the Philippines in 2012–2014. Int J Infect Dis, 2015;35 : 24–26.
51. Miyaji Y, Otsuka N, Toyoizumi-Ajisaka H, et al. Genetic analysis of Bordetella pertussis isolates from the 2008–2010 epidemic in Japan. PLoS ONE, 2013;8(10):e77165.
52. Lindsted BA, Torpdahl M, Verqnaud G, et al. Use of multilocus variable-number tandem repeat analysis (MLVA) in eight European countries, 2012. Euro Surveill, 2013;18(4):20385.
53. van Gent M, Heuvelman CJ, van der Heide HG, et al. Analysis of Bordetella pertussis clinical isolates circulating in European countries during the period 1998–2012. Eur J Clin Microbiol Infect Dis, 2015;34(4):821–830.
54. Litt DJ, Neal SE, Fry NK. Changes in genetic diversity of the Bordetella pertussis population in the United Kingdom between 1920 and 2006 reflect vaccination coverage and emergence of a single dominant clonal type. J Clin Microbiol, 2009;47(3):680–688.
55. Schmidtke AJ, Boney KO, Martin SW, et al. Population diversity among Bordetella pertussis isolates, United States, 1935–2009. Emerg Infect Dis, 2012;18(8):1248–1255.
56. Kurniawan J, Maharjan RP, Chan WF, et al. Bordetella pertussis clones identified by multilocus variable-number tandem-repeat analysis. Emerg Infect Dis, 2010;16(2):297–300.
57. Multiple-Locus Variable number tandem repeat Analysis. Dostupné na www: www.mlva.net
58. van Belkum A, Tassios PT, Dijkshoorn L, et al. Guidelines for the validation and application of typing methods for use in bacterial epidemiology. Clin Microb Infect Dis, 2007;13(Suppl. 3):1–46.
59. Spratt BG, Hanage WP, Li B, et al. Displaying the relatedness among isolates of bacterial species - the eBURST approach. FEMS Microbiol Lett, 2004;241(2):129–134.
60. Hunter PR, Gaston MA. Numerical index of the discriminatory ability of typing systems: an application of Simpson´s idex of diversity. J Clin Microbiol, 1988;26(11):2465–2466.
61. Grundmann H, Hori S, Tanner G. Determining confidence intervals when measuring genetic diversity and the discriminatory abilities of typing methods for microorganisms. J Clin Microbiol, 2001;39(11):4190–4192.
62. Online tool for quantitative assessment of classification agreement. Dostupné na www: www.comparingpartitions.info
63. Musílek M. Osobní sdělení. Nepublikovaná data.
64. van Amersfoorth SCM, Schouls LM, van der Heide HGJ, et al. Analysis of Bordetella pertussis populations in European countries with different vaccination policies. J Clin Microbiol, 2005;43(6):2837–2843.
65. Mooi FR, van Loo IHM, van Gent M, et al. Bordetella pertussis strains with increased toxin production associated with pertussis resurgence. Emerg Infect Dis, 2009;15(8):1206–1213.
66. Lam C, Octavia S, Bahrame Z, et al. Selection and emergence of pertussis toxin promoter ptxP3 allele in the evolution of Bordetella pertussis. Infect Genet Evol, 2012;12(2):492–495.
67. King AJ, van der Lee S, Mohangoo A, et al. Genome-wide expression analysis of Bordetella pertussis isolates associated with a resurgence in pertussis: elucidation of factors involved in the increased fitness of epidemic strains. PLoS ONE, 2013;8(6):e66150.
68. van Gent M, Bart MJ, van der Heide HGJ, et al. SNP-based typing: a useful tool to study Bordetella pertussis populations. PLoS ONE, 2011;6(5):e20340.
69. van Belkum A, Struelens M, de Visser A, et al. Role of genomic typing in taxonomy, evolutionary genetics, and microbial epidemiology. Clin Microbiol Rev, 2001;14(3):547–560.
70. Advani A, van der Heide HGJ, Hallander HO, et al. Analysis of Swedish Bordetella pertussis isolates with three typing methods: characterization of an epidemic lineage. J Microbiol Methods, 2009;78(3):297–301.
71. Wagner B, Melzer H, Freymuller G, et al. Genetic variation of Bordetella pertussis in Austria. PLoS ONE, 2015;10(7):e0132623.
72. Litt DJ, Jauneikaite E, Tchipeva D, et al. Direct molecular typing of Bordetella pertussis from clinical specimens submitted for diagnostic quantitative (real-time) PCR. J Med Microbiol, 2012;61(Pt 12):1662–1668.
73. Vestrheim DF, Steinbakk M, Bjornstad M, et al. Recovery of Bordetella pertussis from PCR-positive nasopharyngeal sample sis dependent on bacterial load. J Clin Microbiol, 2012;50(12):4114–4115.
Štítky
Hygiena a epidemiologie Infekční lékařství Mikrobiologie
Článek vyšel v časopiseEpidemiologie, mikrobiologie, imunologie
Nejčtenější tento týden
2016 Číslo 2- Stillova choroba: vzácné a závažné systémové onemocnění
- Diagnostika virových hepatitid v kostce – zorientujte se (nejen) v sérologii
- Prokalcitonin: marker vhodný pro diagnostiku sepse i hodnocení antimikrobiální léčby
- Diagnostický algoritmus při podezření na syndrom periodické horečky
- Autoinflamatorní onemocnění: prognózu zlepšuje včasná diagnostika a protizánětlivá terapie
-
Všechny články tohoto čísla
- Multiple-locus variable number tandem repeat analýza souboru kmenů Bordetella pertussis v ČR z období 1967–2015: rozšíření varianty adaptované na proočkovanou populaci
- Zlepšení diagnostického přístupu použitím rekombinantních antigenů v detekci intratekální produkce protilátek IgM/IgG u dětí s lymeskou neuroboreliózou
-
Výskyt klíštěte obecného Ixodes ricinus a významných patogenů jím přenášených ve vybraných oblastech se zvýšeným počtem onemocnění klíšťovou encefalitidou v různých nadmořských výškách v České republice
Část I. Klíště obecné Ixodes ricinus a virus klíšťové encefalitidy - Vliv kyslíku na produkci endotoxinu u bakterií ze skupiny Bacteroides fragilis group izolovaných od pacientů s kolorektálním karcinomem
- Rozdíly ve fosforylaci Akt v CD4+ lymfocytech u patogenní a nepatogenní infekce SIV
- Popis familiární formy Creutzfeldt-Jakobovy nemoci
- Pamiatke Charlesa Nicolla, nositeľa Nobelovej ceny za fyziológiu a medicínu
- Kulaté narozeniny doc. Bohumíra Kříže
- Prestižní ocenění Světové organizace pro zdraví zvířat (OIE) získal letos za zásluhy v boji proti vzteklině poprvé v historii český veterinární lékař MVDr. Oldřich Matouch, CSc.
- Nové přístupy v kontrole vzestupu počtu onemocnění pertusí
- Pandemie infekce virem hepatitidy C
- Některé aspekty imunitního systému v patogenezi Alzheimerovy choroby
- Detekcia markerov bunkovej smrti ako nástroj na testovanie citlivosti baktérií na antibiotiká
- Molekulární a antigenní analýza hemaglutininu virů chřipky A H3N2 detekovaných v České republice v epidemické sezoně 2014/2015
- Epidemiologie, mikrobiologie, imunologie
- Archiv čísel
- Aktuální číslo
- Informace o časopisu
Nejčtenější v tomto čísle- Popis familiární formy Creutzfeldt-Jakobovy nemoci
- Nové přístupy v kontrole vzestupu počtu onemocnění pertusí
-
Výskyt klíštěte obecného Ixodes ricinus a významných patogenů jím přenášených ve vybraných oblastech se zvýšeným počtem onemocnění klíšťovou encefalitidou v různých nadmořských výškách v České republice
Část I. Klíště obecné Ixodes ricinus a virus klíšťové encefalitidy - Vliv kyslíku na produkci endotoxinu u bakterií ze skupiny Bacteroides fragilis group izolovaných od pacientů s kolorektálním karcinomem
Kurzy
Zvyšte si kvalifikaci online z pohodlí domova
Revma Focus: Spondyloartritidy
nový kurz
Autoři: prof. MUDr. Vladimír Palička, CSc., Dr.h.c., doc. MUDr. Václav Vyskočil, Ph.D., MUDr. Petr Kasalický, CSc., MUDr. Jan Rosa, Ing. Pavel Havlík, Ing. Jan Adam, Hana Hejnová, DiS., Jana Křenková
Autoři: MUDr. Irena Krčmová, CSc.
Všechny kurzyPřihlášení#ADS_BOTTOM_SCRIPTS#Zapomenuté hesloZadejte e-mailovou adresu, se kterou jste vytvářel(a) účet, budou Vám na ni zaslány informace k nastavení nového hesla.
- Vzdělávání



