-
Články
Top novinky
Reklama- Vzdělávání
- Časopisy
Top články
Nové číslo
- Témata
Top novinky
Reklama- Kongresy
- Videa
- Podcasty
Nové podcasty
Reklama- Kariéra
Doporučené pozice
Reklama- Praxe
Top novinky
ReklamaSurveillance invazivního meningokokového onemocnění založená na sekvenaci celého genomu (WGS), Česká republika, 2015
Surveillance of invasive meningococcal disease based on whole genome sequencing (WGS), Czech Republic, 2015
Aim:
To test the potential of whole genome sequencing (WGS) for molecular surveillance of invasive meningococcal disease in the Czech Republic. To check the success of the new method in the identification of gene and protein variants and to compare the outcomes between WGS and conventional sequencing methods.Material and methods:
WGS was carried out in a set of 20 N. meningitidis isolates from invasive meningococcal disease cases in the Czech Republic in 2015. WGS was performed using the Illumina MiSeq platform. The WGS data were processed by the Velvet de novo Assembler software, and the resultant genome contigs were submitted to the Neisseria PubMLST web database containing allelic and genomic data on strains of the genus Neisseria. The genomes were analysed and compared using the BIGSdb Genome Comparator, which is part of the PubMLST database. WGS data were compared at several levels of resolution: MLST (Multi Locus Sequence Typing), rMLST (ribosomal MLST), cgMLST (core genome MLST), and "all loci", i.e. all genes of N. meningitidis defined in the PubMLST database by 6 November 2017 (3028 loci). The WGS method was used to characterise in detail the genes of antigens involved in vaccines against N. meningitidis B.Results:
The new WGS method provided detailed characteristics of N. meningitidis isolates, which improved the results obtained previously by conventional sequencing methods. High quality WGS data made it possible to identify novel alleles and novel sequence types that could not be recognized by conventional sequencing methods. The analysis of genetic diversity confirmed closer relatedness between isolates belonging to the same clonal complex. The most accurate information on genetic diversity of isolates was obtained by the comparison of WGS data at the cgMLST and "all loci" levels. Distant relatedness of three clonal complexes (cc32, cc35, and cc269) was found. WGS data also provided more accurate information on the coverage of isolates by MenB vaccines in comparison with conventional sequencing data.Conclusions:
The WGS method showed a higher discrimination potential and allowed a more accurate determination of genetic characteristics of N. meningitidis. The integration of the WGS method in routine molecular surveillance of invasive meningococcal disease in the Czech Republic is desirable.Keywords:
invasive meningococcal disease – genomic surveillance – whole genome sequencing (WGS)
Autoři: P. Křížová
; M. Honskus
; Z. Okonji
; M. Musílek
; J. Kozáková
Působiště autorů: Národní referenční laboratoř pro meningokokové nákazy, Centrum epidemiologie a mikrobiologie, Státní zdravotní ústav, Praha
Vyšlo v časopise: Epidemiol. Mikrobiol. Imunol. 67, 2018, č. 2, s. 64-73
Kategorie: Původní práce
Souhrn
Cíl práce:
Ověření možnosti využití metody sekvenace celého genomu (WGS) v molekulární surveillance invazivního meningokokového onemocnění v České republice. Určení úspěšnosti nové metody při stanovování genových a proteinových variant a její porovnání s klasickými sekvenačními metodami.Materiál a metodiky:
Pro celogenomovou sekvenaci byl vybrán soubor dvaceti izolátů N. meningitidis z invazivních meningokokových onemocnění, která byla zachycena v České republice v roce 2015. WGS proběhla na platformě Illumina MiSeq. Celogenomová data byla upravena pomocí softwaru Velvet de novo Assembler a výsledné genomové kontigy byly poslány do Neisseria PubMLST webové databáze obsahující alelové a genomické údaje kmenů rodu Neisseria. Analýza a vzájemné porovnání genomů proběhlo s využitím BIGSdb Genome Comparator, který je součástí databáze PubMLST. Data WGS byla porovnána na několika úrovních rozlišení: MLST (Multi Locus Sequence Typing), rMLST (ribozomální MLST), cgMLST (core genome MLST) a “all loci“ tj. všech definovaných genů pro N. meningitidis v databázi PubMLST k 6. 11. 2017 (3 028 lokusů). Metodou WGS byla provedena detailní charakterizace genů antigenů zařazených do vakcín proti N. meningitidis B.Výsledky:
Nová metoda WGS poskytla detailní charakterizaci izolátů N. meningitidis, která zpřesnila předchozí výsledky získané klasickými sekvenačními metodami. Kvalitnější WGS data umožnila identifikaci nových alel a nových sekvenačních typů, které nebyly klasickými sekvenačními metodami rozpoznány. Analýza genetické diverzity potvrdila vyšší příbuznost mezi izoláty, které patří k jednomu klonálnímu komplexu. Nejpřesnější informace o genetické diverzitě izolátů poskytlo srovnání dat WGS na úrovni schémat cgMLST a “all loci“. Byla zjištěna vzdálená příbuznost tří klonálních komplexů (cc32, cc35 a cc269). WGS data také poskytla přesnější informace o pokrytí izolátů novými vakcínami proti N. meningitidis B, ve srovnání s klasickou sekvenací.Závěry:
WGS metoda prokázala vyšší diskriminační možnosti a přesnější určení genetických vlastností N. meningitidis. Zařazení metody WGS do rutinní molekulární surveillance invazivního meningokokového onemocnění v České republice je žádoucí.Klíčová slova:
invazivní meningokokové onemocnění – genomová surveillance – sekvenace celého genomu (WGS)ÚVOD
Invazivní meningokokové onemocnění (IMO) je celosvětově vnímáno jako závažné, život ohrožující onemocnění, přestože jeho incidence má v posledních letech sestupný trend [24]. Co však zůstává vysoké, je smrtnost tohoto onemocnění a procento celoživotních následků u přeživších. Jedinou účinnou prevencí tohoto závažného onemocnění je vakcinace. Volba vhodné vakcíny a správná vakcinační strategie je pro dostatečnou ochranu nezbytná. Současný vývoj nových vakcín proti meningokokovým onemocněním přináší další možnosti: kromě vakcín proti séroskupinám A, C, Y a W jsou nově k dispozici i vakcíny proti Neisseria meningitidis B (MenB vakcíny) [1, 18, 20].
Podkladem účinné vakcinační strategie je provádění kvalitní surveillance IMO. Kvalita surveillance IMO je výrazně zlepšována zaváděním molekulární charakterizace izolátů N. meningitidis. Zpočátku se jednalo o metody klasické sekvenace: multilokusovou sekvenaci (Multi--Locus Sequence Typing, MLST), sekvenaci variabilních oblastí genů porA, porB, fetA a genů antigenů MenB vakcín [6]. Nově je k dispozici metoda sekvenace celého genomu, Whole Genome Sequencing (WGS), která má absolutní diskriminační schopnost a ukazuje se navíc i jako cenově výhodnější v porovnání s klasickými sekvenačními metodami [13, 14, 15]. WGS přesněji určuje geny antigenů nových MenB vakcín [5] a data WGS perspektivně představují korelát k protilátkové odpovědi na MenB vakcíny [19]. WGS má potenciál klasické sekvenační metody významným způsobem zpřesnit a detailní molekulární charakterizaci meningokoků zefektivnit. Zařazení WGS do surveillance infekčních onemocnění je doporučeno Evropským centrem pro kontrolu infekčních onemocnění (ECDC) a má v Evropě v posledních letech významný vzestupný trend [21].
Nemocnost IMO v České republice je hlášena od roku 1943 a od roku 1993 jsou k dispozici data celorepublikové aktivní surveillance. Tato data surveillance IMO jsou každoročně publikována ve Zprávách Centra epidemiologie a mikrobiologie Státního zdravotního ústavu.
Izoláty z IMO jsou zasílány z mikrobiologických laboratoří celé republiky do Národní referenční laboratoře pro meningokokové nákazy (NRL) k ověření a detailní charakterizaci. Od zahájení aktivní surveillance IMO v roce 1993 jsou do NRL posílány izoláty z více než 60 % onemocnění. Proporce izolátů z IMO je ovlivněna skutečností, že kolem 30 % IMO je v posledních letech potvrzeno pouze bezkultivační metodou polymerázové řetězové reakce (Real-Time Polymerase Chain Reaction, RT-PCR). Molekulární charakterizace N. meningitidis klasickými sekvenačními metodami byla v NRL zavedena v roce 2000 [16] a výsledky jsou hlášeny do mezinárodních databází: PubMLST (Public databases for molecular typing and microbial genome diversity) [10], EMERT (European Meningococcal Epidemiology in Real Time) [8] a TESSy (The European Surveillance System) [11]. NRL v rámci řešení výzkumného projektu zavedla v roce 2015 metodu WGS s cílem její implementace do celorepublikové surveillance IMO [22].
Tato práce prezentuje první použití metody WGS v molekulární surveillance IMO v České republice.
MATERIÁL A METODY
Bakteriální izoláty a extrakce DNA
Pro celogenomovou sekvenaci byl vybrán soubor dvaceti izolátů N. meningitidis z IMO zaznamenaných v programu surveillance v České republice za rok 2015. Mezi jednotlivými případy IMO nebyla zjištěna žádná epidemiologická souvislost. Příslušné bakteriální kultury, které jsou uchovávány v -80 °C (Kryobanka B, ITEST), byly vyočkovány na čokoládový Mueller-Hinton agar a kultivovány 18–24 hodin ve 37 °C a 5% CO2 atmosféře. Séroskupiny izolátů byly určeny klasickými sérologickými metodami a ověřovány metodou RT-PCR. Dalším krokem byla izolace deoxyribonukleové kyseliny (DNA), k níž byl použit izolační kit QIAamp DNA Mini Kit (QIAGEN). Postup izolace probíhal podle pokynů výrobce. Všechny izoláty byly poté charakterizovány klasickými sekvenačními metodami.
Celogenomová sekvenace a zpracování WGS dat
Celogenomová sekvenace dvaceti vybraných izolátů byla realizována externě – na pracovišti EMBL (European Molecular Biology Laboratory, Heidelberg, Německo). Sekvenace proběhla na platformě Illumina MiSeq s využitím referenčního genomu N. meningitidis kmen MC58. Výsledkem byly překrývající se sekvence o délce přibližně 300 bp. Celogenomová data byla následně v NRL zpracována pomocí softwaru Velvet de novo Assembler. Pro optimalizaci tohoto procesu byl použit Velvet-Optimiser script [25]. U jednotlivých izolátů se parametr „K-mer lenght“ pohyboval mezi hodnotami 73 a 111. Výsledné genomové kontigy byly poté poslány do Neisseria PubMLST databáze [10], která využívá platformu BIGSdb (Bacterial Isolate Genome Sequence Database) [12], pod označením Neisseria PubMLST IDs: 35105, 35107, 36325, 36329, 36673, 36674, 38267, 38268, 38276, 38278, 38897, 38899, 38901, 38989, 38990, 40373, 40376, 40377, 41191, 41412.
Genomická analýza a vizualizace WGS dat
Genomové kontigy jednotlivých izolátů byly v PubMLST databázi automaticky skenovány a charakterizovány alelovým profilem genů, které se v NRL standardně určují klasickými sekvenačními metodami (abcZ, adk, aroE, fumC, gdh, pdhC, pgm, porA, fetA, nhba, nadA, fhbp). Na základě alelového profilu sedmi MLST genů byl u izolátů určen sekvenační typ (ST) a příslušnost ke klonálnímu komplexu (cc). U genů porA a fetA byly stanoveny alelové varianty ve variabilních regionech (VR), které tyto geny obsahují (3krát pro porA a 1krát pro fetA). Dále byly stanoveny alelové a peptidové varianty antigenů zahrnutých v MenB vakcínách (nhba, nadA, fhbp) [2, 4, 17, 19, 20, 23]. Nové genové a peptidové varianty byly skenovány manuálně, přidány do databáze, následně anotovány a očíslovány díky automatickému vkládacímu nástroji platformy BIGSdb.
Analýza a vzájemné porovnání genomů proběhlo s využitím BIGSdb Genome Comparator, který je součástí databáze PubMLST [12]. Celogenomová data izolátů byla porovnána na několika úrovních rozlišení: srovnání na úrovni schématu MLST genů (7 lokusů); schématu, které pokrývá geny ribozomální multilokusové sekvenace – rMLST (53 lokusů); schématu „N. meningitidis core genome MLST v1.0“ – cgMLST (1605 lokusů); schématu “all loci“, tj. všech definovaných genů pro N. meningitidis v databázi PubMLST k 6. 11. 2017 (3028 lokusů) [3, 7, 12, 13, 14].
„Distance matrix“, které jsou založeny na počtu a variabilitě alel genů obsažených v jednotlivých schématech, byly automaticky vygenerovány a fylogenetické sítě byly vytvořeny v programu SplitsTree4, který využívá algoritmus NeighborNet. Výsledky fylogenetických analýz byly následně graficky editovány v programu Inkscape.
VÝSLEDKY A DISKUSE
1 Molekulární charakterizace izolátů metodou WGS
V souboru 20 izolátů z IMO roku 2015 převažovala séroskupina B (15 izolátů), následovaná séroskupinou C a W (obě po 2 izolátech). U jednoho izolátu nebylo možno séroskupinu určit (NG, non-groupable) – tabulka 1. Nejčastěji zjištěnými klonálními komplexy v souboru izolátů byly komplexy cc32 a cc41/44. Jedná se o typické klonální komplexy séroskupiny B. Na druhém místě v četnosti výskytu byl hypervirulentní klonální komplex cc11, typický pro séroskupinu C a komplexy cc35 a cc269, typické pro séroskupinu B. Ostatní detekované klonální komplexy byly zastoupeny vždy jedním izolátem: cc18, cc60, cc162, cc213 (séroskupina B), cc22 a cc865 (séroskupina W). Jeden izolát séroskupiny B nebyl databází BIGSdb přiřazen k žádnému známému klonálnímu komplexu (ccUA, unassigned). Izolát, u kterého nebylo možné určit séroskupinu (non-groupable, NG) vykazoval příslušnost ke komplexu cc41/44. U genů porA a fetA byla pozorována vysoká heterogenita v jejich variabilních regionech u všech izolátů.
Tab. 1. Tabulka 1. Molekulární charakterizace 20 izolátů IMO z roku 2015, ČR
Table 1. Molecular characteristics of 20 isolates from IMD from 2015, Czech Republic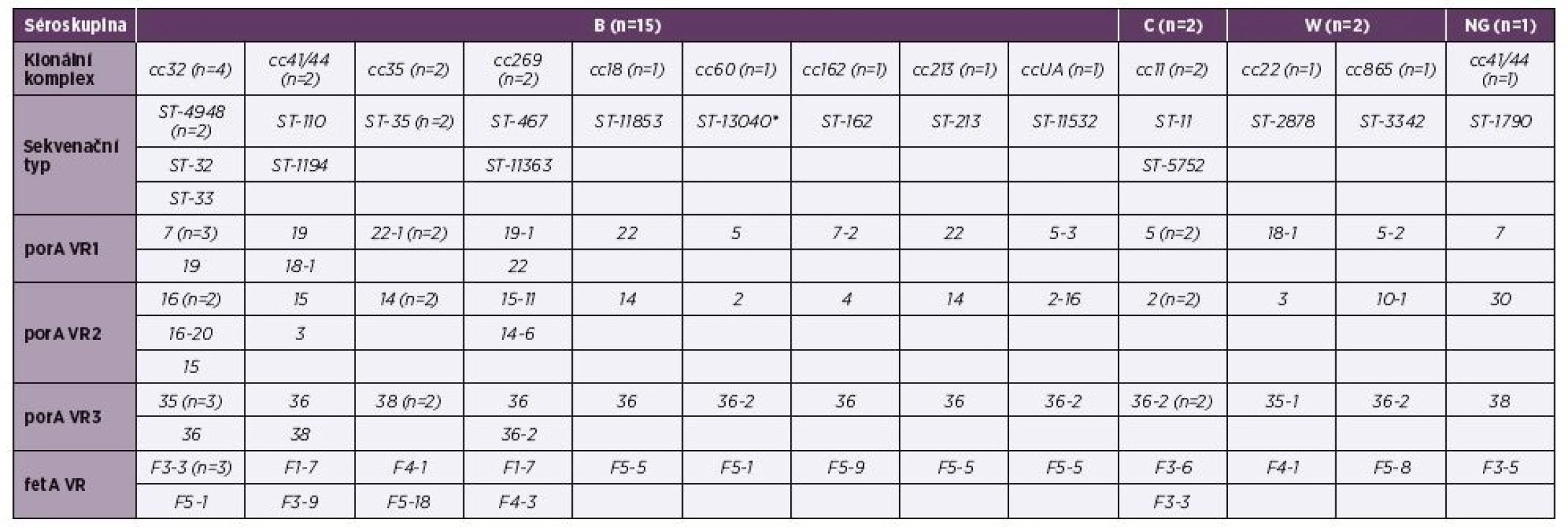
NG = non-groupable = izolát, u kterého nebylo možné určit séroskupinu
UA = unassigned = izolát, který není zařaditelný do klonálního komplexu
* = nově popsaný sekvenační typ
porA VR1,2,3 = variabilní region 1, 2, 3 genu porA; fetA VR = variabilní region genu fetA2 Genetická diverzita izolátů zjištěná metodou WGS
2.1 Srovnání na úrovni schématu MLST
Dva izoláty séroskupiny B klonálního komplexu cc35 jeví na rozlišovací úrovni MLST naprostou shodu a blízkou příbuznost ke klonálnímu komplexu cc32 (séroskupina B). Klonální komplex cc32 vykazuje větší různorodost, nicméně izoláty 90/15, B, cc32 a 14/16, B, cc32 jsou totožné. Dobře odlišené (ovšem mnohem méně vzájemně příbuzné) jsou dva izoláty klonálního komplexu cc269 (séroskupina B). Tři klonální komplexy séroskupiny B (cc32, cc35, cc269) jsou si vzájemně více příbuzné než ostatní. Zcela oddělené jsou vzájemně vysoce příbuzné izoláty séroskupiny C (cc11). Dobře vymezené jsou i izoláty klonálního komplexu cc41/44 (séroskupina B a NG), s přihlédnutím k předchozím klonálním komplexům jsou ale mezi sebou více rozmanité. Ostatní izoláty séroskupin B a W (každý zastoupený jedním cc) jsou postaveny samostatně a relativně nepříbuzné, nicméně vykazují větší příbuznost k sobě navzájem a ke klonálnímu komplexu cc41/44 než ke klonálním komplexům cc32, cc35 a cc269 – obrázek 1. Měřítko ukazuje poměrně malou schopnost rozlišení izolátů na úrovni sedmi MLST genů.
Obr. 1. Vizualizace genetické diverzity na úrovni schématu MLST u 20 izolátů z IMO, 2015, ČR
Figure 1. Visualisation of genetic diversity at the MLST level in 20 isolates from IMD, Czech Republic 2015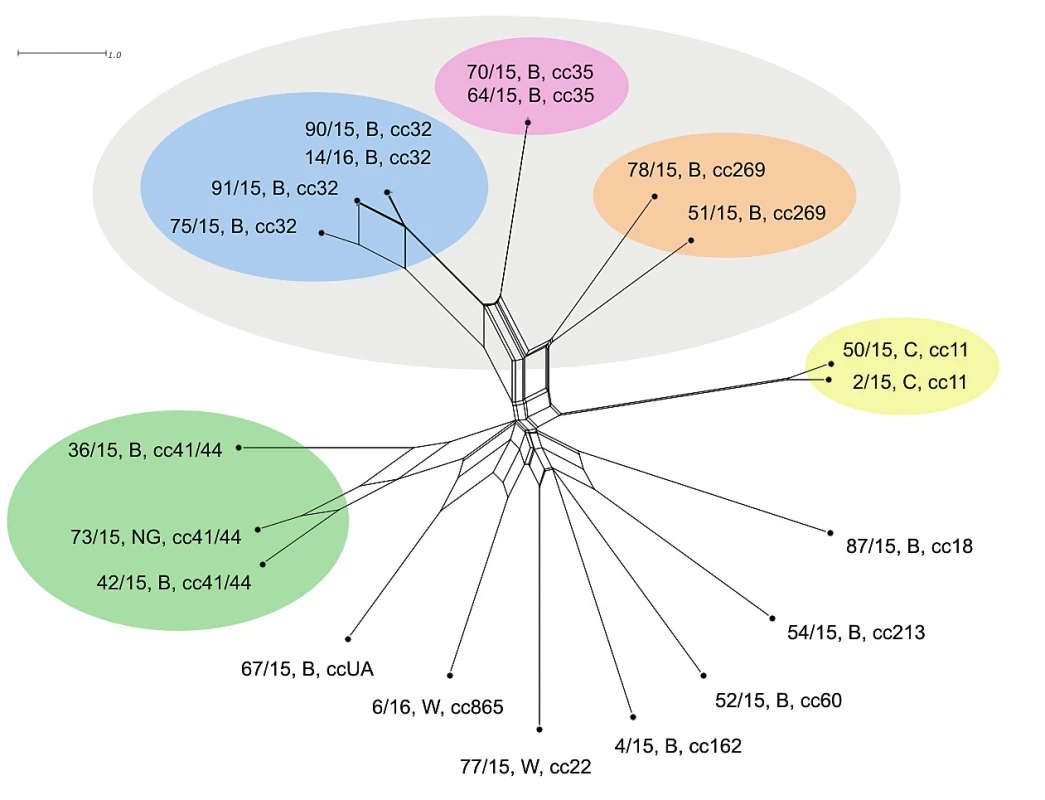
2.2 Srovnání na úrovni schématu rMLST
Dva izoláty klonálního komplexu cc35 již nevykazují při vyšší úrovni rozlišení naprostou shodu, nicméně jsou stále vysoce příbuzné a příbuzensky vysoce vzdálené od ostatních izolátů. U klonálního komplexu cc32 se ještě zvýraznilo odlišení od ostaních izolátů a rozrůznily se dva izoláty, které s nižším rozlišením vykazovaly 100% shodu. Izolát 51/15, B, cc269 se jeví jako příbuznější izolátům klonálního komplexu cc32 než k izolátu stejného klonálního komplexu 78/15, B, cc269, který se příbuzensky významně vzdálil. Izoláty klonálních komplexů cc32, cc35 a cc269 nicméně stále vykazují vzájemnou příbuznost. Izoláty séroskupiny C (cc11) opět stojí zcela odděleně a vykazují vysokou vzájemnou příbuznost (s vyšším rozlišením je však příbuznost menší). Klonální komplex cc41/44 je dobře vymezený, ale vykazuje nižší vzájemnou příbuznost izolátů – výsledek odpovídá úrovni MLST rozlišení, pouze se jednotlivé izoláty více rozrůznily. Ostatní izoláty séroskupin B a W opět stojí převážně izolovaně. Nicméně izoláty 54/15, B, cc213 a 52/15, B, cc60 vykazují vzdálenou příbuznost a zajímavostí je izolát 6/16, W, cc865, který se na této úrovni rozlišení jeví jako vzáleně příbuzný ke klonálnímu komplexu cc11 (séro-skupina C) – obrázek 2. Měřítko ukazuje zhruba trojnásobnou schopnost rMLST v rozlišení izolátů oproti rozlišení na úrovni sedmi MLST genů.
Obr. 2. Vizualizace genetické diverzity na úrovní schématu rMLST u 20 izolátů z IMO, 2015, ČR
Figure 2. Visualisation of genetic diversity at the rMLST level in 20 isolates from IMD, Czech Republic 201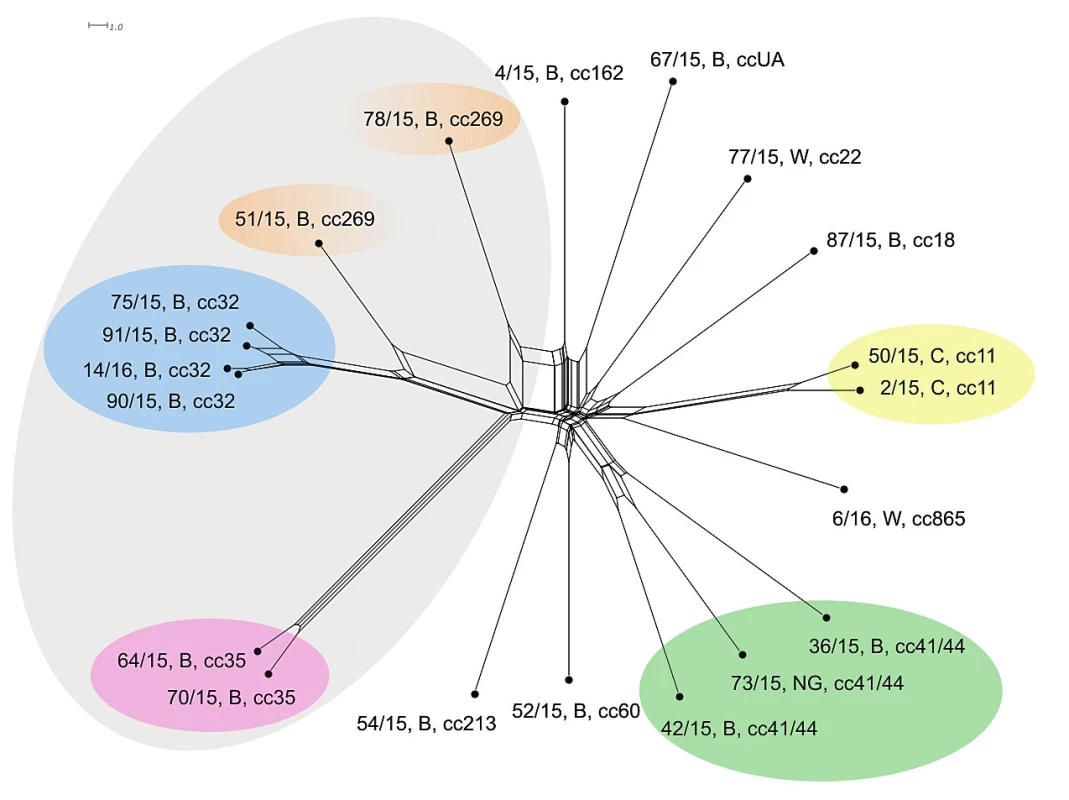
2.3 Srovnání na úrovni schématu cgMLST
Jedná se již o vysoce přesné rozlišení – měřítko značí zhruba 60násobné rozlišení cgMLST oproti MLST úrovni. Potvrdila se vzdálená příbuznost tří klonálních komplexů (cc32, cc35 a cc269). Komplex cc32 je vysoce homogenní (více než na předchozích úrovních rozlišení) a dobře znázorňuje příbuznost mezi jednotlivými izoláty. Dva izoláty klonálního komplexu cc35 jsou opět dobře vymezené, jasně oddělené od ostatních, vzájemně hodně příbuzné a jsou evolučně bližší komplexu cc32, než komplex cc269. Izoláty klonálního komplexu cc269 sice vykazují vzáleného společného předka, nicméně jsou již evolučně velice rozrůzněné. Izoláty séroskupiny C (cc11) opět stojí odděleně a dále vykazují velkou vzájemnou příbuznost (ta ovšem značně poklesla v porovnání s předchozími úrovněmi srovnání s nižším rozlišením). Zřetelně jsou vymezeny také izoláty komplexu cc41/44. Společný původ je u nich průkazný, ovšem jsou evolučně již značně rozrůzněny (podobně jako izoláty cc269). Zajímavé je, že vyšší úroveň rozlišení setřela předchozí náznaky vyšší příbuznosti u dvou ze tří izolátů cc41/44. Nyní je zřejmé, že všechny tři izoláty cc41/44 jsou evolučně vzdáleny (vzájemně odlišné) v podstatě stejně. Všechny ostatní izoláty (séro-skupin B a W) stojí dále izolovaně a vykazují vyšší podobnost k sobě navzájem a ke komplexu 41/44, než k izolátům klonálních komplexů cc32, cc35 a cc269 – obrázek 3.
Obr. 3. Vizualizace genetické diverzity na úrovni schématu cgMLST u 20 izolátů z IMO, 2015, ČR
Figure 3. Visualisation of genetic diversity at the cgMLST level in 20 isolates from IMD, Czech Republic 2015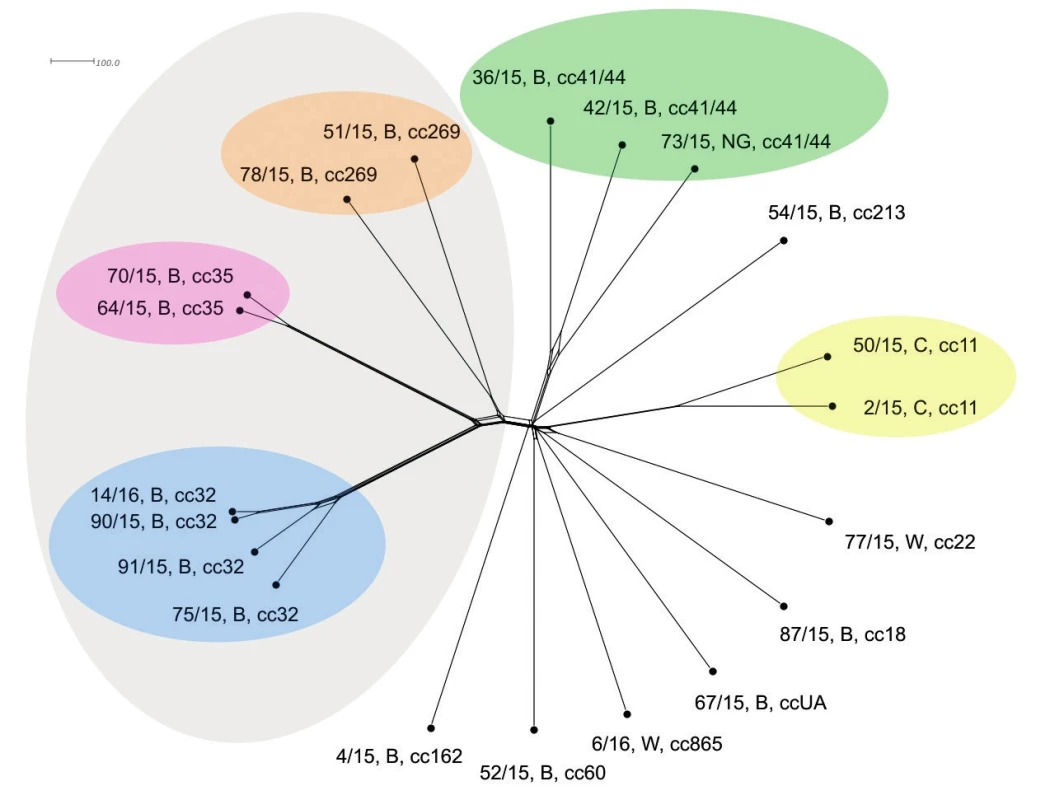
2.4 Srovnání na úrovni schématu “all loci“
Srovnání sekvencí na úrovni schématu “all loci“ nabízí jen nepatrně vyšší rozlišení a neposkytuje další informace ve srovnání s rozlišovací schopností schématu cgMLST – obrázek 4.
Obr. 4. Vizualizace genetické diverzity na úrovni schématu “all loci“ u 20 izolátů z IMO, 2015, ČR
Figure 4. Visualisation of genetic diversity at the all loci level in 20 isolates from IMD, Czech Republic 2015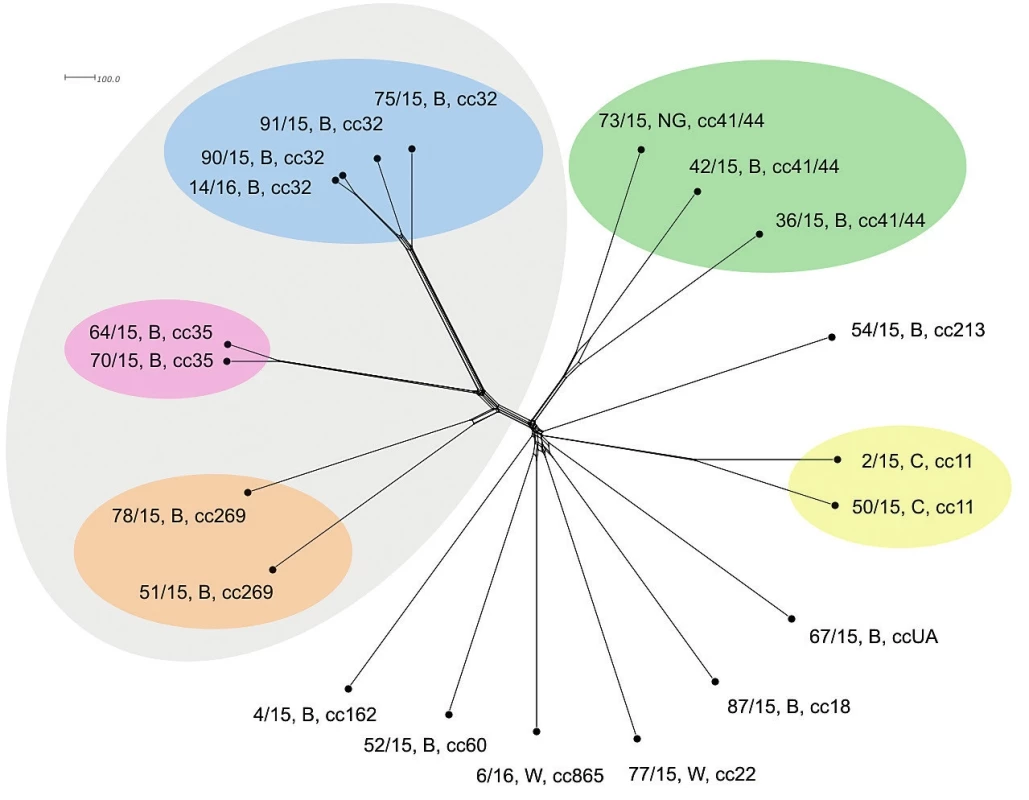
3 Molekulární charakterizace genů antigenů MenB vakcín metodou WGS
V současné době je k dispozici čtyřkomponentní vakcína (MenB-4C) a dvousložková vakcína (MenB-fHbp). Vakcína MenB-4C obsahuje proteinové produkty 4 genů: fHbp (factor H binding protein), NHBA (Neisseria Heparin Binding Antigen), NadA (Neisserial adhesin A) a protein zevní buněčné membrány (OMP) PorA P1.4. Vakcína MenB-fHbp obsahuje dvě různé peptidové varianty genu fHbp. Metodou WGS byly určeny alelové a peptidové varianty genů těchto antigenů.
3.1 Gen porA
U genu porA jsou klíčové dva variabilní regiony – VR1 a VR2. Vakcína MenB-4C obsahuje peptid s VR2 alelové varianty 4, vakcína MenB-fHbp žádný produkt tohoto genu neobsahuje [23]. U genu porA se podařilo metodou WGS určit alelové varianty VR1 a VR2 u všech izolátů – tabulka 2. Bylo zjištěno celkem 10 různých variant pro VR1 (5, 5-2, 5-3, 7, 7-2, 18-1, 19, 19-1, 22 a 22-1) a 12 různých variant pro VR2 (2, 2-16, 3, 4, 10-1, 14, 14-6, 15, 15-11, 16, 16-20, 30). Rozložení alelových variant bylo tedy vysoce variabilní, nicméně častěji se objevují varianty VR1 – 7 (4krát), 5 (3krát) a 22 (3krát) a v případě VR2 varianty 2 (3krát) a 14 (3krát). Podobné charakteristiky genu porA v oblastech VR1 a VR2 lze vypozorovat u izolátů klonálních komplexů cc32, cc35 a cc11. Oba izoláty séroskupiny B, cc35 mají alelové varianty 22-1 (VR1) a 14 (VR2), izoláty séroskupiny C, cc11, mají varianty 5 a 2. U izolátů séro-skupiny B, cc32 bylo významné zastoupení varianty 7 ve VR1 a varianty 16 ve VR2. Ve studovaném souboru byl pouze jeden izolát (4/15, B, cc162), který měl ve variabilní oblasti VR2 genu porA variantu 4.
Tab. 2. Tabulka 2. Molekulární charakterizace antigenů MenB vakcín u 20 izolátů IMO z roku 2015, ČR
Table 2. Molecular characteristics of MenB vaccine antigens in 20 isolates from IMD from 2015, Czech Republic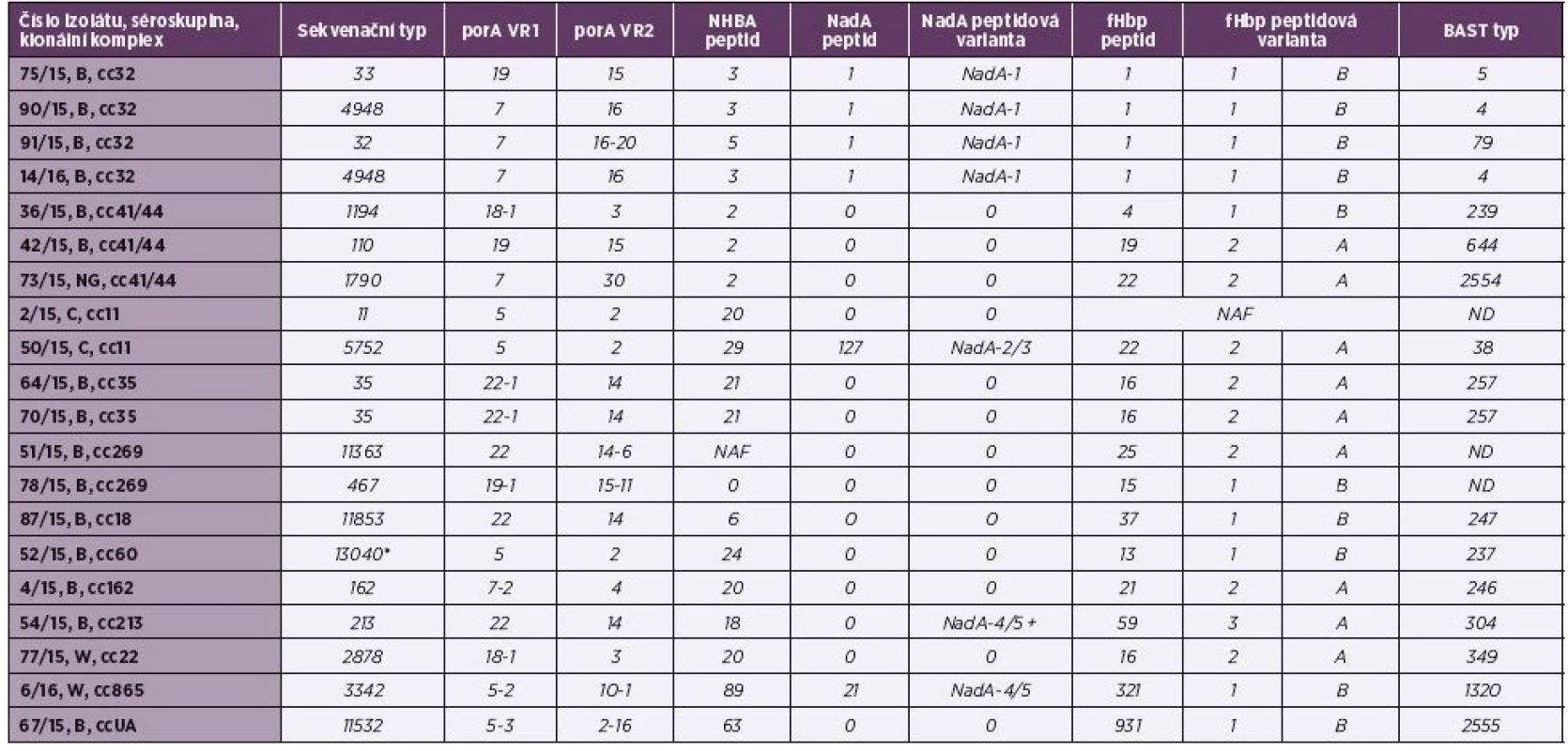
NG = non-groupable = izolát, u kterého nebylo možné určit séroskupinu
UA = unassigned = izolát, který není zařaditelný do klonálního komplexu
porA VR1, VR2 = variabilní regiony 1 a 2 genu porA
0 = izolát nenese žádnou variantu daného genu
* = nově popsaný sekvenační typ
NAF = nová alela genu, které ještě nebylo přiřazeno číselné označení
NadA-4/5 + = příslušná alela genu (nadA 40) je z důvodu posunu čtecího rámce inaktivní a neprodukuje peptid
BAST typ = Bexsero® Antigen Sequence Typing = sekvenační typ izolátu, určený kombinací variant MenB antigenů (porA VR1, VR2, a peptidové varianty genů fhbp, nhba a nadA)
ND = not determined = zjištěná kombinace variant MenB antigenů nebyla dosud definována jako určitý BAST typ3.2 Gen nhba
U genu nhba je podstatné určení jeho peptidové varianty. Vakcína MenB-fHbp produkt tohoto genu neobsahuje a ve vakcíně MenB-4C se vyskytuje peptidová varianta NHBA 2 [23]. U většiny izolátů testovaného souboru (19 z 20) metoda WGS určila alelovou i peptidovou variantu genu nhba (viz tab. 2). Rozložení variant bylo velice rozmanité, celkem bylo určeno 11 různých peptidových variant (2, 3, 5, 6, 18, 20, 21, 24, 29, 63, 89). Nejčastějšími variantami ve studovaném souboru izolátů byly NHBA 2 (3krát), NHBA 3 (3krát) a NHBA 20 (3krát) – graf 1. Asociace peptidové va-rianty genu nhba s klonálním komplexem byla pozorována především u izolátů cc41/44 (NHBA 2) a cc35 (NHBA 21). Tři izoláty ze čtyř, které patřily k cc32, obsahovaly peptidovou variantu NHBA 3. U izolátu 51/15, B, cc269 byla sekvence genu úspěšně detekována a od alely nhba 1, která kóduje peptidovou variantu NHBA 2, se lišila jednou bodovou mutací (A→G) a jednou delecí. Ta může způsobit, že protein v důsledku posunu čtecího rámce nevzniká. Tato skutečnost je v tabulce označena jako NAF (nová alelová forma). U izolátu 78/15, B, cc269 se metodou WGS zjistit sekvenci genu nhba nepodařilo. Ve studovaném souboru jsou tři izoláty (všechny cc41/44; 2krát séroskupina B, 1krát NG), které obsahují peptidovou variantu NHBA 2.
Graf 1. Výskyt a rozložení peptidových variant 3 antigenů vakcín MenB (fHbp, NadA, NHBA) u 20 izolátů IMO z roku 2015, ČR
Chart 1. Distribution of peptide variants of three MenB vaccine antigens (fHbp, NadA, and NHBA) in 20 isolates from IMD from 2015, Czech Republic
3.3 Gen nadA
U tohoto genu byla hodnocena jak varianta exprimovaného peptidu, tak nadřazená skupina “peptidové varianty“ (NadA-1, NadA-2/3, NadA-4/5, NadA-6) – viz tabulka 2. Vakcína MenB-4C obsahuje subvariantu NadA-3.8 [23], která indukuje tvorbu protilátek proti variantám NadA-1 a NadA-2/3 [2]. Vakcína MenB-fHbp neobsahuje žádný produkt genu nadA. Kompletní gen nadA byl zjištěn u 7 izolátů z 20, to představuje 35 %, což koresponduje s ostatními studiemi, které se týkají izolátů z IMO [2, 5]. Ve čtyřech případech se jednalo o varianty NadA-1, jednou NadA-2/3 a dvakrát byla zjištěna varianta NadA-4/5 (viz graf 1). U izolátu 54/15, B, cc213 byla zachycena alela nadA 40 (NadA-4/5), která obsahuje mutaci měnící čtecí rámec a v důsledku toho je tato alela neaktivní (není exprimován peptid). Pozorovatelná je silná korelace izolátů séroskupiny B, cc32, u kterých byla vždy přítomna peptidová varianta NadA-1.Ve studovaném souboru je pět izolátů, které by teoreticky měly být pokryty touto složkou vakcíny MenB-4C (4krát B, cc32 a 1krát C, cc11).
3.4 Gen fhbp
V současnosti existuje dvojí klasifikace genů antigenu fHbp: tři varianty – 1, 2, 3 a dvě podčeledi – A, B. Podčeleď A zahrnuje varianty 2 a 3, podčeleď B variantu 1 [19, 20, 23]. V důsledku této dvojí nomenklatury uvádíme peptidové varianty fHbp 1/B, 2/A a 3/A (viz tab. 2). Metodou WGS byla sekvence genu fhbp detekována u všech izolátů studovaného souboru v tomto zastoupení – 10krát fHbp 1/B, 8krát fHbp 2/A, 1krát fHbp 3/A a jeden izolát byl označen jako NAF (nová alelová forma). Izoláty souboru vykazovaly celou řadu různých typů fHbp peptidů: nejčastěji fHbp1 (4krát), dále fHbp16 (3krát) a fHbp22 (2krát). Další fHbp peptidy byly přítomny pouze u jednoho izolátu (4, 13, 15, 19, 21, 25, 37, 59, 321, 931) – viz graf 1. Korelace klonálního komplexu a peptidu byla pozorována u izolátů B, cc32, u kterých se vždy vyskytoval peptid fHbp1 (peptidová varianta 1/B). U obou izolátů B, cc35 se vyskytoval peptid 16 (varianta 2/A).
3.5 BAST
Kombinace peptidových a alelových variant antigenů vakcíny MenB-4C, jsou v BIGS databázi definovány jako charakteristika BAST typ (Bexsero® Antigen Sequence Typing) izolátu [4]. Ve studovaném souboru bylo zjištěno 15 již definovaných BAST typů: 4 (2krát), 5, 38, 79, 237, 239, 246, 247, 257 (2krát), 304, 349, 644 a 1320 (viz tab. 2). U dvou izolátů (73/15, NG, cc41/44; 67/15, B, ccUA) byly zjištěny nové dosud nedefinované kombinace antigenů a po předložení do BIGS databáze jim bylo přiděleno nové označení: 2554 a 2555. Jedná se tedy o nově popsané BAST typy. U tří izolátů se BAST typ určit nepodařilo: u dvou izolátů z důvodu výskytu nové alelové formy u některého z genů, u jednoho izolátu nebyl jeden z antigenů metodou WGS detekován.
Metoda WGS poskytuje vysoce přesné a spolehlivé výsledky. Z WGS dat všech 20 izolátů byly určeny alelové varianty všech MLST genů i všech variabilních regionů genů porA a fetA.V naprosté většině byla následně prokázána shoda s výsledky klasické sekvenace. V případě rozdílného výsledku byla pak odhalena chyba klasických sekvenačních metod. U izolátu 52/15, B byla metodou WGS detekována dosud nepopsaná alela genu aroE, které bylo databází BIGSdb přiděleno číslo 869. Tato alela vznikla pravděpodobně bodovou mutací z alely 19. Izolát 52/15, B byl následně přiřazen ke klonálnímu komplexu cc60 a označen jako nový sekvenační typ ST-13040.
Při analýze genetické diverzity vykazovaly izoláty, které patří ke stejnému klonálnímu komplexu, vzájemnou příbuznost na všech úrovních rozlišení. Porovnání genomů na úrovni sedmi MLST genů je dostačující pro rozlišení jednotlivých klonálních komplexů a potvrzuje vzájemnou příbuznost příslušných izolátů. S rostoucí rozlišovací schopností (schémata rMLST, cgMLST a “all loci“) roste kvalita výsledků. Jednotlivé klonální komplexy jsou jednak lépe vymezené a také se zvyšuje šance odlišit vysoce příbuzné izoláty (např. analýza diverzity uvnitř jednoho klonálního komplexu). Námi provedené analýzy ilustrují schopnost schématu cgMLST poskytnout kvalitnější a spolehlivější výsledky s mnohem vyšším rozlišením ve srovnání s klasickou MLST či rMLST. Toto zjištění je v souladu se zahraničními studiemi [7, 12, 13, 14, 15].
Metoda WGS dokázala určit alelové varianty genů, jejichž proteinové produkty představují komponenty nových vakcín MenB. Až na jednu výjimku (izolát 78/15, B, cc269; gen nhba) byla schopnost určení sekvencí těchto genů absolutní. Samostatnou kapitolu představuje gen nadA, který se přirozeně vyskytuje pouze cca u 30 % izolátů z IMO. I v tomto případě bylo na základě WGS dat zjištěno několik chyb klasických sekvenačních metod a objeveny nové alelové varianty. Podařilo se také zachytit izoláty s dosud nepopsanou kombinací antigenů MenB-4C vakcíny a byly určeny nové BAST typy. Metoda WGS, která je schopna identifikovat alely antigenů MenB vakcín, může do budoucna představovat alternativní metodu k zjišťování teoretického pokrytí izolátů N. meningitidis MenB vakcínami [19].
Diskuse o implementaci WGS a jejím případném nahrazení klasických sekvenačních metod při rutinním provádění surveillance IMO proběhly i na 14. kongresu EMGM 2017 v Praze [9]. Jednomyslnou shodou byla schválena podpora přístupu, který využívá oba typy metod. Metody klasické sekvenace jsou klíčové pro rychlé použití v surveillance IMO v reálném čase, zatímco metoda WGS poskytuje data pro detailní surveillance, zejména ve vztahu k vakcinačním strategiím.
ZÁVĚRY
Tato publikace předkládá výsledky prvního použití metody WGS v molekulární surveillance IMO v České republice. WGS metoda prokázala svoji absolutní diskriminační kapacitu a schopnost přesnějšího určení genetických vlastností N. meningitidis. Kvalitnější data umožnila identifikaci nových alel a nových sekvenačních typů, které nebyly klasickými sekvenačními metodami rozpoznány. WGS data poskytují také přesnější informace o teoretickém pokrytí izolátů novými MenB vakcínami. Zařazení metody WGS do rutinní molekulární surveillance IMO v České republice je proto žádoucí.
Poděkování
Podpořeno z programového projektu Ministerstva zdravotnictví ČR s reg. č. 15-34887A. Veškerá práva podle předpisů na ochranu duševního vlastnictví jsou vyhrazena. Definování alel a sekvenčních typů bylo provedeno srovnáním sekvenčních dat s MLST databází Neisseria Multi Locus Sequence Typing (https://pubmlst.org/neisseria/).
Do redakce došlo dne 17. 11. 2017.
Adresa pro korespondenci:
MUDr. Pavla Křížová, CSc.
Národní referenční laboratoř
pro meningokokové nákazy, CEM
Státní zdravotní ústav
Šrobárova 48
100 42 Praha10
e-mail: pavla.krizova@szu.cz
Zdroje
1. Bambini S, Piet J, Muzzi A, et al. An Analysis of the Sequence Variability of Meningococcal fHbp, NadA and NHBA over a 50-Year Period in the Netherlands. PLoS One, 2013;8(5):e65043. DOI: 10.1371/journal.pone.0065043.
2. Bambini S, Chiara MD, Muzzi A, et al. Neisseria Adhesin A Variation and Revised Nomenclature Scheme. Clin Vaccine Immunol, 2014;21(7):966–971. DOI: 10.1128/CVI.00825-13.
3. Bratcher HB, Corton C, Jolley KA, et al. A gene-by-gene population genomics platform: de novo assembly, annotation and genealogical analysis of 108 representative Neisseria meningitidis genomes. BCM Genomics, 2014;15(1138). DOI: 10.1186/1471-2164-15-1138.
4. Brehony C, Hill DMC, Lucidarme J, et al. Meningococcal vaccine antigen diversity in global databases. Euro Surveill, 2016;20(49). DOI: 10.2807/1560-7917.ES.2015.20.49.30084.
5. Brehony C, Rodrigues ChMC, Borrow R, et al. Distribution of Bexsero® Antigen Sequence Types (BASTs) in invasive meningococcal disease isolates: Implications for immunization. Vaccine, 2016;34(39):4690–4697. DOI: 10.1016/j.vaccine.2016.08.015.
6. Harrison OB, Brueggemann AB, Caugant DA, et al. Molecular typing methods of outbreak detection and surveillance of invasive disease caused by Neisseria meningitidis, Haemophilus influenzae and Streptococcus pneumoniae, a review. Microbiology, 2011;157(8):2181–2195. DOI: 10.1099/mic.0.050518-0.
7. Hill DMC, Lucidarme J, Gray SJ, et al. Genomic epidemiology of age-associated meningococcal lineages in national surveillance: an observation cohort study. Lancet Infect Dis, 2015;15(12):1420–1428. DOI: 10.1016/S1473-3099(15)00267-4.
8. http://emgm.eu/emert
9. http://emgm.eu/events.shtml
10. https://pubmlst.org/neisseria/
11. https://tessy.ecdc.europa.eu/tessyweb
12. Jolley KA, Maiden MCJ. BIGSdb: Scalable analysis of bacterial genome variation at the population level. BMC Bioinformatics, 2010;11(595). DOI: 10.1186/1471-2105-11-595.
13. Jolley KA, Bliss CM, Bennett JS, et al. Ribosomal multilocus sequence typing: universal characterization of bacteria from domain to strain. Microbiology, 2012;158(4):1005–1015. DOI: 10.1099/mic.0.055459-0.
14. Jolley KA, Hill DMC, Bratcher HB, et al. Resolution of a Meningococcal Disease Outbreak from Whole-Genome Sequence Data with Rapid Web-Based Analysis Methods. J Clin Microbiol, 2012;50(9):3046–3053. DOI: 10.1128/JCM.01312-12.
15. Lucidarme J, Hill DMC, Bratcher HB, et al. Genomic resolution of an aggressive, widespread, diverse and expanding meningococcal serogroup B, C and W lineage. J Infect, 2015;71(5):544–552. DOI: 10.1016/j.jinf.2015.07.007.
16. Maiden MCJ, Bygraves JA, Feil E, et al. Multilocus sequence typing: A portable approach to the identification of clones within populations of pathogenic microorganisms. Proc Natl Acad Sci U S A, 1998;95(6):3140–3145.
17. Masignani V, Comanducci M, Giuliani MM, et al. Vaccination against Neisseria meningitidis Using Three Variants of the Lipoprotien GNA1870. J Exp Med, 2003;197(6):789–799. DOI: 10.1084/jem.20021911.
18. McNeil LK, Zagursky RJ, Lin SL, et al. Role of Factor H Binding Protein in Neisseria meningitidis Virulence and Its Potential as a Vaccine Candidate To Broadly Protect against Meningococcal Disease. Microbiol Mol Biol Rev, 2013;77(2):234–252. DOI: 10.1128/MMBR.00056-12.
19. Mowlaboccus S, Perkins TT, Smith H, et al. Temporal Changes in BEXSERO® Antigen Sequence Type Associated with Genetic Lineages of Neisseria meningitidis over a 15-Year Period in Western Australia. PLoS One, 2016;11(6):e0158315. DOI:10.1371/journal.pone.0158345.
20. Nissen MD, Marshall HS, Richmond PC, et al. A Randomized, Controlled, Phase ½ Trial of a Neisseria meningitidis Serogroup B Bivalent rLP2086 Vaccine in Healthy Children and Adolescents. Pediatr Infect Dis J, 2013;32(4):364–371. DOI:10.1097/INF.0b013e31827b0d24.
21. Revez J, Espinosa L, Albiger B, et al. Survey on the Use of Whole-Genome Sequencing for Infectious Diseases Surveillance: Rapid Expansion of European National Capacities, 2015-2016. Front Public Health, 2017;5 : 347. DOI: 10.3389/fpubh.2017.00347.
22. Vacková Z, Křížová P, Jandová Z, et al. Možnosti implementace sekvenace celého genomu (WGS) Neisseria meningitidis do molekulární surveillance invazivního meningokokového onemocnění v České republice. Epidemiol Mikrobiol Imunol, 2017;66(1):50–52.
23. Vernikos G, Medini D. Bexsero® chronicle. Pathog Glob Health, 2014;108(7):305–316. DOI: 10.1179/2047773214Y.0000000162.
24. Whittaker R, Dias JG, Ramliden M, et al. The epidemiology of invasive meningococcal disease in EU/EEA countries, 2004-2014. Vaccine, 2017;35(16):2034–2041. DOI: 10.1016/j.vaccine.2017.03.007.
25. Zerbino DR. Using the Velvet de novo assembler for short-read sequencing technologies. Curr Protoc Bioinformatics, 2010;11(11.5). DOI: 10.1002/0471250953.bi1105s31.
Štítky
Hygiena a epidemiologie Infekční lékařství Mikrobiologie
Článek vyšel v časopiseEpidemiologie, mikrobiologie, imunologie
Nejčtenější tento týden
2018 Číslo 2- Stillova choroba: vzácné a závažné systémové onemocnění
- Diagnostika virových hepatitid v kostce – zorientujte se (nejen) v sérologii
- Prokalcitonin: marker vhodný pro diagnostiku sepse i hodnocení antimikrobiální léčby
- Autoinflamatorní onemocnění: prognózu zlepšuje včasná diagnostika a protizánětlivá terapie
- Infekční komplikace virových respiračních infekcí – sekundární bakteriální a aspergilové pneumonie
-
Všechny články tohoto čísla
- Stav imunitního systému u pacientů se subklinickou formou urogenitální tuberkulózy
- Molekulární epidemiologie tuberkulózy v hlavním městě Praha v letech 2013 a 2014
- Surveillance invazivního meningokokového onemocnění založená na sekvenaci celého genomu (WGS), Česká republika, 2015
- Boj s rezistencí mikroorganismů na antibiotika
- Streptokokový syndrom toxického šoku – život ohrožující stav vyvolaný různými druhy streptokoků
- Bakteriémie vyvolaná Staphylococcus aureus – význam správného přístupu k diagnostice a léčbě
- Aktualizace českých doporučených postupů pro laboratorní diagnostiku infekcí vyvolaných Clostridium difficile
- Epidemiologie, mikrobiologie, imunologie
- Archiv čísel
- Aktuální číslo
- Informace o časopisu
Nejčtenější v tomto čísle- Streptokokový syndrom toxického šoku – život ohrožující stav vyvolaný různými druhy streptokoků
- Bakteriémie vyvolaná Staphylococcus aureus – význam správného přístupu k diagnostice a léčbě
- Boj s rezistencí mikroorganismů na antibiotika
- Aktualizace českých doporučených postupů pro laboratorní diagnostiku infekcí vyvolaných Clostridium difficile
Kurzy
Zvyšte si kvalifikaci online z pohodlí domova
Revma Focus: Spondyloartritidy
nový kurz
Autoři: prof. MUDr. Vladimír Palička, CSc., Dr.h.c., doc. MUDr. Václav Vyskočil, Ph.D., MUDr. Petr Kasalický, CSc., MUDr. Jan Rosa, Ing. Pavel Havlík, Ing. Jan Adam, Hana Hejnová, DiS., Jana Křenková
Autoři: MUDr. Irena Krčmová, CSc.
Všechny kurzyPřihlášení#ADS_BOTTOM_SCRIPTS#Zapomenuté hesloZadejte e-mailovou adresu, se kterou jste vytvářel(a) účet, budou Vám na ni zaslány informace k nastavení nového hesla.
- Vzdělávání



