-
Články
Top novinky
Reklama- Vzdělávání
- Časopisy
Top články
Nové číslo
- Témata
Top novinky
Reklama- Kongresy
- Videa
- Podcasty
Nové podcasty
Reklama- Kariéra
Doporučené pozice
Reklama- Praxe
Top novinky
ReklamaSekvenování – klasická metodika
Sequencing – classical method
In this article the basic methods of reading nucleotide sequences in DNA molecules are summarized. Sanger sequencing is described most thoroughly as it is the most frequent routine method currently being utilized. The article describes in detail the principle of sequence determination through the production of fragments with a known end base using chain termination synthesis of DNA and ways of separation and detection of the fragments. Some alternative methods of sequencing are mentioned in short. Basic approaches of analyzing sequence data are explained as well as different outcomes, obstacles and challenges.
Key words:
sequencing – Sanger – synthesis – ddNTP – fluorescence – capillary electrophoresis – mutation – electropherogram
Autoři: Monika Šedivcová 1; Petr Martínek 1; Jan Stehlík 1; Petr Grossmann 1; Jana Kašpírková 1,2; Tomáš Vaneček 1,2
Působiště autorů: Bioptická laboratoř, s. r. o., Plzeň 1; Šiklův ústav patologie LF UK a FN, Plzeň 2
Vyšlo v časopise: Čes.-slov. Patol., 49, 2013, No. 3, p. 122-128
Kategorie: Přehledový článek
Souhrn
V krátkém přehledu je pojednáno o základních metodách čtení pořadí jednotlivých bází v molekule DNA. Podrobněji je popsáno tzv. Sangerovo sekvenování, které je v současnosti zdaleka nejrozšířenější klasickou metodou. Detailně je osvětlen princip určení pořadí bází pomocí tvorby fragmentů se známou koncovou bází prostřednictvím terminační syntézy DNA a možnosti separace a detekce získaných produktů. Krátce jsou též zmíněny některé alternativní metodiky. Dále je vysvětlen postup při analýze získaných sekvenačních dat, možné výstupy z těchto dat a také omezení a možná úskalí Sangerovy sekvenační analýzy.
Klíčová slova:
sekvenování – Sanger – syntéza – ddNTP – fluorescence – kapilární elektroforéza – mutace – sekvenogram
Sekvenování je metoda molekulární biologie, která slouží k určení přesného pořadí bází, tedy adeninu (A), cytosinu (C), guaninu (G) a thyminu (T) v řetězci DNA. Využívá se jak k určení pořadí bází v dosud neznámých genech, chromozomech či genomech (v roce 2001 bylo např. úspěšně dokončeno sekvenování celého lidského genomu (1)), tak k detekci odchylek, tedy mutací či polymorfizmů, od již známé sekvence. Toto takzvané resekvenování slouží k odhalení substitucí (báze nahrazena jinou), delecí (ztráta bází), inzercí (vložení bází) nebo duplikací (zdvojení bází) v genech, které hrají důležitou úlohu v řadě dědičných i sporadických onemocnění člověka. Sekvenování tak představuje užitečný nástroj patologické diagnostiky.
Počátky sekvenování, ve smyslu jak o něm hovoříme nyní, se datují do poloviny 70. let, kdy byly vyvinuty a publikovány dvě různé sekvenační metody – Sangerova (označována také jako „dideoxymetoda“) a Maxam-Gilbertova (2,3), obě využívající elektroforetické separace různě dlouhých fragmentů DNA se známou koncovou bází. Zejména z technických důvodů však časem zcela převážila metoda Sangerova, která je nyní využívána ve všech oblastech molekulární biologie.
PRINCIP SANGEROVY METODY
Principem Sangerovy metody je terminace syntézy nových DNA řetězců prostřednictvím náhodné inkorporace modifikovaných nukleotidů, tzv. dideoxynukleotidů (ddNTPs), separace těchto řetězců a jejich vizualizace.
Nejprve je zkoumaný úsek DNA namnožen na vysoký počet kopií (řádově miliardy z jedné molekuly). Tato amplifikace probíhá in vitro za použití polymerázové řetězové reakce (PCR), popř. příbuzných metod, či in vivo pomocí klonování a replikace v laboratorním mikroorganizmu.
Vlastní sekvenační reakce obsahuje několik komponent. První z nich je namnožená templátová DNA. Původně byla používána výhradně jednořetězcová DNA, s postupem času však byly vypracovány protokoly umožňující analyzovat jednořetězcovou DNA připravenou z dvouřetězcové přímo v sekvenační reakci. Další složkou je replikační enzym DNA polymeráza, který syntetizuje komplementární vlákno k sekvenované DNA. Syntéza probíhá obdobně jako při replikaci ve směru 5’→3’. K syntéze využívá DNA polymeráza tzv. primer, což je zhruba 20 bází dlouhý syntetický oligonukleotid komplementární ke konkrétní části templátu, a také jednotlivé deoxynukleotidy (dNTPs), které začleňuje na základě principu komplementarity bází do nově vznikajícího řetězce. V původním Sangerově provedení s radioaktivně značeným dATP (značení prostřednictvím 32P) je výše zmíněná reakční směs rozdělena do čtyř zkumavek označených G, A, T, C a do každé z nich je přidán i příslušný ddNTP a to v koncentraci řádově nižší (specificky dle konkrétní použité DNA polymerázy), než je koncentrace ostatních dNTP. Do zkumavky označené G je tedy přidán ddGTP, do zkumavky A je přidán ddATP atd. Tyto ddNTP se během replikace náhodně začlení do nově syntetizované DNA, což zastaví prodlužování řetězce v tomto místě. Důvodem je nepřítomnost -OH skupiny na 3’ uhlíku ddNTP, která znemožní vytvoření fosfodiesterové vazby s dalším dNTP (obr. 1). Vznikne tak směs produktů různé délky, která vždy končí známým ddNTP. Tyto 4 směsi produktů jsou naneseny do 4 sousedních drah (označených G, A, T a C) v denaturační polyakrylamidové gelové elektroforéze (PAGE). Denaturační PAGE rozdělí působením stejnosměrného elektrického pole směs vzniklých fragmentů od nejmenšího k největšímu s přesností na jednu bázi. Takto rozdělené fragmenty se pak vizualizují autoradiografií a z jednotlivých pruhů na fotografii (tzv. sekvenogram) se odečte výsledná sekvence (obr. 2) (2).
Obr. 1. Rozdíl mezi 2-deoxynukleosidem a 2,3-dideoxynukleosidem. Vlevo: 2-deoxy (je možné začlenění dalšího nukleotidu na 3´-OH skupinu). Vpravo: 2,3-dideoxy (začlenění dalšího nukleotidu není možné z důvodu chybějící 3´-OH skupiny). 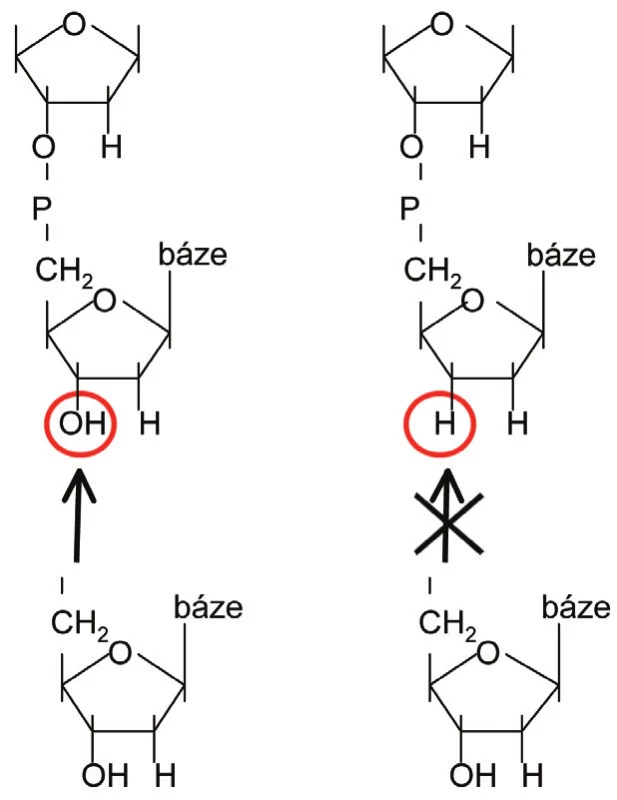
Obr. 2. Schéma principu sekvenování Sangerovou metodou. Syntéza nového řetězce DNA probíhá ve směru 5´→3` za přítomnosti specifického primeru, jednotlivých deoxynukleotidů (dNTP), dideoxynukleotidů (ddNTP) a DNA polymerázy. V levé části je znázorněna Sangerova metoda s radioaktivním značením templátu nebo fluorescenčně značenými primery, kdy reakce probíhá ve čtyřech zkumavkách a sekvence je získaná po denaturační polyakrylamidové gelové elektroforéze (PAGE). V pravé části je znázorněna tato metoda s fluorescenčním značením ddNTP (L = label-značka), kdy reakce probíhá v jedné zkumavce a sekvence je získána po kapilární elektroforéze (15). 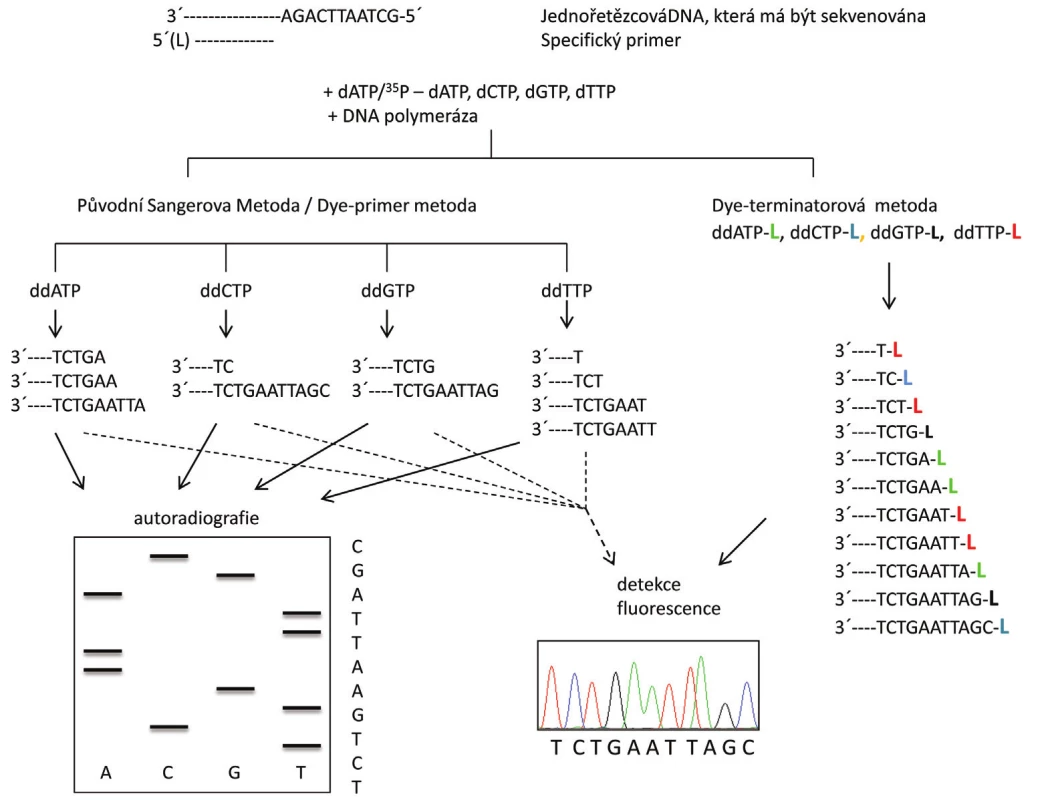
Sangerova metoda zaznamenala od dob svého vzniku četná vylepšení. Nejprve bylo nahrazeno radioaktivní značení templátu značením fluorescenčním primerem a v současné době převažuje použití fluorescenčních ddNTP, jež se inkorporují do vznikajícího řetězce při lineární amplifikaci, tj. cyklické terminační syntéze z jednoho primeru. Každý ze čtyř ddNTP nese jinou barvu, jejíž emisní spektrum je dobře odlišitelné od ostatních (v nejčastěji používané tzv. „BigDye“ chemii je to karboxyfluorescein (FAM), karboxy-4’, 5’-dichloro-2’, 7’-dimethoxyfluorescein (JOE), karboxytetramethyl-rhodamin (TAMRA) a karboxy-X-rhodamine (ROX)), což umožňuje provádět sekvenační reakci v jedné zkumavce. Spolu s tím byla PAGE nahrazena elektroforézou kapilární, která využívá vlastnosti pohybu vláken DNA v tekuté nebo gelové matrici (polymeru). Fluorescenčně značené produkty sekvenační reakce se separují v kapiláře podle velikosti a prostřednictvím laserové excitace a detekce emise fluorescenčního značení jsou postupně snímány detektorem umístěným na konci kapiláry. Získané signály jsou převáděny softwarem do podoby sekvenogramu jako po sobě jdoucí barevné píky odpovídající sekvenci nukleotidů analyzované DNA. Čtyři různé použité fluorescenční barvy označují jednotlivé nukleotidy, přičemž barevná symbolika je nejčastěji následující: adenin zeleně, cytosin modře, guanin žlutě nebo černě a thymin červeně (obr. 2) (4–6).
PRINCIP MAXAM-GILBERTOVY METODY
Obdobně jako u Sangerovy metody se zkoumaný úsek nejdříve namnoží in vitro či in vivo. Následně se radioaktivně označí a na tyto označené řetězce DNA se působí chemickými látkami, které specificky štěpí sekvenovanou DNA v místě příslušné báze (např. směs dimetylsulfátu a piperidinu štěpí DNA pouze v místě, kde je v sekvenci přítomen guanin). Tyto fragmenty se opět elektroforeticky separují, vizualizují a vyhodnotí (3). Jak již bylo ale řečeno, tato metoda se nerozšířila, a to i přes počáteční větší oblíbenost související především s faktem, že bylo možno analyzovat dvouřetězcovou DNA.
DALŠÍ SEKVENAČNÍ METODY
K určení pořadí bází v DNA se používají i další metodiky a detekční systémy. Jedná se např. o pyrosekvenování využívající detekci světla produkované enzymem luciferázou po inkorporaci nukleotidu a uvolnění pyrofosfátu konvertovaného dále na ATP jako zdroje energie pro enzymatickou reakci (např. 454 sekvenátory firmy Roche); metodu iontového polovodičového sekvenování založenou na principu změny pH roztoku po inkorporaci nukleotidu (např. Ion Torrent, Applied Biosystems); sekvenování systémem firmy Illumina (např. sekvenátor HiSeq), které je založeno na principu postupné inkorporace laserem modifikovatelných terminátorů a detekce fluorescence; sekvenování ligací (např. systém SOLiD, Applied Biosystems) na principu ligace oligonukleotidů s adaptorem s využitím sekvenačního procesu katalyzovaného DNA ligázou atd. Tyto techniky jsou však téměř výhradně součástí tzv. “next generation“ sekvenování (7) (viz další článek v této sérii).
AUTOMATIZACE SEKVENOVÁNÍ
Moderní diagnostické laboratoře používají téměř výhradně automatizovanou sekvenaci s kapilárně elektroforetickým systémem separace sekvenovaných fragmentů, jejíž koncept navrhnuli v roce 1992 Huang a kol. (8). Obecně tyto přístroje používají systém jedné či více kapilár, do kterých je vnesen přečištěný produkt sekvenační reakce. Počet kapilár určuje, kolik vzorků je paralelně separováno/čteno. Kompletní sekvenační analyzátor má vlastní detekční a vyhodnocovací systém, jež umožní přečtení sekvence dlouhé až 1000 bází na jednu reakci, v závislosti na použité délce kapiláry a typu separační matrice (polymeru) (obr. 3).
Obr. 3. Automatický sekvenátor ABI3130xl (Applied Biosystems) – popis základních funkčních částí přístroje. 
POSTUP PŘI ZÍSKÁVÁNÍ SEKVENCÍ
Jak již bylo řečeno, vstupem do sekvenační reakce je purifikovaný namnožený úsek DNA. Pokud se jedná o plazmid nesoucí inzert analyzované DNA, je tento izolován z bakteriální kultury zpravidla již v dostatečné čistotě. Pokud se jedná o unikátní PCR produkt, je nutné z něj odstranit především soli, nezainkorporované nukleotidy a primery, které mohou negativně ovlivnit kvalitu získaných dat (viz dále). V současnosti se tato purifikace provádí nejčastěji pomocí silikátové matrice (kolonky), popř. prostřednictvím paramagnetických partikulí. V případě, že při PCR získáme více produktů, je nutná prvotní separace těchto produktů v gelu, rozpuštění a přečištění DNA, opět nejčastěji na silikátových matricích (kolonkách). Další alternativou k separaci více produktů vzniklých při PCR může být též klonování produktů do plazmidu s následnou sekvenační analýzou široké palety klonů.
Přečištěné PCR produkty jsou sekvenovány, nejlépe z obou stran pro kontrolu dané sekvence a správnosti čtení. Sekvenace probíhá nejčastěji prostřednictvím primerů použitých v PCR. Po proběhnutí sekvenační reakce je tato znovu přečištěna. Tento krok opět zajistí odsolení a také odstranění nezainkorporovaných značených dideoxynukleotidů, které komplikují či znemožňují čtení výsledné sekvence (viz dále). Produkty sekvenační reakce lze opět čistit mnoha způsoby, s úspěchem lze, obdobně jako u PCR, použít paramagnetické partikule nebo sephadexové matrice. Takto připravené produkty jsou rozpuštěny ve vodě či formamidu a elektroforeticky vtaženy do kapiláry, kde se provede separace fragmentů a jejich postupná vizualizace. Příslušný software data vyhodnotí, provede označení bází (tzv. „basecalling“), u každé báze vyhodnotí její kvalitu prostřednictvím tzv. Q-hodnoty (Q-value), což je pravděpodobnost, se kterou je báze správně označena, a vytvoří grafický výstup získaných dat – sekvenogram (obr. 4) (9).
Obr. 4. Výstup ze sekvenátoru s analýzou části exonu 15 genu APC (sekvence v rozsahu bází 46-91) s graficky vyznačenými Q-hodnotami (sloupec nad jednotlivými bázemi). Většina Q hodnot se pohybuje mezi 20 a 50 (pravděpodobnost správného vyhodnocení báze softwarem 99 % až 99,999 %). Zakroužkovány jsou problematicky určené báze. Báze nazvaná M na pozici 49 s Q hodnotou 1, je chybně určená jako C/A pík v jedné pozici. Jedná se o klasickou „basecall“ chybu vyhodnocovacího software. Správně je CA ve dvou za sebou jdoucích pozicích. Báze zvaná Y na pozici 52 má Q hodnotu 9 a jde o správně určenou mutaci genu APC. Mutace je v heterozygotním stavu, na jednom chromozomu „wild-type“ C, na druhém mutace vedoucí k záměně tohoto C za T. Báze C na pozici 74 opět s nízkou Q hodnotou je určená správně. Software pouze vzal v úvahu lehký překryv C a A píku. 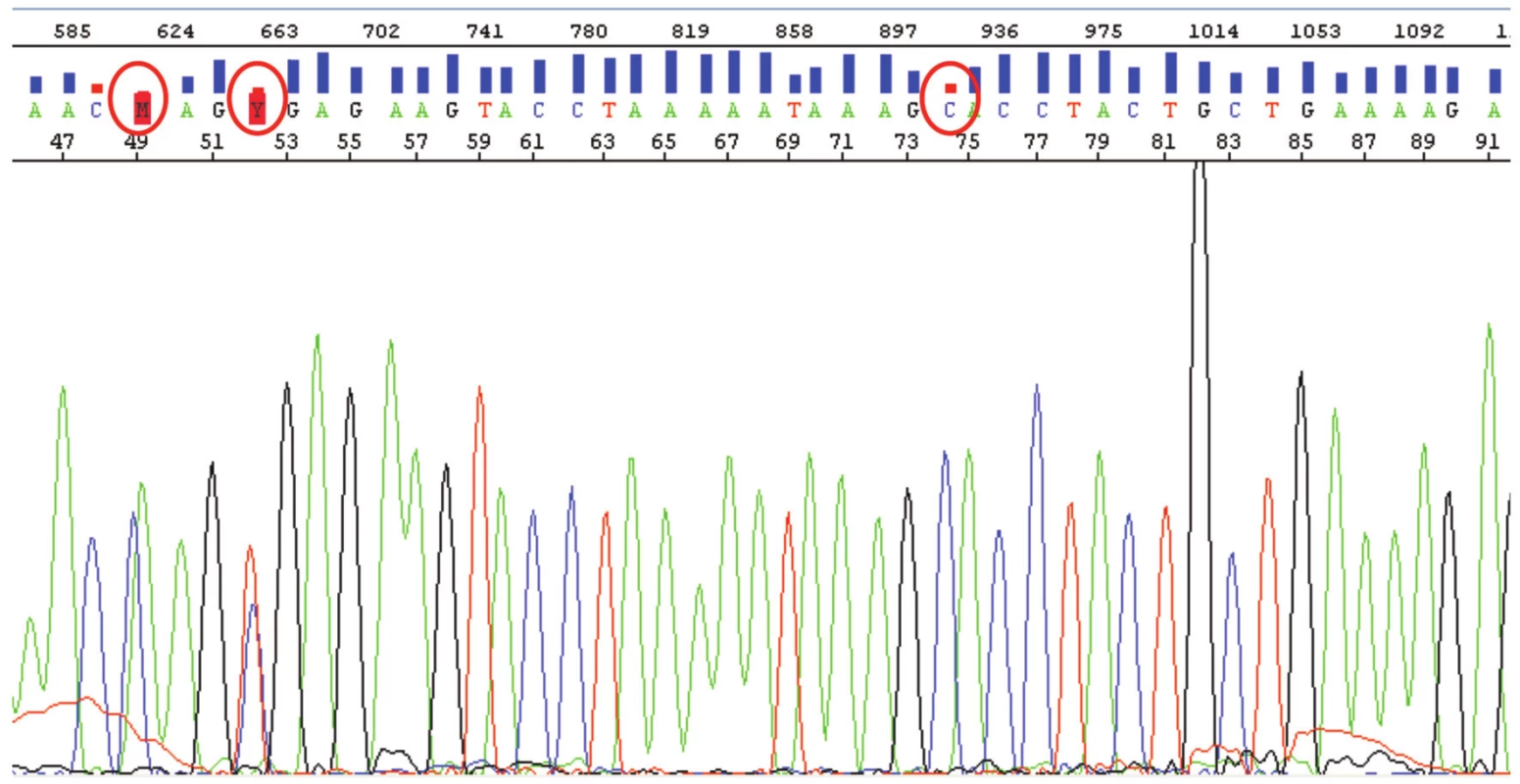
POSTUP PŘI ANALÝZE SEKVENCÍ
U získaného sekvenogramu je nejprve nutné zjistit, zda jeho kvalita umožňuje další analýzy. Toto vyhodnocení provádí školený pracovník, který vizuálně zkontroluje získaná data a Q-hodnotu a tam, kde je to možné, provede patřičné úpravy. Mezi ně patří například odfiltrování bází s nízkým rozlišením na počátku a sekvence primeru na konci sekvenogramu, oprava očividně chybně označených bází apod. Pokud školený pracovník zjistí, že kvalita sekvenogramu neumožňuje správné přečtení sekvence, a to již z jakéhokoliv důvodu (viz dále), sekvenování je nutné opakovat.
Čitelná sekvence je analyzována in silico porovnáním s referenční sekvencí v on-line databázi (např. genová banka, nástroj BLAST - http://blast.ncbi.nlm.nih.gov/Blast.cgi) či pomocí k tomu určených, většinou komerčně dostupných, softwarů (např. Lasergene, SeqScape, Sequencher). Porovnáním analyzované sekvence se sekvencí referenční je získána informace o případné přítomnosti změny (mutace) a to jak v heterozygotním, tak i homozygotním stavu.
TYPY ZJIŠTĚNÝCH ZMĚN (MUTACÍ)
Jedním z nálezů při vyhodnocování sekvenogramů jsou tzv. substituční mutace. Jedná se o záměny jednotlivých nukleotidů za jiné. Ve vlastním sekvenogramu jsou mutace v heterozygotním stavu detekovány jako dva překrývající se píky v jedné pozici, v in silico porovnání v genové bance jako místo bez homologie (obr. 5). Homozygotní mutace se zobrazí jako pík odlišný od referenčního sekvenogramu a v porovnání v genové bance, obdobně jako u heterozygotní mutace, jako místo bez homologie.
Obr. 5. Sekvenogram genu APC (vlevo referenční „wild type“ sekvence, vpravo analyzovaná sekvence). Jedná se o záměnu báze C za T (označení Y pro takovéto báze na jedné pozici), mutace je v heterozygotním stavu; je spojena s onemocněním familiární adenomatózní polypóza střev (FAP). B: Výstup z databáze BLAST – Porovnání analyzované sekvence genu APC (horní řádky „Query“) s referenční sekvencí (spodní řádky „Sbjct“). Označeno je místo bez homologie. V případě homozygotní či hemizygotní mutace by označená báze C byla v sekvenogramu zcela nahrazena bází T. 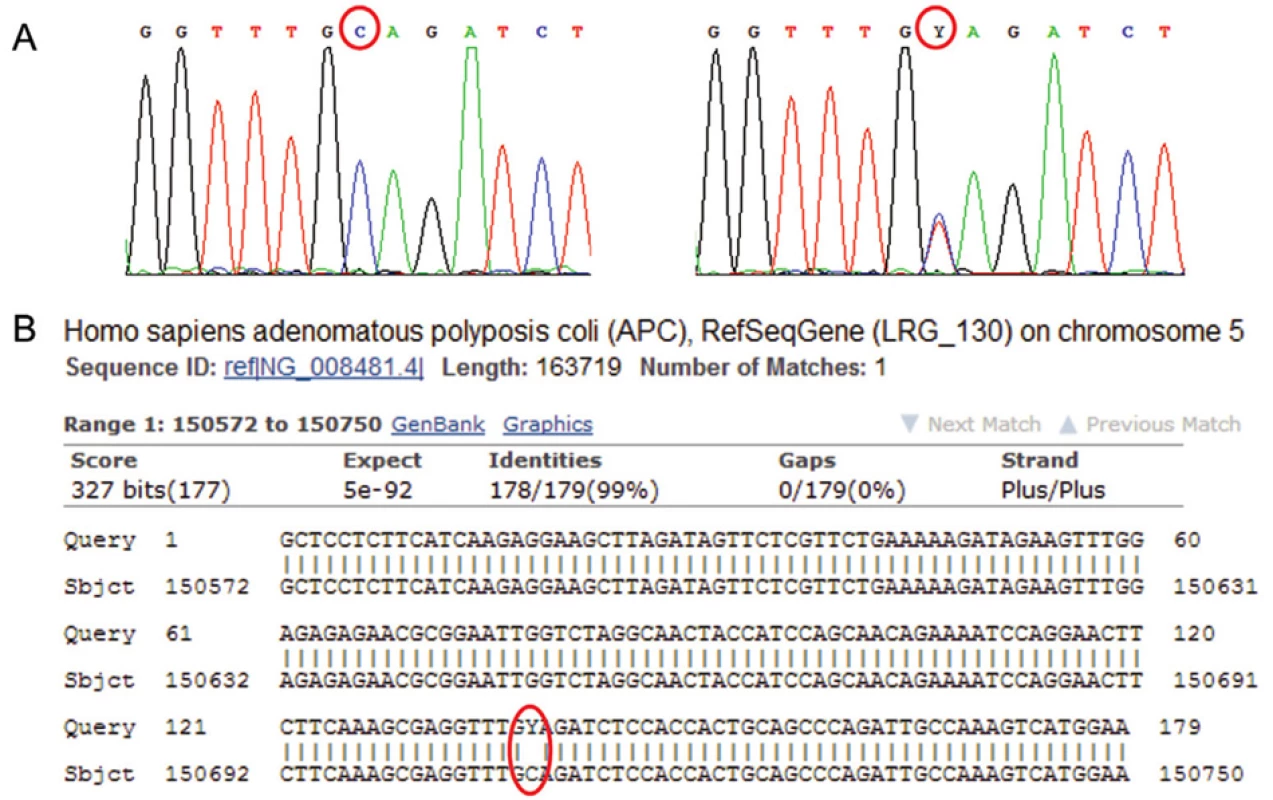
Dalším typem mutací jsou delece, inzerce nebo duplikace, které zasahují jeden nebo více nukleotidů. V sekvenogramu jsou u heterozygotů tyto změny detekovány jako dvě překrývající se sekvence, které začínají v místě mutace a pokračují dále ve směru čtení. Porovnáním s referenčním sekvenogramem, popř. pomocí specializovaného software, lze charakterizovat přesnou formu a rozsah delece, inzerce či duplikace. Tyto mutace lze také analyzovat prostřednictvím porovnání v genové bance, přičemž na výstupu nalézáme úsek bez homologie. U homozygotů není změna nukleotidové sekvence na první pohled patrná a je zjistitelná porovnáním s referenčním sekvenogramem. Komparace v genové bance pak vykazuje, opět obdobně jako u heterozygotní mutace, sled míst bez homologie (obr. 6).
Obr. 6. A: Sekvenogram genu pro GJB2 (Connexin 26). Jedná se o deleci báze G (vlevo referenční „wild type“ sekvence, uprostřed heterozygotní stav, vpravo homozygotní stav). Mutace je asociována s tzv. Nesyndromovou hluchotou (DFNB1). (Za poskytnutí sekvenogramů děkujeme RNDr. Martině Putzové, Ph.D.) B: Výstup z databáze BLAST – porovnání analyzované sekvence genu pro GJB2 (horní řádky „Query“) s referenční sekvencí (spodní řádky „Sbjct“). Označeno je místo bez homologie. 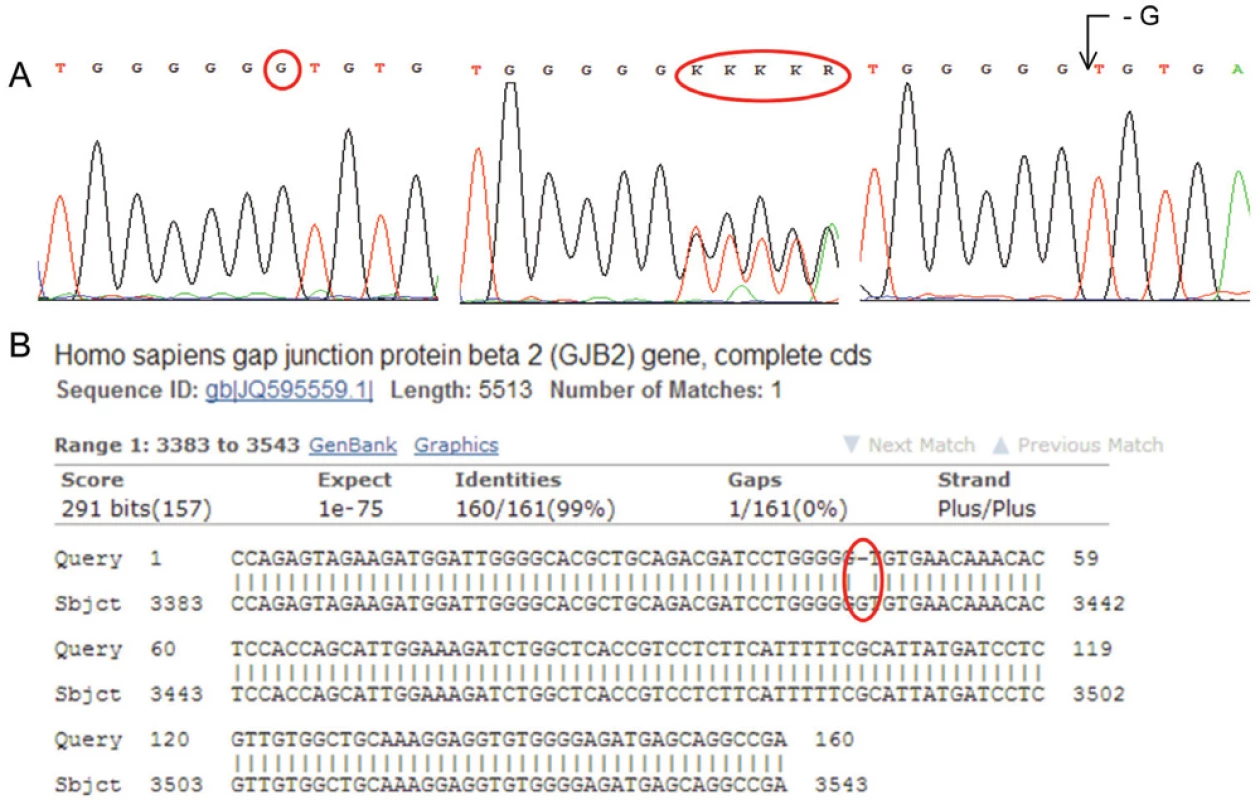
Výše zmíněné případy jsou idealizovaným modelem, který platí např. u analýzy germinálních mutací, kdy se u heterozygota nachází v sekvenogramu dva píky (jednobázová substituce) či sled dvojitých píků (delece/inzerce/duplikace) v poměru ideálně 1 : 1 a u mutantního homozygota pouze mutované píky. U analýzy somatických mutací může zejména v důsledku kontaminace nenádorovou tkání či nehomogenity přítomnosti mutovaných buněk dojít ke snížení či úplné absenci mutantního píku/ů zaznamenaného/ých v sekvenogramu (viz také analytická citlivost metody) (obr. 7). Z těchto důvodů je též obtížně rozlišitelná homo/hemizygotní mutace od heterozygotní.
Obr. 7. Sekvenogram s analýzou genu K-RAS (nahoře: referenční „wild type“ sekvence, dole: analyzovaná sekvence). Jde o somatickou mutaci se záměnou báze G na A. Mutantní pík (A) je snížený pravděpodobně v důsledku přiítomnosti nenádorové komponenty v izolovaném materiálu. Mutace byla potvrzena druhou nezávislou metodou. 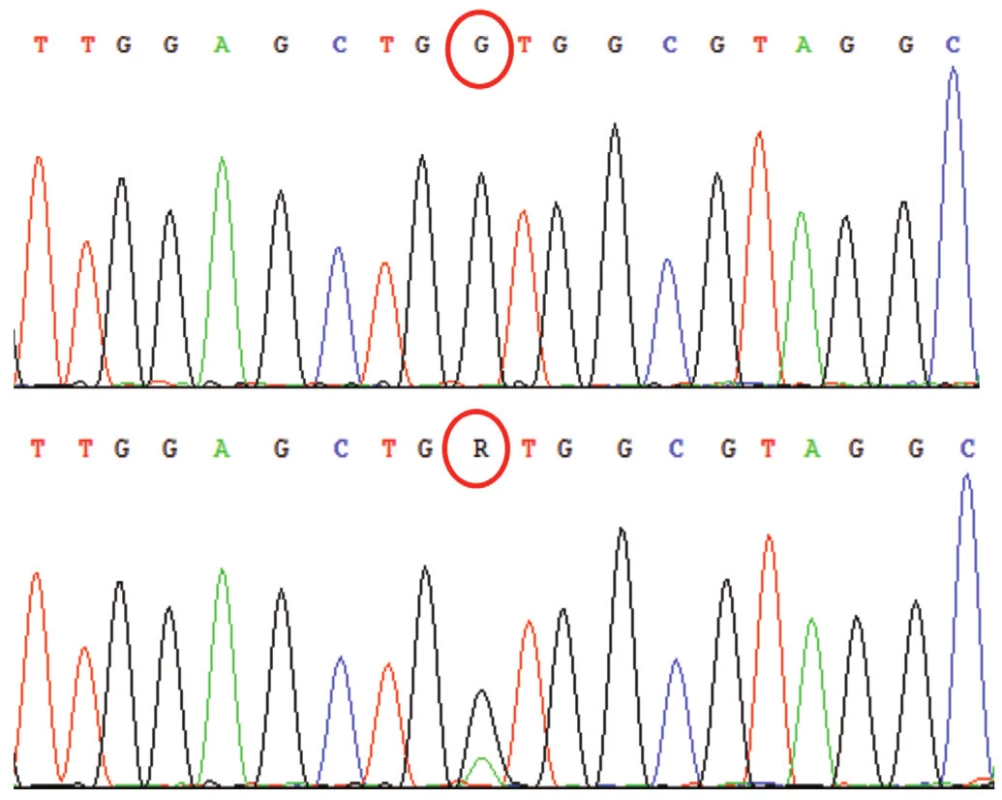
OMEZENÍ SANGEROVA SEKVENOVÁNÍ
Metoda Sangerova sekvenování má některá principiální a technická omezení. Zde uvedeme některé z nich.
Především, sekvenování není z principu kvantifikační metodou. Nelze tedy při analýze sekvenogramu rozlišit, zda daná sekvence pochází ze dvou homologních lokusů, nebo pouze z jednoho, přičemž druhý zcela chybí, či přebývá. Tento fakt má význam v případě přítomnosti změn velkého rozsahu zahrnující velké delece i duplikace, které nelze touto metodu detekovat.
Důležitou charakteristikou, zejména pro molekulární patology, je též analytická citlivost metody. Ta se u Sangerova sekvenování pohybuje mezi 10 – 20 % (10). V praxi to znamená, že touto metodou lze spolehlivě zachytit změnu v sekvenci DNA pouze v případě, kdy je tato změna přítomná alespoň u 10 – 20 % kopií DNA ve vzorku. V opačném případě nemusí být alterace detekována a vzorek je interpretován jako falešně negativní.
Existuje i celá řada dalších technických omezení. Například prvních 20 – 30 bází za sekvenačním primerem bývá zpravidla nečitelných, což je způsobeno ztrátou těchto velmi krátkých produktů sekvenační reakce během jejího přečištění. Z tohoto důvodu je třeba navrhnout sekvenační primer minimálně 40 – 50 bp před místo, které je třeba sekvenovat. Podobně nelze použít sekvenci posledních několika bází sekvenované DNA, protože se vždy ve skutečnosti jedná o sekvenci použitého PCR primeru a ne o sekvenci templátové DNA. Dalším omezením je maximální délka čitelné sekvence, která se v závislosti na délce kapiláry a hustotě polymeru pohybuje přibližně do 1000 bází. Na konci sekvence pak dochází k postupnému zhoršení rozlišení jednotlivých píků, protože s narůstající délkou fragmentů klesá rozlišovací schopnost elektroforézy. V molekulárně patologické praxi je však tato limitace bezpředmětná, neboť z formolem fixovaného materiálu lze amplifikovat a následně sekvenovat fragment do maximální délky cca 600 bp, většinou pak v rozmezí 200 – 400 bp.
ÚSKALÍ SEKVENAČNÍ ANALÝZY A ZDROJE CHYB
I přes velkou robustnost automatického sekvenování existuje řada faktorů, které mohou ovlivnit kvalitu výsledku. Nejběžnější problémy jsou spojeny s předsekvenačními kroky. Patří sem např. kontaminace PCR, nízká koncentrace (či fragmentace) templátové DNA, templát příliš bohatý na báze cytosin a guanin, či výskyt sekundárních struktur templátu, které mohou způsobit nedostatečnou efektivitu amplifikace během PCR. Další problém může představovat nedostatečná specifičnost nasedání primerů, což může vést ke vzniku nespecifických PCR produktů, či vzájemná komplementarita části primerů vedoucí ke vzniku tzv. primer-dimerů, krátkých PCR produktů interferujích s vlastní sekvenační analýzou v podobě pozadí analyzované sekvence. Také výskyt jednonukleotidových polymorfizmů (SNP) v místě nasedání 3’ konce primeru může natolik znesnadnit hybridizaci primeru k templátu, že dojde k preferenční amplifikaci pouze jedné alely (bez SNP) a případná mutace na druhé alele není zachycena.
Dalším významným zdrojem chyb je přirozená chybovost in vitro používané DNA polymerázy, která je cca 2,5x10-5, tj. 1 chybně vsazená báze na každých 2,5x105 bází nasyntetizovaných v každém cyklu PCR. Výsledný produkt PCR tedy není uniformní, ale jedná se o směs řetězců s chybami v mnoha různých místech. Například po 30ti cyklech obsahuje více než polovina PCR produktů alespoň jeden chybný nukleotid (11). Většinou však řetězec s jednou konkrétní chybnou bází tvoří zanedbatelnou část celkového produktu, takže na sekvenogramu není takováto chybná substituce patrná. Pouze pokud dojde k chybě v některém z počátečních cyklů, řetězec s chybou zaujímá značný podíl ve finálním PCR produktu. Při nalezení mutace na sekvenogramu je proto nutné zopakovat oboustranné sekvenování příslušného fragmentu od PCR kroku z důvodu vyloučení takovéto chyby.
Dalším možným problémem je přítomnost poly–mononukleotidových úseků (např. poly-A) v templátu. V takovém místě dochází k tzv. sklouzávání DNA polymerázy, které vede ke vzniku jednobázové delece (či inzerce) u velkého podílu celkového PCR produktu. Na výsledném sekvenogramu je sekvence od tohoto místa zdvojená či ztrojená (obr. 8). Obě tyto chyby se mohou vyskytnout jak v předsekvenačních krocích tak v sekvenační reakci, byť v prvně jmenované je již z principu metody zachytitelná s výrazně vyšší pravděpodobností. Obě výše zmíněné chyby lze pak částečně omezit použitím tzv. „proofreading“ polymerázy, která má reparační aktivitu a tím nižší frekvenci inkorporace chybného nukleotidu (12).
Obr. 8. Sekvenogram – příklad tzv. sklouzávání polymerázy v místě polymononukleotidových úseků (zde poly-T), které vede ke vniku jednobázové deleci. 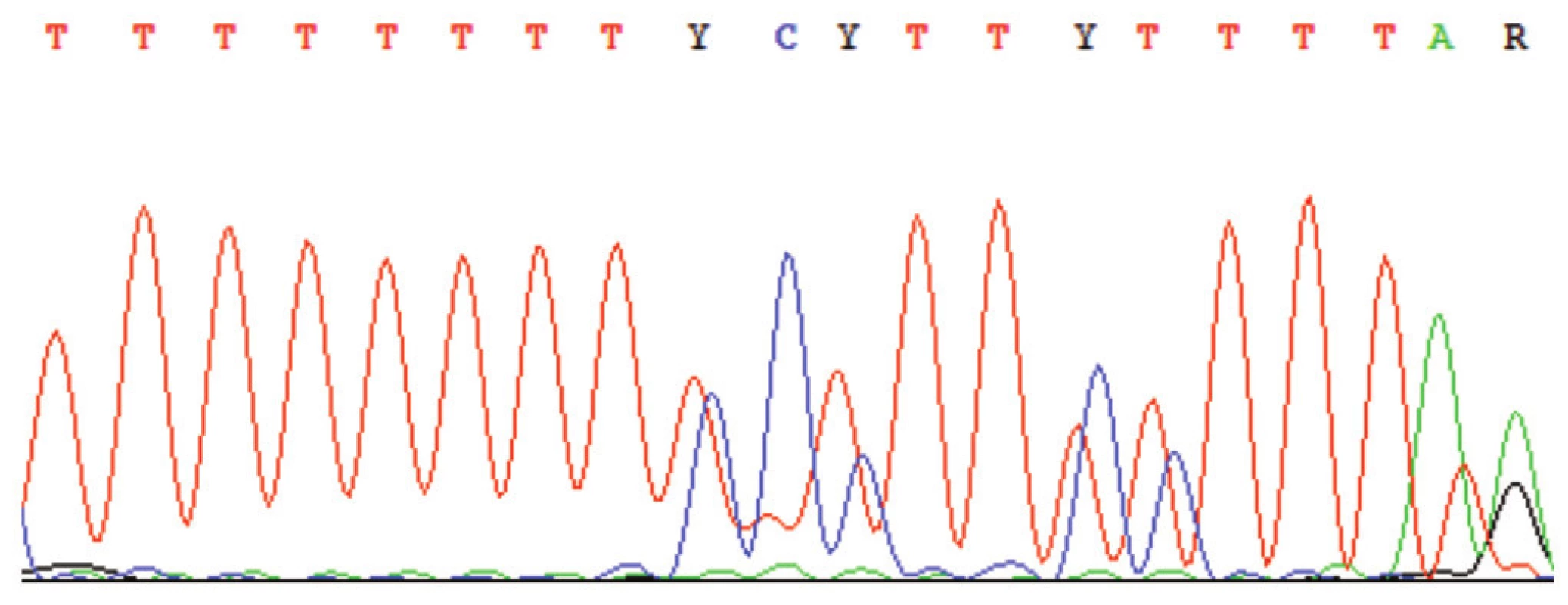
Příčinou chyb mohou být i chemikálie používané k fixaci tkání, které způsobují modifikaci některých bází. Například formaldehydová fixace často vede k přeměně některých cytosinů na uracil procesem zvaným deaminace cytosinu. K uracilu je následně během syntézy připojen jeho komplementární partner adenin a původní pár bází C:G je tak nahrazen párem T:A. Čím méně je templátové DNA v PCR, tím více PCR produkt s chybou převládne v celkovém produktu (13).
Možných problémů spjatých s vlastní sekvenací je též celá řada. Zde vyjmenujeme alespoň ty nejčastější. Především může dojít k úplnému selhání sekvenační reakce. To může být způsobeno např. použitím nevhodných sekvenačních primerů, nebo případným nedostatkem templátové DNA v sekvenační reakci. Nečitelnost sekvenogramu má také řadu možných příčin. Mezi ně patří především nedostatečné přečištění PCR od primerů, kdy na sekvenogramu nacházíme několik (většinou dvě) překrývajících se sekvencí (obr. 9). Obdobný obraz pak např. dostáváme i v případě přítomnosti více produktů v reakci, či při použití primeru, který při dané teplotě hybridizuje na více míst templátu. Případný nadbytek DNA pak způsobuje předčasný pokles píků a tím zkrácení přečteného úseku. Pokud je zvolen nevhodný poměr mezi primery, templátem a ostatními sekvenačními reagenciemi, nebo pokud je produkt reakce špatně přečištěn, dochází k tvorbě tzv. “Dye blobs”, tj. vysokých a širokých píků ve specifických pozicích sekvenogramu (obr. 10). Čitelnost sekvenogramu může být zhoršena i nevhodným nastavením vlastní kapilární elektroforézy. Mezi chyby tohoto druhu se řadí například kontaminace kapiláry předchozím vzorkem DNA, proteiny nebo solemi. Předčasný pokles rozlišení jednotlivých píků může způsobit vysoké napětí či příliš mnoho vzorku v kapiláře (14). Sekvenogram pak ukazuje velmi široké a neostré píky.
Obr. 9. Sekvenogram – příklad výskytu nespecifických píků; nejčastěji z důvodu nedostatečného přečištění PCR od primerů, přítomnosti více produktů v sekvenační reakci, nízké specifičnosti primerů aj. (Označení píků K, M, R, S, W, Y – nomenklatura dle IUPAC-IUB, např. http://www.ncbi.nlm.nih.gov/staff/tao/tools/tool_lettercode.html.) 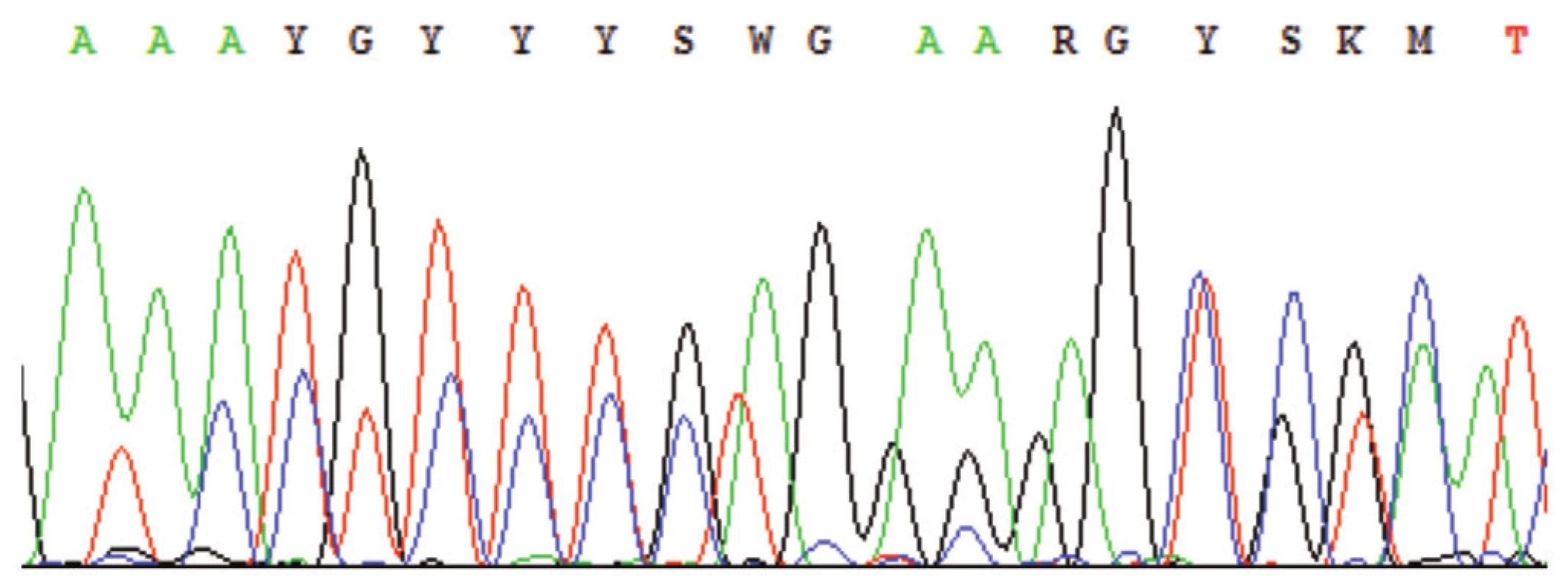
Obr. 10. Sekvenogram – příklad tzv. „Dye blobs“ (vysokých a širokých píků), které vznikají např. při nevhodném poměru mezi primery, reagenciemi BigDye a templátem nebo je produkt sekvenační reakce špatně přečištěn. 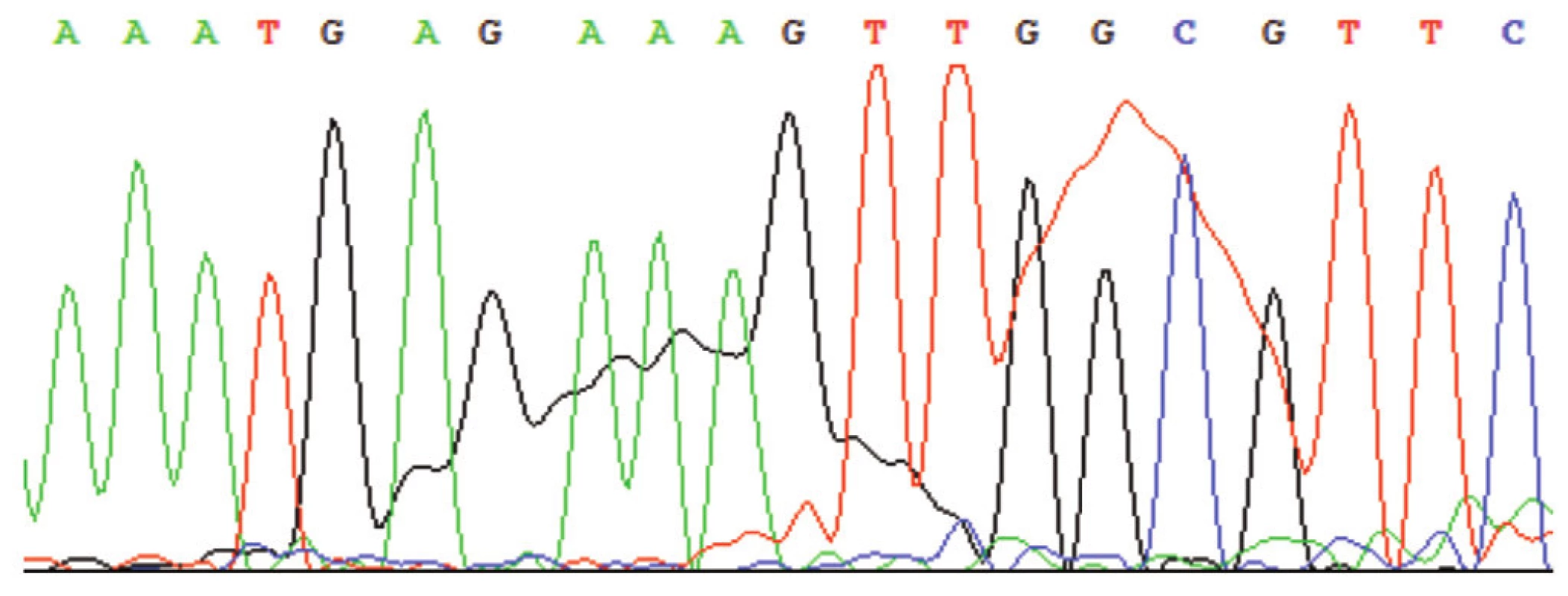
V tomto výčtu by bylo možné dále pokračovat, avšak zmíněné příklady postačují pro informativní nástin problematických zákoutí klasického sekvenování. Případné zájemce odkazujeme např. na: http://www.nucleics.com/DNA_sequencing_support/DNA-sequencing-troubleshooting.html nebo https://www.neb.com/tools-and-resources/troubleshooting-guides/pcr-troubleshooting-guide.
ZÁVĚR
Sangerova metoda určování pořadí bází v nukleové kyselině představovala milník ve vývoji molekulární biologie. Díky ní bylo možné získat obrovské množství dat o dědičné informaci nejrůznějších organizmů a otevřít tak dveře do fascinujícího světa molekulární genetiky a genomiky. Tato revoluční technologie přinesla řadu nových objevů a umožnila rozvoj celé plejády technologických postupů včetně těch, které se dotýkají detekce a léčby nejrůznějších dědičných i sporadických chorob člověka. V současné době jde o nejrozšířenější, a i přes výše zmíněná omezení, nejpřesnější a nejspolehlivější metodu stanovení pořadí bází v nukleové kyselině, která je používána především samostatně při sekvenaci konkrétního úseku DNA a také pro konfirmaci dat získaných metodami „next generation“ sekvenování. S modernizací „next generation“ technik však přestane nést hlavní tíhu zodpovědnosti za získávání nových dat a bude se zřejmě užívat pouze omezeně (pokud vůbec) ke specializovaným účelům.
PODĚKOVÁNÍ
Studie byla podpořena z výzkumného grantu Univerzity Karlovy (projekt číslo P36).
SLOVNÍČEK POJMŮ
Alela – konkrétní varianta genu.
Báze – základní součást nukleových kyselin. Existuje 5 typů: adenin, guanin (purinové báze) a cytosin, thymin, uracil (pyrimidinové báze), které se vzájemně vážou vodíkovými vazbami; uracil se vyskytuje v RNA místo thyminu.
bp – označení pro pár bází
Denaturace – rozvolnění řetězce DNA fyzikálním a/nebo chemickým působením.
Deoxynukleotid (deoxynukleotidtrifosfát, dNTP) – stavební kámen nukleových kyselin složený z nukleové báze (adenin, guanin, cytosin, thymin), monosacharidu (dohromady tvořící deoxynukleozid ) a zbytku kyseliny fosforečné.
Dideoxynukleotid (dideoxynukleotidtrifosfát, ddNTP) – modifikovaný nukleotid, kterému chybí 3’-hydroxylová skupina a na který se proto v řetězci nemůže navázat další deoxy - nebo dideoxynukleotid.
DNA polymeráza – enzym katalyzující polymeraci řetězce DNA.
Homozygot – jedinec se dvěma stejnými alelami pro sledovaný znak.
Heterozygot – jedinec, u kterého se vyskytují u sledovaného znaku dva typy alel.
Hemizygot – jedinec, u kterého je přítomna pouze jedna alela (místo obvyklých dvou), např. při LOH (loss of heterozygosity).
Hybridizace – navázání jednovláknové molekuly RNA či DNA na jinou jednovláknovou molekulu DNA či RNA na základě komplementarity bází.
In silico – prováděné prostřednictvím výpočetní techniky, např. porovnání sekvencí v databázi BLAST.
Komplementarita bází – vytváření dvojic bází prostřednictvím vodíkových můstků podle schématu cytosin – guanin, adenin – thymin/uracil. Toto párování umožňuje přenos genetické informace.
Primer – krátký DNA oligonukleotid komplementární ke konkrétní části genomové DNA či PCR produktu. Jeho přítomnost je nutná pro zahájení syntézy u většiny DNA polymeráz.
Pufr – směs slabé kyseliny a její konjugované zásady, která je schopná udržovat v jistém rozmezí stabilní pH.
Templát, templátová DNA – molekula, která slouží jako předloha při syntéze nového řetězce DNA.
Adresa pro korespondenci:
Ing. Monika Šedivcová
Bioptická laboratoř s.r.o.
Mikulášské nám. 4, 326 00 Plzeň
e-mail: sedivcova@medima.cz
Zdroje
1. Venter JC. The Sequence of the Human Genome. Science 2001; 291(5507): 1304–1351.
2. Sanger F, Nicklen S, Coulson R. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci 1977; 74 : 5463–5467.
3. Maxam AM, Gilbert W. A new method for sequencing DNA. Proc Natl Acad Sci 1977; 74 : 560–564.
4. Murphy KM, Eshleman JR. Simultaneous sequencing of multiple polymerase chain reaction products and combined polymerase chain reaction with cycle sequencing in single reactions. Am J Pathol 2002; 161(1): 27–33.
5. Smith LM, Fung S, Hunkapiller MW, Hunkapiller TJ, Hood LE. The synthesis of oligonucleotides containing an aliphatic amino group at the 5’ terminus: synthesis of fluorescent DNA primers for use in DNA sequence analysis. Nucleic Acids Res 1985; 13 (7): 2399–2412.
6. Skoog DA, Holler FJ, Crouch SR. Principles of Instrumental Analysis. 6th ed. Thomson Brooks/Cole Publishing: Belmont, CA 2007.
7. Shendure J, Ji H. Next-generation DNA sequencing. Nature Biotechnology 2008; 26 : 1135–1145.
8. Huang XC, Quesada MA, Mathies RA. DNA sequencing using capillary array electrophoresis. Anal Chem 1992; 64 : 2149–2154.
9. Franća LTC, Carrilho E, Kist TBL. A review of DNA sequencing techniques. Quarterly Reviews of Biophysics 2002; 35(2): 169–200.
10. Rohlin A, Wernersson J, Engwall Y, Wiklund L, Björk J, Nordling M. Parallel sequencing used in detection of mosaic mutations: comparison with four diagnostic DNA screening techniques. Hum Mutat 2009; 30(6): 1012–1020.
11. Pfeifer JD, Arber DA, Fuller ChE, et al. Molecular Genetic Testing in Surgical Pathology. Philadelphia: Lippincott Williams & Wilkins 2006; 89–90.
12. Dietrich J, Schmitt P, Zieger M, et al. PCR performance of the highly thermostable proof-reading B-type DNA polymerase from Pyrococcus abyssi. FEMS Microbiol Lett 2002; 217 : 89–94.
13. Do H, Dobrovic A. Dramatic reduction of sequence artefacts from DNA isolated from formalin-fixed cancer biopsies by treatment with uracil - DNA glycosylase. Oncotarget 2012; 3(5): 546–558.
14. DNA sequencing by capillary electrophoresis. Applied Biosystems Chemistry Guide. Second Edition. 2009; Part Number 4305080. Rev. C. 05/2009.
15. Coleman WB, Tsongalis GJ. Molecular Diagnostic For the Clinical Laboratorian. Second Edition. Humana Press Inc. Totowa, New Jersey 2006 : 89–102.
Štítky
Patologie Soudní lékařství Toxikologie
Článek Jak se vám líbí?Článek Sekvenování nové generace
Článek vyšel v časopiseČesko-slovenská patologie

2013 Číslo 3-
Všechny články tohoto čísla
- Molekulární a personalizovaná medicína - budoucnost patologie v 21. století
- Rád se dívám na svět i jinak než mikroskopem
- MONITOR aneb nemělo by vám uniknout, že ...
- ÚVOD DO MOLEKULÁRNÍ PATOLOGIE
-
Prof. MUDr. Zdeněk Lukáš, CSc.
In Memoriam - Polymerázová řetězová reakce: princip metody a využití v molekulární patologii
- Sekvenování – klasická metodika
- Jak se vám líbí?
- Sekvenování nové generace
- MONITOR aneb nemělo by vám uniknout, že ...
- Endometrióza v mezotelovej cyste tunica vaginalis testis. Kazuistika
- MONITOR aneb nemělo by vám uniknout, že ...
- Nepravá výduť stěny žilního štěpu u pacienta s implantovaným MGuard typem koronárního stentu: kazuistické sdělení a popis mikroskopických změn
- Obrovskobuněčná intersticiální pneumonie bez souvislosti s expozicí tvrdokovům
- Praktická poznámka pro elektronové mikroskopiky
- Česko-slovenská patologie
- Archiv čísel
- Aktuální číslo
- Informace o časopisu
Nejčtenější v tomto čísle- Polymerázová řetězová reakce: princip metody a využití v molekulární patologii
- Sekvenování – klasická metodika
- Sekvenování nové generace
- Rád se dívám na svět i jinak než mikroskopem
Kurzy
Zvyšte si kvalifikaci online z pohodlí domova
Autoři: prof. MUDr. Vladimír Palička, CSc., Dr.h.c., doc. MUDr. Václav Vyskočil, Ph.D., MUDr. Petr Kasalický, CSc., MUDr. Jan Rosa, Ing. Pavel Havlík, Ing. Jan Adam, Hana Hejnová, DiS., Jana Křenková
Autoři: MUDr. Irena Krčmová, CSc.
Autoři: MDDr. Eleonóra Ivančová, PhD., MHA
Všechny kurzyPřihlášení#ADS_BOTTOM_SCRIPTS#Zapomenuté hesloZadejte e-mailovou adresu, se kterou jste vytvářel(a) účet, budou Vám na ni zaslány informace k nastavení nového hesla.
- Vzdělávání



