-
Články
Top novinky
Reklama- Vzdělávání
- Časopisy
Top články
Nové číslo
- Témata
Top novinky
Reklama- Kongresy
- Videa
- Podcasty
Nové podcasty
Reklama- Kariéra
Doporučené pozice
Reklama- Praxe
Top novinky
ReklamaDlouhé nekódující RNA a karcinom z renálních buněk
Long non-coding RNAs and renal cell carcinoma
Background: To provide an overview of the importance of long non-coding RNAs (lncRNAs) in the pathogenesis of renal cell carcinoma and their utility as biomarkers for diagnosis, prognosis and prediction of treatment response. Materials and methods: A literature search in the Pubmed and Web of Science databases using the keywords variations of “long non-coding RNA” (“lncRNA”, “long noncoding RNA”, “long non-coding RNA”) and “renal cell carcinoma” (“renal cancer”, “renal cell carcinoma”, “kidney cancer”) was performed. The results related to the pathogenesis, diagnosis, prognosis and use as therapeutic targets were separated. Results: Long non-coding RNAs regulate gene expression at different levels. They act both as oncogenes and tumor suppressors. The mechanism of their action has not been fully elucidated, but they are actively involved in the regulation of hypoxia inducible factors pathway, epithelial-mesenchymal transition, cell proliferation, cell cycle regulation, apoptosis, local invasion and development of metastases. Aberrant expression in tumor tissue compared to healthy parenchyma and the correlation of expression levels with clinical-pathological features allow the potential use of many lncRNAs as biomarkers for early detection and prognosis of the disease, including the response to targeted therapy. In vitro assays indicate the potential use of lncRNAs as therapeutic targets.
Conclusion: Our knowledge of long non-coding RNAs in relation to renal cell carcinoma is increasing rapidly. At present, some of them can be considered as promising biomarkers. Further research is needed before they can be introduced into routine clinical practice.
Keywords:
Long non-coding RNA – Prognosis – biomarker – diagnosis – Renal cell carcinoma
Autoři: MUDr. Fedorko Michal, Ph.D.; Febu 1,2; Mgr. Bohošová Júlia 3; doc. MUDr. Poprach Alexandr, Ph.D. 4; Prof. MUDr. Pacík Dalibor, CSc. 1,2
Působiště autorů: Urologická klinika FN Brno 1; LF MU Brno 2; CEITEC, MU Brno 3; Klinika komplexní onkologické péče MOÚ Brno 4
Vyšlo v časopise: Klin Onkol 2020; 33(5): 340-349
Kategorie: Přehled
doi: https://doi.org/10.14735/amko2020340Souhrn
Východiska: Poskytnout přehlednou informaci o významu dlouhých nekódujících RNA (lncRNA) v patogenezi karcinomu z renálních buněk (renal cell carcinoma – RCC) a možnostech jejich využití v diagnostice, stanovení prognózy onemocnění a predikci léčebné odpovědi. Materiál a metody: Vyhledávání v databázích PubMed a Web of Science s využitím variant klíčových slov „dlouhé nekódující RNA“ („lncRNA“, „long noncoding RNA“, „long non-coding RNA“) a „karcinom z renálních buněk“ („renal cancer“, „renal cell carcinoma“, „kidney cancer“). Separace výsledků týkajících se patogeneze, diagnózy, prognózy a využití jako terapeutických cílů. Výsledky: Dlouhé nekódující RNA regulují genovou expresi na všech úrovních. Uplatňují se jako onkogeny i jako nádorové supresory. Mechanizmus jejich působení je objasněn pouze částečně, v patogenezi renálního karcinomu však aktivně regulují kaskádu faktorů indukovaných hypoxií, epiteliálně-mezenchymální tranzici, buněčnou proliferaci, buněčný cyklus, apoptózu, lokální invazi a vznik metastáz. Aberantní exprese ve tkáni nádoru ve srovnání se zdravým renálním parenchymem a korelace expresních hladin s klinicko-patologickými charakteristikami tumoru umožňují potenciální využití mnoha lncRNA jako biomarkerů pro časnou detekci a stanovení prognózy onemocnění vč. odpovědi na cílenou léčbu. Testy in vitro naznačují potenciální využití lncRNA jako terapeutických cílů.
Závěr: Poznatků o dlouhých nekódujících RNA ve vztahu ke karcinomu z renálních buněk rychlým tempem přibývá. V současné době lze některé z nich považovat za slibné biomarkery. Před uvedením do rutinní klinické praxe je potřeba dalšího výzkumu.
Klíčová slova:
biomarker – diagnostika – dlouhé nekódující RNA – karcinom z renálních buněk – prognóza
Úvod
Karcinom z renálních buněk (renal cell carcinoma – RCC) tvoří 4,2 % ze všech zhoubných nádorů u mužů a 2,6 % u žen [1]. Jeho incidence má stoupající tendenci. Nejvyšších hodnot dosahuje celosvětově v ČR, kde byla v roce 2016 standardizovaná incidence 15,17 případů na 100 000 obyvatel [2]. Pětiletým relativním přežitím na úrovni přibližně 75 % se řadí mezi nejletálnější urologické malignity [3]. Poměrně významná část pacientů je stále diagnostikována v pokročilém stadiu onemocnění, v ČR je až 20 % případů diagnostikováno v klinickém stadiu IV [2]. Kromě zobrazovacích vyšetření není k dispozici spolehlivý biomarker, který by byl použitelný v rutinní praxi pro časnou detekci či stanovení prognózy RCC.
Dlouhé nekódující RNA (long non-coding RNAs – lncRNA) patří do skupiny tzv. nekódujících RNA, což jsou transkripty genomu, které nejsou překládány, tedy nekódují funkční proteiny. Hranice mezi krátkými a dlouhými nekódujícími RNA je přibližně 200 nukleotidů. LncRNA regulují genovou expresi na více úrovních – v jádře buňky se uplatňují při modifikacích chromatinu, transkripční regulaci (aktivace i represe) i posttranskripčních úpravách mRNA (sestřih, transport, translace), v cytoplasmě regulují genovou expresi na posttranskripční (stabilita mRNA, vazba s miRNA), translační i posttranslační úrovni [4]. Uplatňují se v patogenezi různých onemocnění vč. zhoubných nádorů [5]. Regulují totiž zásadní projevy malignity, jako jsou buněčný růst, proliferace, invaze, angiogeneze či metastazování [6]. Cílem předkládané práce je přehled deregulovaných lncRNA u renálního karcinomu, popis jejich role v patogenezi RCC a možností využití v diagnostice, stanovení prognózy, příp. jako potenciálních terapeutických cílů.
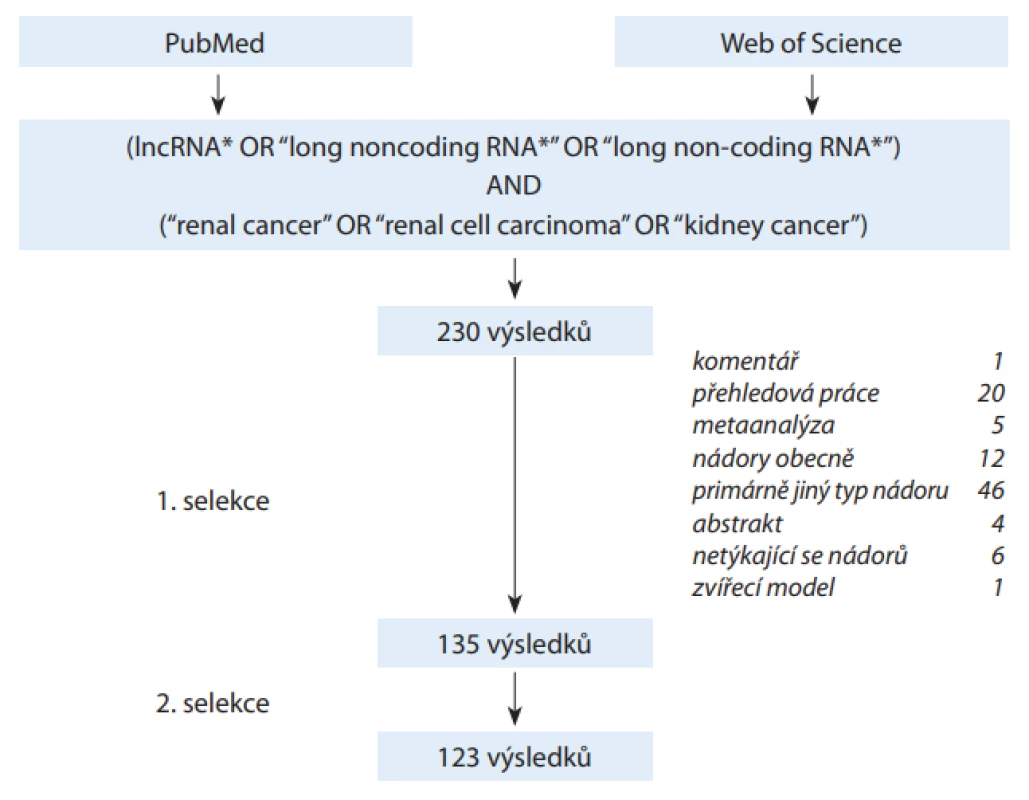
Materiál a metody
Bylo provedeno systematické vyhledávání v databázích Web of Science a Pubmed k datu 3. 10. 2019, zahrnující časové období 2010–2019 a klíčová slova „lncRNA“, „long noncoding RNA“, „long non-coding RNA“, „renal cancer“, „renal cell carcinoma“ a „kidney cancer“. Zadáním („lncRNA*“ OR „long noncoding RNA*“ OR „long non-coding RNA*“) AND („renal cancer“ OR „renal cell carcinoma“ OR „kidney cancer“) v předmětu vyhledávání bylo nalezeno 230 prací, přičemž první relevantní výsledek byl z roku 2011. V první selekci bylo na základě abstrakt vyřazeno 95 prací odpovídajících předem stanoveným vylučovacím kritériím: duplikáty, nedostupný abstrakt, konferenční abstrakta, editorialy, komentáře, přehledové práce, metaanalýzy, zvířecí modely a práce týkající se primárně jiných malignit, zhoubných nádorů obecně nebo nenádorových onemocnění. Následně byla prvním autorem revidována abstrakta zbylých výsledků a ve druhé selekci bylo vyřazeno 12 výsledků, které nepřinášely novou informaci nebo by mohly být pro čtenáře matoucí. Přehled sumarizuje 123 výsledků, u kterých byly revidovány plné texty (schéma 1). Ačkoli se v mnoha pracích překrývá popis biologických funkcí konkrétních lncRNA, jejich prognostický význam i testy in vitro, jsou kvůli přehlednosti uvedeny samostatně. Nejvíce studovaným lncRNA je věnován samostatný prostor.
LncRNA v patogenezi karcinomu z renálních buněk
Tumor supresorové lncRNA
Přehled tumor supresorových lncRNA uvádí tab. 1. Jejich funkce spočívá zejména ve snížení exprese onkogenů nebo útlumu patogenetických drah a regulátorů epiteliálně mezenchymální tranzice (EMT), dále mohou vázat mikroRNA (miRNA, tzv. miRNA „sponging“), příp. destabilizují androgenní receptor (AR). Hladiny těchto lncRNA jsou ve tkáních RCC snížené, což se ve výsledku projevuje stimulací buněčného cyklu, EMT, nadprodukcí onkogenních proteinů, a tím pádem zvýšenou proliferací nádorových buněk, jejich migrací a útlumem apoptózy [7–28].
Tab. 1. Přehled nádorově supresorových lncRNA v patogenezi karcinomu z renálních buněk a jejich biologických funkcí. 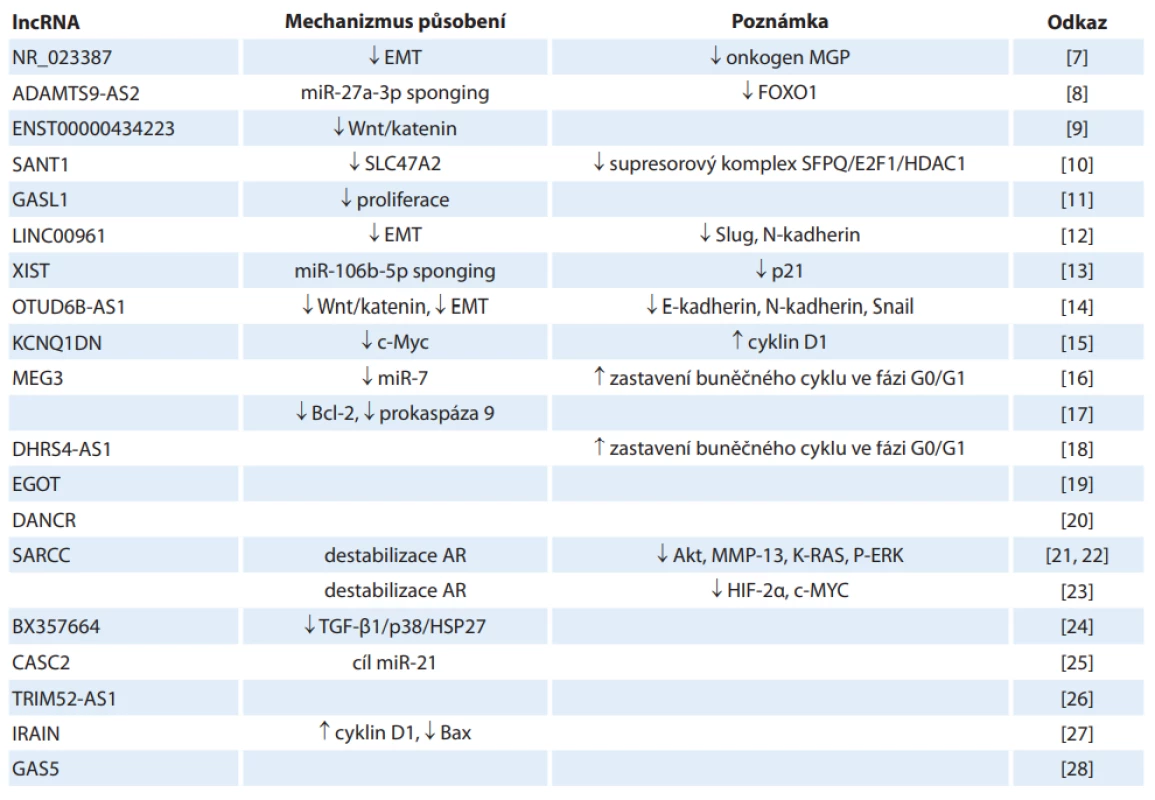
ADAMTS9-AS2 – antisense RNA 2 ADAM metalopeptidáza s trombospondinem typu 1 motivu 9, CASC2 – kandidát na náchylnost k nádorům 2, DANCR – protein nekódující RNA antagonizující diferenciaci, DHRS4-AS1 – antisense RNA 4 z rodiny SDR dehydrogenázy/reduktázy, EGOT – transkript onkogeneze eozinofi lních granulí, EMT – epiteliálně-mezenchymální tranzice, FOXO1 – vidličkový protein O1, GAS5 – transkript 5 specifi cký pro zástavu růstu, HDAC1 – histonová deacetyláza 1, IRAIN – intragenní antisense lncRNA na lokusu IGF1R, MGP – matrixový Gla protein, HSP27 – protein tepelného šoku 27, KCNQ1DN – následný soused KCNQ genu, MEG3 – maternálně exprimovaný gen 3, MMP-13 – matricová metaloproteináza 3, OTUD6B-AS1 – antisense RNA 1 obsahující OTU doménu 6B, SARCC – lncRNA potlačující androgenní receptor u renálního karcinomu, SFPQ – sestřihový faktor bohatý na prolin a glutamin, SLC47A2 – protein 2 vytlačující více léků a toxinů, TGF – transformující růstový faktor, TRIM52-AS1 – antisense RNA 1 z rodiny tripartitních motivů 52, XIST – X-neaktivní specifický transkript
šipka nahoru – zvýšení exprese, šipka dolů – utlumení expreseMEG3
Exprese „maternally expressed gene 3“ je u RCC signifikantně snížená. Indukuje apoptózu buněk RCC aktivací vnitřní mitochondriální dráhy, což vede ke snížení hladiny Bcl-2 a prokaspázy 9 a naopak zvýšení hladin kaspázy 9 a uvolnění cytochromu c do cytoplasmy [17]. Dále tlumí buněčný cyklus („G0/G1 arrest“) prostřednictvím snížení exprese miR-7, která vede k nadprodukci RASL11B (Ras-like protein family member 11B), čímž navíc inhibuje proliferaci, invazi a migraci buněk ccRCC [16].
SARCC
Tato nádorově supresorová lncRNA se váže na androgenní receptor, čímž dochází k jeho destabilizaci a inhibici jeho funkce. Následné potlačení exprese miR-143-3p inhibuje další signály, jako jsou Akt, MMP13, K-RAS a P-ERK, tedy známé onkogeny [21,22]. Destabilizace AR tlumí i další onkogenní dráhu, HIF-2/c-Myc, SARCC se tedy uplatňuje i v regulaci kaskády HIF (hypoxia-inducible factor) [23].
Onkogenní lncRNA
Přehled onkogenních lncRNA je uveden v tab. 2. Tyto lncRNA přímo stimulují produkci jiných onkogenů nebo tlumí expresi nádorových supresorů. V mnoha případech vážou miRNA, čímž přímo blokují jejich působení na cílové mRNA. Stimulují známé patogenetické dráhy RCC – HIF kaskádu, Wnt/katenin, PI3K/Akt, EMT. Ve tkáni nádorů jsou exprimovány ve zvýšené míře [29–88].
Tab. 2. Přehled onkogenních lncRNA v patogenezi karcinomu z renálních buněk a jejich biologických funkcí. 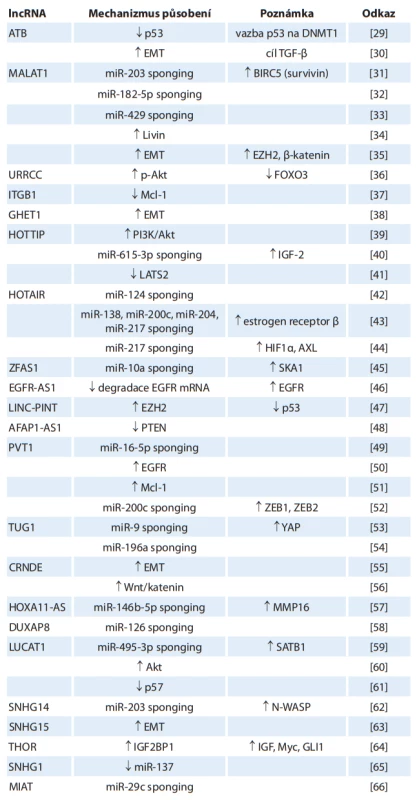
Tab. 2 – pokračování. Přehled onkogenních lncRNA v patogenezi karcinomu z renálních buněk a jejich biologických funkcí. 
šipka dolů – snížení, šipka nahoru – zvýšení
AFAP1-AS1 – antisense RNA 1 asociovaná s proteinem spojeným s aktinovými vlákny 1, Akt – protein kináza 2, ATB – lncRNA aktivovaná transformujícím růstovým faktorem , ANRIL – antisense nekódující RNA na locusu INK4, AVL9 – AVL9 protein spojený s migrací buněk, AXL – tyrozin-protein kinázový receptor UFO, BCL-W – protein 2 podobný Bcl-2, BIRC5 – bakulovirový inhibitor 5 obsahující opakování apoptózy, CCAT1 – transkript 1 spojený s karcinomem tlustého střeva, c-MET – receptor hepatocytového růstového faktoru, CRNDE – protein nekódující gen odlišně exprimovaný u kolorektální neoplázie, CRPAT4 – transkript 4 spojený s prognózou světlobuněčného RCC, DNMT1 – DNA metyltransferáza 1, DUXAP8 – pseudogen 8 dvojitého homeoboxu A, EGFR – receptor epidermálního růstového faktoru, EZH2 – zesilovač zeste homologu 2, EMT – epiteliálně-mezenchymální tranzice, E2F1 – transkripční faktor E2F1, FOXO3 – vidličkový protein O3, GHET1 – transkript 1 vysoce exprimovaný u karcinomu žaludku, GIHCG – lncRNA postupně zvýšená během hepatokarcinogeneze, GLI1 – onkogen spojený s gliomem, HEIRCC – vysoce exprimovaný u renálního karcinomu, HIF1 – hypoxií indukovaný faktor 1 alfa, HOTAIR – antisense RNA transkript HOX genu, HOTTIP – transkript HOX A na distální špičce, HOX – homeobox, IGF – růstový faktor podobný inzulinu, IGF2BP1 – protein 1 vázající mRNA růstového faktoru podobného inzulinu 2, IL-6 – interleukin 6, ITGB1 – integrin beta1, LATS2 – velká nádorově supresorová kináza 2, LINC-PINT – dlouhý intergenní protein nekódující transkript RNA indukovaný p53, lncARSR – lncRNA aktivovaná u RCC rezistentního na sunitinib, LUCAT1 – transkript 1 spojený s karcinomem plic, MALAT1 – transkript 1 spojený s metastazujícím karcinomem plic, MAPK – mitogenem aktivovaná protein kináza, Mcl-1 – protein diferenciace buněk myeloidní leukémie 1, MIAT – transkript spojený s infarktem myokardu, MMP16 – matrixová metaloproteináza 16, MRCCAT1 – transkript 1 spojený s metastatickým renálním karcinomem, Myc – myelocytomatóza, NEAT1 – jaderně obohacený abundantní transkript 1, NPR3 – C receptor natriuretického peptidu, N-WASP – nervový protein Wiskott-Aldrichova syndromu, PI3K – fosfatydilinositol-3-kináza, PTEN – homolog fosfatázy a tenzinu, PVT1 – translokace varianty plazmocytomu 1, RCC – karcinom z renálních buněk, ROR – regulátor přeprogramování, SATB1 – spaciální protein vázající AT bohatou sekvenci, SDC1 – syndekan 1, SKA1 – protein 1 spojený s vřeténkem a kinetochorem, SNHG1, SNHG14, SNHG15 – hostitelský gen pro malou nukleolární RNA 1, 14, 15, SOX4 – transkripční faktor SOX-4, SRLR – lncRNA spojená s rezistencí RCC na sorafenib, STAT3 – signální měnič a aktivátor transkripce 3, TGF- – transformující růstový faktor beta, THOR – vysoce konzervovaná onkogenní lncRNA spojená s varletem, TP73-AS1 – antisense RNA 1 spojená s nádorovým proteinem 73, TUG1 – gen regulovaný taurinem 1, UCA1 – spojená s uroteliálním karcinomem, URRCC – lncRNA BX649059, XIST – X-neaktivní specifi cký transkript, YAP – yes-asociovaný protein 1, ZEB1, ZEB2 – homeobox vázající E-box zinkových prstů 1, 2, ZFAS1 – antisense RNA 1 zinkového prstu NFXHOTAIR
„HOX transcript antisense RNA“ je jednou z nejznámějších onkogenních lncRNA. V patogenezi RCC se typicky uplatňuje miRNa „sponging“. Tímto mechanizmem inhibuje funkci nádorově supresorové miR-124, což vede k nadprodukci 2,8-sialyltransferázy 4, která stimuluje proliferaci, migraci a invazi RCC [42]. Vazba s miR-138, miR-200c, miR-204 nebo miR-217 stimuluje produkci onkogenů, jako jsou ADAM9, EZH2, ZEB1 či ZEB2, přičemž zvýšená exprese HOTAIR je stimulována působením estrogenového receptoru , který se kromě RCC uplatňuje i u jiných nádorových onemocnění [43]. Kompetitivní inhibice miR-217 stimuluje produkci HIF-1 a následně AXL [44].
MALAT1
„Metastasis-associated lung adenocarcinoma transcript 1“, označovaný i jako NEAT2 (nuclear-enriched abundant transcript 2), stimuluje EMT a tlumí expresi miR-205 [35]. Inhibice miR-203 zvyšuje expresi onkogenu BIRS5 (survivinu), což vede ke zvýšení proliferace buněk RCC [31]. MALAT1 váže i další tumor supresorové miRNA, jako jsou miR-182-5p a miR-429 [32,33]. Zvýšená exprese MALAT1 u RCC vede k nadprodukci proteinu Livin, který stimuluje nádorový růst zejména blokováním apoptózy [34].
LUCAT1
„Lung cancer associated transcript 1“ je důležitým regulátorem proliferace. Stimuluje Akt – signální dráhu, dále se váže na polycomb represivní komplex 2 (PRC2), a tlumí tím exprese nádorového supresoru p57 [60,61]. Inhibice miR-495-3p stimuluje proliferaci a invazi nadprodukcí genu SATB1 [59].
HOTTIP
„HOX A transcript at the distal tip“ stimuluje signální dránu PI3K/akt [39]. Kompetitivní inhibice miR-615-3p odblokuje produkci jejího cílového proteinu IGF-2, který má stimulační efekt na růst nádorových buněk [40]. Vazba HOTTIP na EZH2 a specifickou lyzinovou demetylázu 1 tlumí expresi nádorově supresorové kinázy LATS2 [41].
PVT1
„Plasmacytoma variant translocation 1“se aktivně zapojuje do procesu EMT. Vazba s miR-200c stimuluje expresi ZEB1 a ZEB2, které snižují hladinu E-kadherinu. Ve tkáni RCC byla prokázána i vyšší exprese sestřihové varianty bez exonu 4 [52]. Ke stimulaci proliferace, invaze a EMT dochází interakcí PVT1 s miR-16-5p [49]. Zvýšená hladina PVT1 dále stimuluje expresi onkogenu Mcl-1, který je významným inhibitorem apoptózy [51]. Dalším popsaným mechanizmem působení PVT1 je aktivace signální dráhy EGFR [50].
UCA1
„Urothelial cancer associated 1“, původně spojovaný s uroteliálními nádory močového měchýře, se uplatňuje jako onkogen i v patogenezi RCC [71]. Inhibicí exprese miR-129 stimuluje expresi cílového genu SOX4 s antiapoptotickým efektem [69]. Ke stimulaci buněčné proliferace buněk RCC dochází zvýšením exprese EZH2 a interakcí s miR-495. EZH2 dále tlumí expresi nádorového supresoru p21 [70].
lncRNA jako diagnostické biomarkery RCC
Ve srovnání s nenádorovou tkání jsou ve tkáni RCC aberantně exprimovány stovky lncRNA (tab. 3). Výrazně deregulované lncRNA v nádorové tkáni ve srovnání s tkání nenádorovou tedy mohou představovat potenciální diagnostické biomarkery pro odlišení pacientů s RCC od pacientů bez nádoru. Expresní profily lncRNA jsou stanoveny vysokokapacitními metodami jako microarray assay, sekvenování nové generace nebo sekvenování na čipu. Novější práce obvykle využívají dostupné informace z TCGA (The Cancer Genome Atlas), kdy jsou vybrané lncRNA poté validovány na kohortě pacientů s RCC [60,89–96].
Tab. 3. Vybrané studie stanovující expresní profi ly lncRNA u karcinomu z renálních buněk. 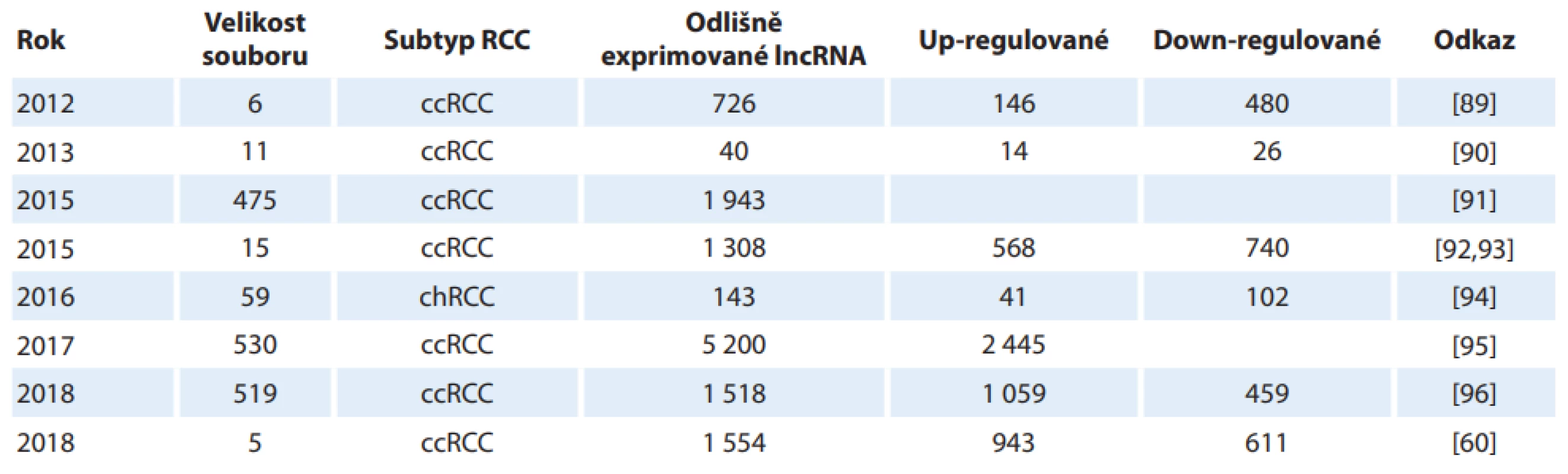
RCC – karcinom z renálních buněk Ačkoli množství prací udává signifikantní rozdíly v expresi jednotlivých lncRNA mezi nádorovou a nenádorovou tkání, pouze v několika z nich se objevují údaje o diagnostické přesnosti umožňující odlišení pacientů od zdravých kontrol, jako je ROC analýza či stanovení senzitivity a specificity [14,18,97]. Výsledky u PVT1 (AUC 0,8567, senzitivita 86,67 %, specificita 76,67 %), LUCAT1 (AUC 0,7756, senzitivita a specificita 90 %) a LINC00982 (AUC 0,9578, senzitivita 76,67 %, specificita 66,67 %) se jeví z hlediska diagnostiky jako velmi slibné [98].
Stanovení hladin cirkulujících lncRNA jako minimálně invazivního způsobu diagnostiky RCC zapadá do konceptu tzv. tekuté biopsie (liquid biopsy). Kromě cirkulujících nádorových buněk a cirkulující nádorové DNA jsou již dostupné práce i o využití lncRNA v detekci různých nádorových onemocnění [99]. Informace o možném využití cirkulujících lncRNA jako biomarkerů RCC jsou zatím nedostatečné. Ojedinělé studie popisují dobrou diskriminační schopnost pro odlišení pacientů s RCC od zdravých kontrol (AUC 0,920, senzitivita 87 %, specificita 84,8 %) a dokonce odlišení pacientů v časném stadiu RCC od zdravých kontrol (AUC 0,886, senzitivita 80,7 %, specificita 84,8 a více) u onkogenní lncRNA GIHCG [72]. Panel pěti cirkulujících lncRNA (LET – low expression in tumor, PVT1 – plasmacytoma variant translocation, PANDAR – promoter of CDKN1A antisense DNA damage activated RNA, PTENP1 – phosphatase and tensin homolog pseudogene 1, linc00963) dokáže spolehlivě odlišit ccRCC od zdravých kontrol v tréninkové (AUC 0,90, senzitivita 79,2 %, specificita 88,9 %) i testovací kohortě (AUC 0,823, senzitivita 67,6 %, specificita 91,4 %). Pro odlišení ccRCC stadia I byla dosažena AUC 0,85 [100]. Sérové hladiny ACTB a MALAT1 nejsou u pacientů s urologickými nádory (vč. RCC) signifikantně vyšší než u nenádorových onemocnění [101].
LncRNA jako prognostické biomarkery
Aberantní exprese lncRNA koreluje u mnoha lncRNA s klinickopatologickými charakteristikami tumoru a ovlivňuje prognózu pacienta. Nejčastěji sledovanými parametry jsou celkové přežití a nádorově specifické přežití. Dostupná data se opírají buď o výzkum jednotlivých lncRNA, nebo se jedná o analýzu dat z TGCA databáze, kdy je obvykle jako prognostický biomarker hodnocen panel několika lncRNA, které jsou použity ke kalkulaci rizikového skóre. V případě onkogenních lncRNA je vyšší exprese vesměs spojena s horší prognózou, u tumor supresorových lncRNA je vztah opačný. Studie zkoumající prognostický význam panelů o různém počtu lncRNA jsou prezentovány v tab. 4 [95,96,102–114]. Přehled jednotlivých lncRNA ve vztahu k prognóze RCC je uveden v tab. 5 [8,9,14,18,35,39,40,45–50,52,55,59–61,66,67,70,72,74,75,78–80,85,97,115–130].
Tab. 4. Panely prognostických lncRNA u karcinomu z renálních buněk. 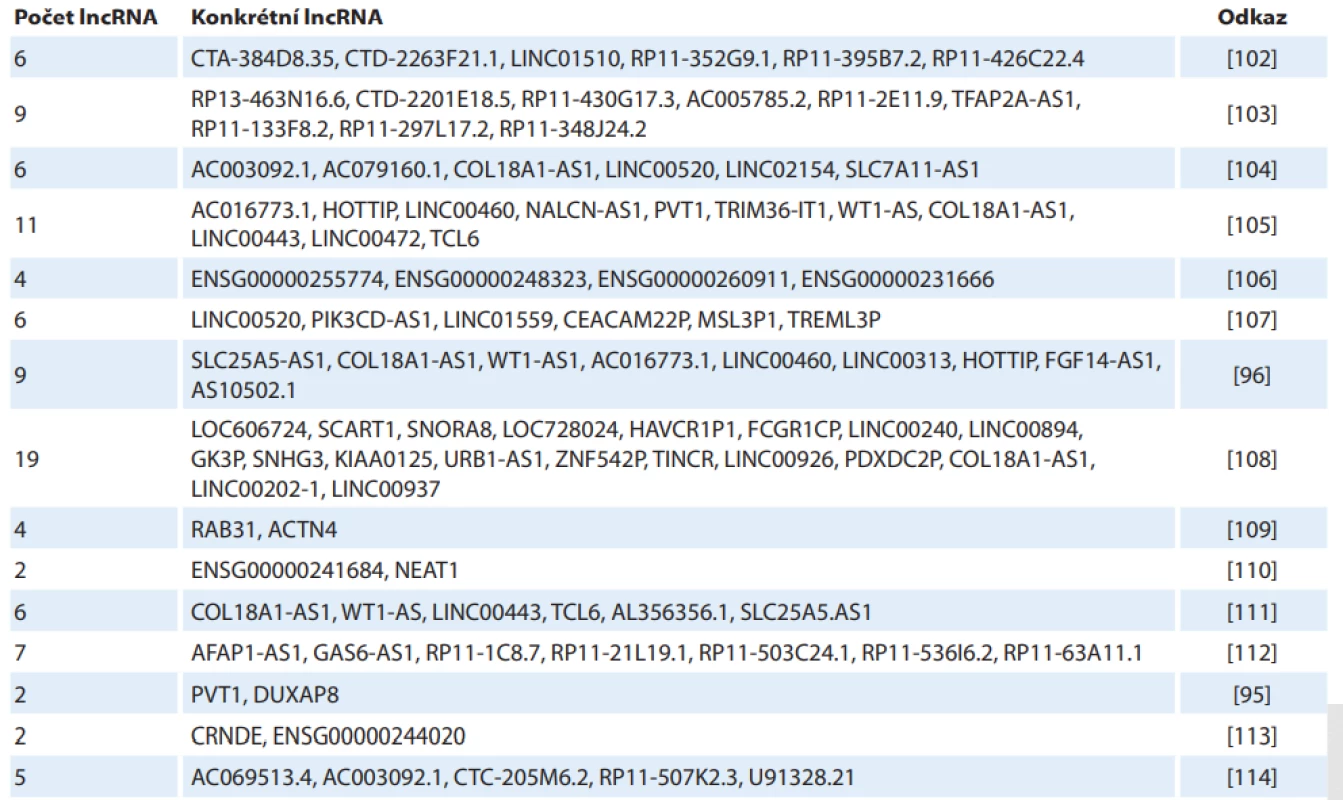
Tab. 5. Vztah expresních hladin jednotlivých lncRNA s prokazatelným vztahem k prognóze karcinomu z renálních buněk. 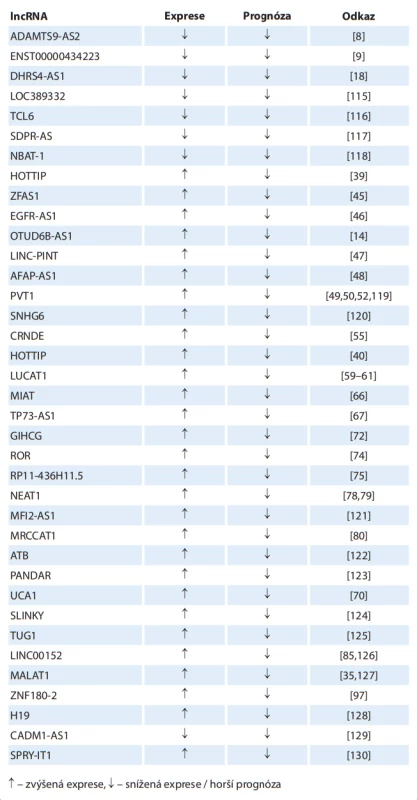
šipka nahoru – zvýšená exprese, šipka dolů – snížená exprese / horší prognóza LncRNA jako prediktivní biomarkery odpovědi na cílenou léčbu
Byla identifikována ARSR (lncRNA activated in RCC with sunitinib resistance), jejíž vysoká hladina exprese koreluje se špatnou odpovědí na sunitinib. Mechanizmus tohoto působení spočívá v kompetitivní inhibici miR-34 a miR-449, čímž dochází ke zvýšení exprese AXL a c-MET v nádorových buňkách. Navíc se ARSR může inkorporovat do exozomů a přenášet i na senzitivní buňky, což dále zvyšuje rezistenci na sunitinib [87]. Některé genetické varianty ARSR, např. rs7859384ARSR, jsou však spojeny s lepší citlivostí na léčbu [131]. Hladina nádorově supresorové lncRNA SARCC (supressing androgen receptor in RCC) se během léčby sunitinibem zvyšuje, což potencuje efekt této léčby a citlivost na ni [21]. Ve vztahu k léčbě sorafenibem je popsána lncRNA SRLR (sorafenib resistence-associated lncRNA in RCC), která je ve zvýšené míře exprimována u RCC rezistentních na sorafenib. Její utlumení senzitizuje původně neodpovídající buňky k léčbě sorafenibem [81]. In vitro testování prokázalo zvýšení senzitivity buněk RCC na sorafenib i po utlumení exprese onkogenní NEAT1 [78].
LncRNA jako potenciální terapeutické cíle
Úvahy o využití lncRNA jako terapeutických cílů vychází z in vitro (příp. in vivo na zvířecích modelech) experimentů, při kterých je exprese studované lncRNA utlumena nebo zvýšena, případně je provedena transfekce buněčných linií konkrétní lncRNA. Následně je pozorován vliv na biologické vlastnosti buněčných linií, jako je proliferace, migrace či apoptóza. Tab. 6 prezentuje výsledky in vitro experimentů u konkrétních lncRNA, které autoři považují za potenciální terapeutické cíle u pacientů s RCC [8,11,14,15,17–20,24–30,32–35,37–40,44,45,47–49,51,56,60,63–65,67–77,79,80,82–84,86,98,117,118,123,124,126–130,132,133].
Tab. 6. lncRNA jako potenciální terapeutické cíle léčby karcinomu z renálních buněk. 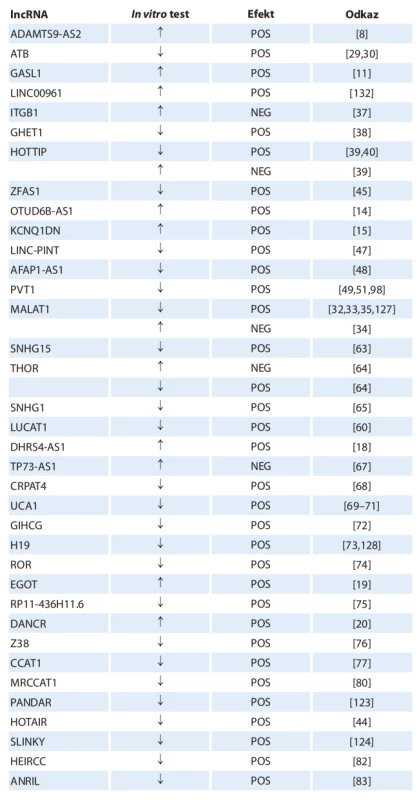
Tab. 6. lncRNA jako potenciální terapeutické cíle léčby karcinomu z renálních buněk 
šipka nahoru – zvýšení exprese, šipka dolů – utlumení exprese, POS – příznivý efekt (potlačení nádorových vlastností v buněčné linii), NEG – nepříznivý efekt (stimulace nádorových vlastností v buněčné linii) Závěr
U RCC je popsána aberantní exprese mnoha lncRNA. Uplatňují se jako onkogeny i jako nádorové supresory a v patogenezi RCC sehrávají důležitou roli při regulaci buněčné proliferace, buněčného cyklu, apoptózy, migrace, invaze a metastazování. Mechanizmus jejich působení zahrnuje známé dráhy jako VHL/HIF kaskáda, Wnt-katenin, PI3K/Akt, dále EMT či přímou regulaci známých onkogenů nebo represi tumor supresorů. V mnoha případech vážou miRNA („sponging“), a brání tak jejich vazbě na mRNA. Odlišné expresní profily, vztah k prognóze onemocnění a reakce na cílenou léčbu naznačují potenciální využití lncRNA jako biomarkerů pro časnou detekci RCC a stanovení jeho prognózy. Výsledky testů na buněčných liniích in vitro („knockdown“ onkogenních lncRNA nebo stimulace nádorově supresorových lncRNA) jsou příslibem pro využití lncRNA jako terapeutických cílů. V době nastupující imunoterapie RCC lze očekávat další intenzivní výzkum lncRNA ve vztahu k receptoru PD-1, resp. PD-L1.
Práce byla podpořena grantovým projektem Ministerstva zdravotnictví ČR AZV NV18-03 - 00554.
This work was supported by grant project of the Ministry of Health of the Czech Republic No. NV18-03-00554.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
The authors declare they have no potential confl icts of interest concerning drugs, products, or services used in the study.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do bi omedicínských časopisů.
The Editorial Board declares that the manuscript met the ICMJE recommendation for biomedical papers.
MUDr. Michal Fedorko, Ph.D., FEBU
Urologická klinika FN Brno
Jihlavská
20625 00 Brno
e-mail: fedorko.michal@fnbrno.cz
Obdrženo/Submitted: 27. 11. 2019
Přijato/Accepted: 10. 12. 2019
Zdroje
1. Miller KD, Nogueira L, Mariotto AB et al. Cancer treatment and survivorship statistics, 2019. CA Cancer J Clin 2019; 69(5): 363–385. doi: 10.3322/caac.21565.
2. Dušek L, Mužík J, Kubásek M et al. Epidemiologie zhoubných nádorů v České republice [online]. Dostupné z: http://www.svod.cz.
3. Howlader N, Noone A, Krapcho M et al. SEER Cancer Statistics Review, 1975–2016. [online]. Dostupné z: http://seer.cancer.gov/csr/1975_2016.
4. Yang L, Froberg JE, Lee JT. Long noncoding RNAs: fresh perspectives into the RNA world. Trends Biochem Sci 2014; 39(1): 35–43. doi: 10.1016/j.tibs.2013.10.002.
5. Schmitt AM, Chang HY. Long noncoding RNAs in cancer pathways. Cancer Cell 2016; 29(4): 452–463. doi: 10.1016/j.ccell.2016.03.010.
6. Gutschner T, Diederichs S. The hallmarks of cancer: a long non-coding RNA point of view. RNA Biol 2012; 9(6): 703–719. doi: 10.4161/rna.20481.
7. Zhou H, Guo L, Yao WM et al. Silencing of tumor-suppressive NR_023387 in renal cell carcinoma via promoter hypermethylation and HNF4A deficiency. J Cell Physiol 2019. [in press]. doi: 10.1002/jcp.29115.
8. Song EL, Xing L, Wang L et al. LncRNA ADAMTS9-AS2 inhibits cell proliferation and decreases chemoresistance in clear cell renal cell carcinoma via the miR-27a-3p/FOXO1 axis. Aging 2019; 11(15): 5705–5725. doi: 10.18632/aging.102154.
9. Luo NQ, Ma DR, Yang XC et al. Long non-coding RNA ENST00000434223 inhibits the progression of renal cancer through Wnt/hygro-catenin signaling pathway. Eur Rev Med Pharmacol Sci 2019; 23(16): 6868–6877. doi: 10.26355/eurrev_201908_18726.
10. Gao ZZ, Chen MJ, Tian XK et al. A novel human lncRNA SANT1 cis-regulates the expression of SLC47A2 by altering SFPQ/E2F1/HDAC1 binding to the promoter region in renal cell carcinoma. RNA Biol 2019; 16(7): 940–949. doi: 10.1080/15476286.2019.1602436.
11. Dong JP, Zheng SP, Yang XY et al. Cell proliferation in kidney carcinoma is inhibited by lncRNA GASL1. Eur J Inflamm 2019; 17 : 1–6. doi: 10.1177/2058739219854598.
12. Chen DM, Zhu M, Su H et al. LINC00961 restrains cancer progression via modulating epithelial-mesenchymal transition in renal cell carcinoma. J Cell Physiol 2019; 234(5): 725–765. doi: 10.1002/jcp.27483.
13. Sun K, Jia ZK, Duan RR et al. Long non-coding RNA XIST regulates miR-106b-5p/P21 axis to suppress tumor progression in renal cell carcinoma. Biochem Biophys Res Commun 2019; 510(3): 416–420. doi: 10.1016/j.bbrc.2019.01.116.
14. Wang G, Zhang ZJ, Jian WG et al. Novel long noncoding RNA OTUD6B-AS1 indicates poor prognosis and inhibits clear cell renal cell carcinoma proliferation via the Wnt/beta-catenin signaling pathway. Mol Cancer 2019; 18 : 1–15. doi: 10.1186/s12943-019-0942-1.
15. Yang F, Wu QJ, Zhang L et al. The long noncoding RNA KCNQ1DN suppresses the survival of renal cell carcinoma cells through downregulating c-Myc. J Cancer 2019; 10(19): 4662–4670. doi: 10.7150/jca.29280.
16. He HC, Dai J, Zhuo R et al. Study on the mechanism behind lncRNA MEG3 affecting clear cell renal cell carcinoma by regulating miR-7/RASL11B signaling. J Cell Physiol 2018; 233(12): 9503–9515. doi: 10.1002/jcp.26849.
17. Wang M, Huang T, Luo G et al. Long non-coding RNA MEG3 induces renal cell carcinoma cells apoptosis by activating the mitochondrial pathway. J Huazhong Univ Sci Technol Med Sci 2015; 35(4): 541–545. doi: 10.1007/s11596-015-1467-5.
18. Wang CL, Wang G, Zhang ZJ et al. The downregulated long noncoding RNA DHRS4-AS1 is protumoral and associated with the prognosis of clear cell renal cell carcinoma. Onco Targets Ther 2018; 11 : 5631–5646. doi: 10.2147/OTT.S164984.
19. Jin L, Quan J, Pan X et al. Identification of lncRNA EGOT as a tumor suppressor in renal cell carcinoma. Mol Med Rep 2017; 16(5): 7072–7079. doi: 10.3892/mmr.2017.7470.
20. Jin L, Fu HF, Quan J et al. Overexpression of long non-coding RNA differentiation antagonizing non-protein coding RNA inhibits the proliferation, migration and invasion and promotes apoptosis of renal cell carcinoma. Mol Med Rep 2017; 16(4): 4463–4468. doi: 10.3892/mmr.2017.7135.
21. Zhai W, Sun Y, Guo CC et al. LncRNA-SARCC suppresses renal cell carcinoma (RCC) progression via altering the androgen receptor (AR)/miRNA-143-3p signals. Cell Death and Differ 2017; 24(9): 1502–1517. doi: 10.1038/cdd.2017.74.
22. Zhai W, Chang C, Zheng JH et al. LncRNA-SARCC suppresses clear cell renal cell carcinoma (RCC) progression via altering the androgen receptor (AR)/miRNA-143-3p signals. Cell Death Differ 2017; 24(9): 1502–1517. doi: 10.1038/cdd.2017.74.
23. Zhai W, Sun Y, Jiang M et al. Differential regulation of LncRNA-SARCC suppresses VHL-mutant RCC cell proliferation yet promotes VHL-normal RCC cell proliferation via modulating androgen receptor/HIF-2 alpha/C-MYC axis under hypoxia. Oncogene 2016; 35(37): 4866–4880. doi: 10.1038/onc.2016.19.
24. Liu YY, Qian J, Li X et al. Long noncoding RNA BX357664 regulates cell proliferation and epithelial-to-mesenchymal transition via inhibition of TGF-beta 1/p38/HSP27 signaling in renal cell carcinoma. Oncotarget 2016; 7(49): 81410–81422. doi: 10.18632/oncotarget.12937.
25. Cao YJ, Xu RF, Xu XL et al. Downregulation of lncRNA CASC2 by microRNA-21 increases the proliferation and migration of renal cell carcinoma cells. Mol Med Rep 2016; 14(1): 1019–1025. doi: 10.3892/mmr.2016.5337.
26. Liu Z, Yan HY, Xia SY et al. Downregulation of long non-coding RNA TRIM52-AS1 functions as a tumor suppressor in renal cell carcinoma. Mol Med Rep 2016; 13(4): 3206–3212. doi: 10.3892/mmr.2016.4908.
27. Wang ZQ, Liu Q, Li TQ et al. Abnormal expressed long non-coding RNA IRAIN inhibits tumor progression in human renal cell carcinoma cells. Open Life Sci 2016; 11(1): 200–205. doi: 10.1515/biol-2016-0026.
28. Qiao HP, Gao WS, Huo JX et al. Long non-coding RNA GAS5 functions as a tumor suppressor in renal cell carcinoma. Asian Pac J Cancer Prev 2013; 14(2): 1077–1082. doi: 10.7314/apjcp.2013.14.2.1077.
29. Song C, Xiong YH, Liao WB et al. Long noncoding RNA ATB participates in the development of renal cell carcinoma by downregulating p53 via binding to DNMT1. J Cell Physiol 2019; 234(8): 12910–12917. doi: 10.1002/jcp.27957.
30. Xiong J, Liu Y, Jiang L et al. High expression of long non-coding RNA lncRNA-ATB is correlated with metastases and promotes cell migration and invasion in renal cell carcinoma. Jpn J Clin Oncol 2016; 46(4): 378–384. doi: 10.1093/jjco/hyv214.
31. Zhang HM, Li W, Gu WY et al. MALAT1 accelerates the development and progression of renal cell carcinoma by decreasing the expression of miR-203 and promoting the expression of BIRC5. Cell Prolif 2019; 52(5): e12640. doi: 10.1111/cpr.12640.
32. Kulkarni P, Dasgupta P, Bhat NS et al. Elevated miR-182-5p associates with renal cancer cell mitotic arrest through diminished MALAT-1 expression. Mol Cancer Res 2018; 16(11): 1750–1760. doi: 10.1158/1541-7786.MCR-17-0762.
33. Jiang LT, Wan CH, Guo QH et al. Long noncoding RNA metastasis-associated lung adenocarcinoma transcript 1 (MALAT1) promotes renal cell carcinoma progression via sponging miRNA-429. Med Sci Monit 2018; 24 : 1794–1801. doi: 10.12659/MSM.909450.
34. Chen SA, Ma PP, Zhao Y et al. Biological function and mechanism of MALAT-1 in renal cell carcinoma proliferation and apoptosis: role of the MALAT-1-Livin protein interaction. J Physiol Sci 2017; 67(5): 577–585. doi: 10.1007/s12576-016-0486-8.
35. Hirata H, Hinoda Y, Shahryari V et al. Long noncoding RNA MALAT1 promotes aggressive renal cell carcinoma through Ezh2 and interacts with miR-205. Cancer Res 2015; 75(7): 1322–1331. doi: 10.1158/0008-5472.CAN-14-2931.
36. Zhai W, Zhu R, Ma J et al. A positive feed-forward loop between LncRNA-URRCC and EGFL7/P-AKT/FOXO3 signaling promotes proliferation and metastasis of clear cell renal cell carcinoma. Mol Cancer 2019; 18(1): 81. doi: 10.1186/s12943-019-0998-y.
37. Zheng XL, Zhang YY, Lv WG. Long noncoding RNA ITGB1 promotes migration and invasion of clear cell renal cell carcinoma by downregulating Mcl-1. Eur Rev Med Pharmacol Sci 2019; 23(5): 1996–2002. doi: 10.26355/eurrev_201903_17238.
38. Xie WJ, Chen Q, Liu X et al. Silencing of the long non-coding RNA GHET1 inhibits cell proliferation and migration of renal cell carcinoma through epithelial-mesenchymal transition. Oncol Lett 2019; 17(3): 3173–3180. doi: 10.3892/ol.2019.9967.
39. Su Y, Lu JX, Chen XG et al. Long non-coding RNA HOTTIP affects renal cell carcinoma progression by regulating autophagy via the PI3K/Akt/Atg13 signaling pathway. J Cancer Res Clin Oncol 2019; 145(3): 573–588. doi: 10.1007/s00432-018-2808-0.
40. Wang QF, Wu GZ, Zhang ZW et al. Long non-coding RNA HOTTIP promotes renal cell carcinoma progression through the regulation of the miR-615/IGF-2 pathway. Int J Oncol 2018; 53(5): 2278–2288. doi: 10.3892/ijo.2018.4539.
41. Peng FF, Shi XL, Meng Y et al. Long non-coding RNA HOTTIP is upregulated in renal cell carcinoma and regulates cell growth and apoptosis by epigenetically silencing of LATS2. Biomed Pharmacother 2018; 105 : 1133–1140. doi: 10.1016/j.biopha.2018.06.081.
42. Pan Y, Wu YJ, Hu JL et al. Long noncoding RNA HOTAIR promotes renal cell carcinoma malignancy through alpha-2,8-sialyltransferase 4 by sponging microRNA-124. Cell Prolif 2018; 51(6): e12507. doi: 10.1111/cpr.12507.
43. Ding J, Yeh CR, Sun Y et al. Estrogen receptor beta promotes renal cell carcinoma progression via regulating LncRNA HOTAIR-miR-138/200c/204/217 associated CeRNA network. Oncogene 2018; 37(37): 5037–5053. doi: 10.1038/s41388-018-0175-6.
44. Hong Q, Li O, Zheng W et al. LncRNA HOTAIR regulates HIF-1 alpha/AXL signaling through inhibition of miR-217 in renal cell carcinoma. Cell Death Dis 2017; 8: e2772. doi: 10.1038/cddis.2017.181.
45. Dong D, Mu ZY, Wei N et al. Long non-coding RNA ZFAS1 promotes proliferation and metastasis of clear cell renal cell carcinoma via targeting miR-10a/SKA1 pathway. Biomed Pharmacother 2019; 111 : 917–925. doi: 10.1016/j.biopha.2018.12.143.
46. Wang AB, Bao Y, Wu ZJ et al. Long noncoding RNA EGFR-AS1 promotes cell growth and metastasis via affecting HuR mediated mRNA stability of EGFR in renal cancer. Cell Death Dis 2019; 10 : 154. doi: 10.1038/s41419-019-1331-9.
47. Duan JY, Ma X, Shi J et al. Long noncoding RNA LINC-PINT promotes proliferation through EZH2 and predicts poor prognosis in clear cell renal cell carcinoma. Onco Targets Ther 2019; 12 : 4729–4740. doi: 10.2147/OTT.S202938.
48. Mu ZY, Dong D, Wei N et al. Silencing of lncRNA AFAP1-AS1 inhibits cell growth and metastasis in clear cell renal cell carcinoma. Oncol Res 2019; 27(6): 653–661. doi: 10.3727/096504018X15420748671075.
49. Ren Y, Huang WP, Weng GB et al. LncRNA PVT1 promotes proliferation, invasion and epithelial-mesenchymal transition of renal cell carcinoma cells through downregulation of miR-16-5p. Onco Targets Ther 2019; 12 : 2563–2575. doi: 10.2147/OTT.S190239.
50. Li WC, Zheng ZS, Chen HC et al. Knockdown of long non-coding RNA PVT1 induces apoptosis and cell cycle arrest in clear cell renal cell carcinoma through the epidermal growth factor receptor pathway. Oncol Lett 2018; 15(5): 7855–7863. doi: 10.3892/ol.2018.8315.
51. Wu QJ, Yang F, Yang ZX et al. Long noncoding RNA PVT1 inhibits renal cancer cell apoptosis by up-regulating Mcl-1. Oncotarget 2017; 8(60): 101865–101875. doi: 10.18632/oncotarget.21706.
52. Yang T, Zhou H, Liu PJ et al. lncRNA PVT1 and its splicing variant function as competing endogenous RNA to regulate clear cell renal cell carcinoma progression. Oncotarget 2017; 8(49): 85353–85367. doi: 10.18632/oncotarget.19743.
53. Liu S, Yang YT, Wang WW et al. Long noncoding RNA TUG1 promotes cell proliferation and migration of renal cell carcinoma via regulation of YAP. J Cell Biochem 2018; 119(12): 9694–9706. doi: 10.1002/jcb.27284.
54. Yang Y, Sun DM, Yu JF et al. Long noncoding RNA TUG1 promotes renal cell carcinoma cell proliferation, migration and invasion by downregulating microRNA-196a. Mol Med Rep 2018; 18(6): 5791–5798. doi: 10.3892/mmr.2018.9608.
55. Ding CG, Han F, Xiang HL et al. LncRNA CRNDE is a biomarker for clinical progression and poor prognosis in clear cell renal cell carcinoma. J Cell Biochem 2018; 119(12): 10406–10414. doi: 10.1002/jcb.27389.
56. Shao K, Shi TM, Yang Y et al. Highly expressed lncRNA CRNDE promotes cell proliferation through Wnt/beta-catenin signaling in renal cell carcinoma. Tumor Biol 2016; 37(12): 15997–16004. doi: 10.1007/s13277-016-5440-0.
57. Yang FQ, Zhang JQ, Jin JJ et al. HOXA11-AS promotes the growth and invasion of renal cancer by sponging miR-146b-5p to upregulate MMP16 expression. J Cell Physiol 2018; 233(12): 961–969. doi: 10.1002/jcp.26864.
58. Huang T, Wang X, Yang XK et al. Long non-coding RNA DUXAP8 enhances renal cell carcinoma progression via downregulating miR-126. Med Sci Monit 2018; 24 : 7340–7347. doi: 10.12659/MSM.910054.
59. Wang LN, Zhu XQ, Song XS et al. Long noncoding RNA lung cancer associated transcript 1 promotes proliferation and invasion of clear cell renal cell carcinoma cells by negatively regulating miR-495-3p. J Cell Biochem 2018; 119(9): 7599–7609. doi: 10.1002/jcb.27099.
60. Zheng ZS, Zhao FJ, Zhu DJ et al. Long non-coding RNA LUCAT1 promotes proliferation and invasion in clear cell renal cell carcinoma through AKT/GSK-3 beta signaling pathway. Cell Physiol Biochem 2018; 48(3): 891–904. doi: 10.1159/000491957.
61. Xiao HB, Bao L, Xiao W et al. Long non-coding RNA Lucat1 is a poor prognostic factor and demonstrates malignant biological behavior in clear cell renal cell carcinoma. Oncotarget 2017; 8(69): 113622–113634. doi: 10.18632/oncotarget.21185.
62. Liu GH, Ye ZX, Zhao X et al. SP1-induced up-regulation of lncRNA SNHG14 as a ceRNA promotes migration and invasion of clear cell renal cell carcinoma by regulating N-WASP. Am J Cancer Res 2017; 7(12): 2515–2525.
63. Du Y, Kong CZ, Zhu YY et al. Knockdown of SNHG15 suppresses renal cell carcinoma proliferation and EMT by regulating the NF-B signaling pathway. Int J Oncol 2018; 53(1): 384–394. doi: 10.3892/ijo.2018.4395.
64. Ye XT, Huang H, Huang WP et al. LncRNA THOR promotes human renal cell carcinoma cell growth. Biochem Biophys Res Commun 2018; 501(3): 661–667. doi: 10.1016/j.bbrc.2018.05.040.
65. Zhao SY, Wang YW, Luo MY et al. Long noncoding RNA small nucleolar RNA host gene 1 (SNHG1) promotes renal cell carcinoma progression and metastasis by negatively regulating miR-137. Med Sci Monit 2018; 24 : 3824–3831. doi: 10.12659/MSM.910866.
66. Qu Y, Xiao HB, Xiao W et al. Upregulation of MIAT regulates LOXL2 expression by competitively binding MiR-29c in clear cell renal cell carcinoma. Cell Physiol Biochem 2018; 48(3): 1075–1087. doi: 10.1159/000491974.
67. Liu GH, Zhao X, Zhou JM et al. LncRNA TP73-AS1 promotes cell proliferation and inhibits cell apoptosis in clear cell renal cell carcinoma through repressing KISS1 expression and inactivation of PI3K/Akt/mTOR signaling pathway. Cell Physiol Biochem 2018; 48(1): 371–384. doi: 10.1159/000491767.
68. Zhang WH, Wang J, Chai R et al. Hypoxia-regulated lncRNA CRPAT4 promotes cell migration via regulating AVL9 in clear cell renal cell carcinomas. Onco Targets Ther 2018; 11 : 4537–4545. doi: 10.2147/OTT.S169155.
69. Liu Q, Li Y, Lv WL et al. UCA1 promotes cell proliferation and invasion and inhibits apoptosis through regulation of the miR129-SOX4 pathway in renal cell carcinoma. Onco Targets Ther 2018; 11 : 2475–2487. doi: 10.2147/OTT.S160192.
70. Lu Y, Liu WG, Lu JH et al. LncRNA UCA1 promotes renal cell carcinoma proliferation through epigenetically repressing p21 expression and negatively regulating miR-495. Tumor Biol 2017; 39(5). doi: 10.1177/1010428317701632.
71. Li YF, Wang TT, Li YC et al. Identification of long-non coding RNA UCA1 as an oncogene in renal cell carcinoma. Mol Med Rep 2016; 13(4): 3326–3334. doi: 10.3892/mmr.2016.4894.
72. He ZH, Qin XH, Zhang XL et al. Long noncoding RNA GIHCG is a potential diagnostic and prognostic biomarker and therapeutic target for renal cell carcinoma. Eur Rev Med Pharmacol Sci 2018; 22(1): 46–54. doi: 10.26355/eurrev_201801_14099.
73. He HW, Wang NN, Yi XM et al. Long non-coding RNA H19 regulates E2F1 expression by competitively sponging endogenous miR-29a-3p in clear cell renal cell carcinoma. Cell Biosci 2017; 7 : 65. doi: 10.1186/s13578-017-0193-z.
74. Shi JG, Zhang W, Tian HY et al. LncRNA ROR promotes the proliferation of renal cancer and is negatively associated with favorable prognosis. Mol Med Rep 2017; 16(6): 9561–9565. doi: 10.3892/mmr.2017.7775.
75. Wang KF, Jin W, Song Y et al. LncRNA RP11-436H11.5, functioning as a competitive endogenous RNA, upregulates BCL-W expression by sponging miR-335-5p and promotes proliferation and invasion in renal cell carcinoma. Mol Cancer 2017; 16(1): 166. doi: 10.1186/s12943-017-0735-3.
76. He XF, Liu HJ, Guo FF et al. Long non-coding RNA Z38 promotes cell proliferation and metastasis in human renal cell carcinoma. Mol Med Rep 2017; 16(4): 5489–5494. doi: 10.3892/mmr.2017.7218.
77. Chen SA, Ma PP, Li B et al. LncRNA CCAT1 inhibits cell apoptosis of renal cell carcinoma through up-regulation of Livin protein. Mol Cell Biochem 2017; 434(1–2): 135–142. doi: 10.1007/s11010-017-3043-8.
78. Liu F, Chen N, Gong YC et al. The long non-coding RNA NEAT1 enhances epithelial-to-mesenchymal transition and chemoresistance via the miR-34a/c-Met axis in renal cell carcinoma. Oncotarget 2017; 8(38): 62927–62938. doi: 10.18632/oncotarget.17757.
79. Ning L, Li ZG, Wei DJ et al. LncRNA, NEAT1 is a prognosis biomarker and regulates cancer progression via epithelial-mesenchymal transition in clear cell renal cell carcinoma. Cancer Biomark 2017; 19(1): 75–83. doi: 10.3233/CBM-160376.
80. Li JK, Chen C, Liu JY et al. Long noncoding RNA MRCCAT1 promotes metastasis of clear cell renal cell carcinoma via inhibiting NPR3 and activating p38-MAPK signaling. Mol Cancer 2017; 16(1): 111. doi: 10.1186/s12943-017-0681-0.
81. Xu Z, Yang F, Wei D et al. Long noncoding RNA-SRLR elicits intrinsic sorafenib resistance via evoking IL-6/STAT3 axis in renal cell carcinoma. Oncogene 2017; 36(14): 1965–1977. doi: 10.1038/onc.2016.356.
82. Xiong J, Liu Y, Luo SJ et al. High expression of the long non-coding RNA HEIRCC promotes renal cell carcinoma metastasis by inducing epithelial-mesenchymal transition. Oncotarget 2017; 8(4): 6555–6563. doi: 10.18632/oncotarget.14149.
83. Li QC, Tian Y, Hu GR et al. Highly expressed antisense noncoding RNA in the INK4 locus promotes growth and invasion of renal clear carcinoma cells via the beta-catenin pathway. Oncol Res 2017; 25(8): 1373–1382. doi: 10.3727/096504017X14878509668646.
84. Zhang JL, Cao ZJ, Ding X et al. The lncRNA XIST regulates the tumorigenicity of renal cell carcinoma cells via the miR-302c/SDC1 axis. Int J Clin Exp Pathol 2017; 10(7): 7481–7491.
85. Wang YJ, Liu JZ, Bai HZ et al. Long intergenic non-coding RNA 00152 promotes renal cell carcinoma progression by epigenetically suppressing P16 and negatively regulates miR-205. Am J Cancer Res 2017; 7(2): 312–322.
86. Ren XF, Lan TB, Chen Y et al. LncRNA uc009yby.1 promotes renal cell proliferation and is associated with poor survival in patients with clear cell renal cell carcinomas. Oncol Lett 2016; 12(3): 1929–1934. doi: 10.3892/ol.2016.4856.
87. Qu L, Ding J, Chen C et al. Exosome-transmitted lncARSR promotes sunitinib resistance in renal cancer by acting as a competing endogenous RNA. Cancer Cell 2016; 29(5): 653–668. doi: 10.1016/j.ccell.2016.03.004.
88. Bertozzi D, Iurlaro R, Sordet O et al. Characterization of novel antisense HIF-1 alpha transcripts in human cancers. Cell Cycle 2011; 10(18): 3189–3197. doi: 10.4161/cc.10.18.17183.
89. Yu G, Yao WM, Wang J et al. LncRNAs expression signatures of renal clear cell carcinoma revealed by microarray. Plos One 2012; 7(8): e42377. doi: 10.1371/journal.pone.0042377.
90. Fachel AA, Tahira AC, Vilella-Arias SA et al. Expression analysis and in silico characterization of intronic long noncoding RNAs in renal cell carcinoma: emerging functional associations. Mol Cancer 2013; 12(1): 140. doi: 10.1186/1476-4598-12-140.
91. Malouf GG, Zhang JP, Yuan Y et al. Characterization of long non-coding RNA transcriptome in clear-cell renal cell carcinoma by next-generation deep sequencing. Mol Oncol 2015; 9(1): 32–43. doi: 10.1016/j.molonc.2014.07.007.
92. Blondeau JJ, Deng M, Syring I et al. Identification of novel long non-coding RNAs in clear cell renal cell carcinoma. Clin Epigenetics 2015; 7(1): 10. doi: 10.1186/s13148-015-0047-7.
93. Deng M, Blondeau JJ, Schmidt D et al. Identification of novel differentially expressed lncRNA and mRNA transcripts in clear cell renal cell carcinoma by expression profiling. Genom Data 2015; 5 : 173–175. doi: 10.1016/j.gdata.2015.06.016.
94. He HT, Xu M, Kuang Y et al. Biomarker and competing endogenous RNA potential of tumor-specific long noncoding RNA in chromophobe renal cell carcinoma. Onco Targets Ther 2016; 9 : 6399–6406. doi: 10.2147/OTT.S116392.
95. Xu X, Xu YC, Shi CQ et al. A genome-wide comprehensively analyses of long noncoding RNA profiling and metastasis associated lncRNAs in renal cell carcinoma. Oncotarget 2017; 8(50): 87773–87781. doi: 10.18632/oncotarget.21206.
96. Yin H, Wang XY, Zhang X et al. Integrated analysis of long noncoding RNA associated-competing endogenous RNA as prognostic biomarkers in clear cell renal carcinoma. Cancer Sci 2018; 109(10): 3336–3349. doi: 10.1111/cas.13778.
97. Ellinger J, Alam J, Rothenburg J et al. The long non-coding RNA lnc-ZNF180-2 is a prognostic biomarker in patients with clear cell renal cell carcinoma. Am J Cancer Res 2015; 5(9): 2799–2807.
98. Fedorko M, Kubíčková A, Kováčová J et al. Dlouhé nekódující RNA jako diagnostické biomarkery renálního karcinomu. Ces Urol 2019; 23 (Suppl. A): 64.
99. Jiang N, Pan J, Fang S et al. Liquid biopsy: Circulating exosomal long noncoding RNAs in cancer. Clin Chim Acta 2019; 495 : 331–337. doi: 10.1016/j.cca.2019.04.082.
100. Wu Y, Wang YQ, Weng WW et al. A serum-circulating long noncoding RNA signature can discriminate between patients with clear cell renal cell carcinoma and healthy controls. Oncogenesis 2016; 5: e192. doi: 10.1038/oncsis.2015.48.
101. Kohls K, Schmidt D, Holdenrieder S et al. Detection of cell-free lncRNA in serum of cancer patients. Urologe A 2015; 54(6): 819–825. doi: 10.1007/s00120-014-3655-5.
102. Zeng JH, Lu W, Liang L et al. Prognosis of clear cell renal cell carcinoma (ccRCC) based on a six-lncRNA-based risk score: an investigation based on RNA-sequencing data. J Transl Med 2019; 17(1): 281. doi: 10.1186/s12967-019-2032-y.
103. Jiang W, Guo Q, Wang CH et al. A nomogram based on 9-lncRNAs signature for improving prognostic prediction of clear cell renal cell carcinoma. Cancer Cell Int 2019; 19(1): 208. doi: 10.1186/s12935-019-0928-5.
104. Zuo SG, Wang LP, Wen YQ et al. Identification of a universal 6-lncRNA prognostic signature for three pathologic subtypes of renal cell carcinoma. J Cell Biochem 2019; 120(5): 7375–7385. doi: 10.1002/jcb.28012.
105. Wang JW, Zhang CY, He WY et al. Construction and comprehensive analysis of dysregulated long non-coding RNA-associated competing endogenous RNA network in clear cell renal cell carcinoma. J Cell Biochem 2019; 120(2): 2576–2593. doi: 10.1002/jcb.27557.
106. Qu L, Wang ZL, Chen Q et al. Prognostic value of a long non-coding RNA signature in localized clear cell renal cell carcinoma. Eur Urol 2018;74(6):756–763. doi: 10.1016/j.eururo.2018.07.032.
107. Chen BH, Wang CY, Zhang J et al. New insights into long noncoding RNAs and pseudogenes in prognosis of renal cell carcinoma. Cancer Cell Int 2018; 18 : 157. doi: 10.1186/s12935-018-0652-6.
108. Liu T, Sui J, Zhang Y et al. Comprehensive analysis of a novel lncRNA profile reveals potential prognostic biomarkers in clear cell renal cell carcinoma. Oncol Rep 2018; 40(3): 1503–1514. doi: 10.3892/or.2018.6540.
109. Gao HW, Chen XR, Shang ZQ et al. Prognostic signatures for renal cancer as identified by long non-coding and miRNA competing endogenous network analysis. Oncol Rep 2018; 40(2): 959–967. doi: 10.3892/or.2018.6476.
110. Su HC, Wang HK, Shi GH et al. Downregulation of long non-coding RNA ENSG00000241684 is associated with poor prognosis in advanced clear cell renal cell carcinoma. Eur J Surg Oncol 2018; 44(6): 840–846. doi: 10.1016/j.ejso.2018.01.013.
111. Yang K, Lu XF, Luo PC et al. Identification of six potentially long noncoding RNAs as biomarkers involved competitive endogenous RNA in clear cell renal cell carcinoma. Biomed Res Int 2018; 9303486 : 1–13. doi: 10.1155/2018/9303486.
112. Lan HH, Zeng JH, Chen G et al. Survival prediction of kidney renal papillary cell carcinoma by comprehensive LncRNA characterization. Oncotarget 2017; 8(67): 110811–110829. doi: 10.18632/oncotarget.22732.
113. Yang FY, Wang Y, Wu JG et al. Analysis of long non-coding RNA expression profiles in clear cell renal cell carcinoma. Oncol Lett 2017; 14(3): 2757–2764. doi: 10.3892/ol.2017.6563.
114. Shi D, Qu QH, Chang QM et al. A five-long non-coding RNA signature to improve prognosis prediction of clear cell renal cell carcinoma. Oncotarget 2017; 8(35): 58699–58708. doi: 10.18632/oncotarget.17506.
115. Jin P, Wang J, Liu Y. Downregulation of a novel long non-coding RNA, LOC389332, is associated with poor prognosis and tumor progression in clear cell renal cell carcinoma. Exp Ther Med 2017; 13(3): 1137–1142. doi: 10.3892/etm.2017.4080.
116. Su HC, Sun TT, Wang HK et al. Decreased TCL6 expression is associated with poor prognosis in patients with clear cell renal cell carcinoma. Oncotarget 2017; 8(4): 5789–5799. doi: 10.18632/oncotarget.11011.
117. Ni WJ, Song EL, Gong MC et al. Downregulation of lncRNA SDPR-AS is associated with poor prognosis in renal cell carcinoma. Onco Targets Ther 2017; 10 : 3039–3047. doi: 10.2147/OTT.S137641.
118. Xue S, Li QW, Che JP et al. Decreased expression of long non-coding RNA NBAT-1 is associated with poor prognosis in patients with clear cell renal cell carcinoma. Int J Clini Exp Pathol 2015; 8(4): 3765–3774.
119. Bao X, Duan JY, Yan YJ et al. Upregulation of long noncoding RNA PVT1 predicts unfavorable prognosis in patients with clear cell renal cell carcinoma. Cancer Biomark 2018; 21(1): 55–63. doi: 10.3233/CBM-170251.
120. An HX, Xu B, Wang Q et al. Up-regulation of long non-coding RNA SNHG6 predicts poor prognosis in renal cell carcinoma. Eur Rev Med Pharmacol Sci 2018; 22(24): 8624–8629. doi: 10.26355/eurrev_201812_16626.
121. Flippot R, Mouawad R, Spano JP et al. Expression of long non-coding RNA MFI2-AS1 is a strong predictor of recurrence in sporadic localized clear-cell renal cell carcinoma. Sci Rep 2017; 7(1): 8540. doi: 10.1038/s41598-017-08363-6.
122. Qi JJ, Liu YX, Lin L. High expression of long non-coding RNA ATB is associated with poor prognosis in patients with renal cell carcinoma. Eur Rev Med Pharmacol Sci 2017; 21(12): 2835–2839.
123. Xu Y, Tong YY, Zhu JY et al. An increase in long non-coding RNA PANDAR is associated with poor prognosis in clear cell renal cell carcinoma. BMC Cancer 2017; 17(1): 373. doi: 10.1186/s12885-017-3339-9.
124. Gong X, Siprashvili Z, Eminaga O et al. Novel lincRNA SLINKY is a prognostic biomarker in kidney cancer. Oncotarget 2017; 8(12): 18657–18669. doi: 10.18632/oncotarget.15703.
125. Wang PQ, Wu YX, Zhong XD et al. Prognostic significance of overexpressed long non-coding RNA TUG1 in patients with clear cell renal cell carcinoma. Eur Rev Med Pharmacol Sci 2017; 21(1): 82–86.
126. Wu Y, Tan C, Weng WW et al. Long non-coding RNA Linc00152 is a positive prognostic factor for and demonstrates malignant biological behavior in clear cell renal cell carcinoma. Am J Cancer Res 2016; 6(2): 285–299.
127. Zhang HM, Yang FQ, Chen SJ et al. Upregulation of long non-coding RNA MALAT1 correlates with tumor progression and poor prognosis in clear cell renal cell carcinoma. Tumor Biol 2015; 36(4): 2947–2955. doi: 10.1007/s13277-014-2925-6.
128. Wang L, Cai Y, Zhao X et al. Down-regulated long non-coding RNA H19 inhibits carcinogenesis of renal cell carcinoma. Neoplasma 2015; 62(3): 412–418. doi: 10.4149/neo_2015_049.
129. Yao J, Chen Y, Wang YC et al. Decreased expression of a novel lncRNA CADM1-AS1 is associated with poor prognosis in patients with clear cell renal cell carcinomas. Int J Clin Exp Pathol 2014; 7(6): 2758–2767.
130. Zhang HM, Yang FQ, Yan Y et al. High expression of long non-coding RNA SPRY4-IT1 predicts poor prognosis of clear cell renal cell carcinoma. Int J Clin Exp Pathol 2014; 7(9): 5801–5809.
131. Xing QW, Li R, Xu AM et al. Genetic variants in a long noncoding RNA related to Sunitinib Resistance predict risk and survival of patients with renal cell carcinoma. Cancer Med 2019; 8(6): 2886–2896. doi: 10.1002/cam4.2160.
132. Chen D, Zhu M, Su H et al. LINC00961 restrains cancer progression via modulating epithelial-mesenchymal transition in renal cell carcinoma. J Cell Physiol 2019; 234(5): 7257–7265. doi: 10.1002/jcp.27483.
133. He XF, Sun FG, Guo FF et al. Knockdown of long noncoding RNA FTX inhibits proliferation, migration, and invasion in renal cell carcinoma cells. Oncol Res 2017; 25(2): 157–166. doi: 10.3727/096504016X14719078133203.
Štítky
Dětská onkologie Chirurgie všeobecná Onkologie
Článek vyšel v časopiseKlinická onkologie
Nejčtenější tento týden
2020 Číslo 5- Vliv regulace pH na snížení výskytu peristomálních komplikací a bolesti
- INFOGRAFIKA: Inovace v péči o kůži – síla regulace pH v reálné praxi
- Metamizol jako analgetikum první volby: kdy, pro koho, jak a proč?
-
Všechny články tohoto čísla
- Editorial
- Současný pohled na možnosti léčby BRAF mutovaného kolorektálního karcinomu
- Dlouhé nekódující RNA a karcinom z renálních buněk
- Výzvy a riešenia v manažmente kardiotoxicity checkpoint inhibítorov
- Systémová liečba hepatocelulárneho karcinómu
- Nové trendy v neoadjuvantní léčbě lokálně pokročilého karcinomu rekta z pohledu chirurga – komentář
- Implementácia imunoterapie do liečby neuroblastómu – skúsenosti jedného centra s podávaním a manažovaním nežiaducich účinkov dinutuximabu
- Skvamocelulárny karcinóm hrubého čreva – kazuistika
- Prvý pacient s COVID-19 na Slovensku
- Extravazace (paravazace) cytostatik – aktualizované doporučení (2020) pro standardní péči v rámci České republiky ze spolupráce Sekce podpůrné léčby České onkologické společnosti ČLS JEP, České hematologické společnosti ČLS JEP, Onkologické sekce České asociace sester a Společnosti pro porty a permanentní katétry
- Entrektinib – tumor-agnostická léčba penetrující do centrálního nervového systému s prokázanou účinností i u pediatrické populace
- Aktuality z odborného tisku
- Klinická onkologie
- Archiv čísel
- Aktuální číslo
- Informace o časopisu
Nejčtenější v tomto čísle- Nové trendy v neoadjuvantní léčbě lokálně pokročilého karcinomu rekta z pohledu chirurga – komentář
- Současný pohled na možnosti léčby BRAF mutovaného kolorektálního karcinomu
- Extravazace (paravazace) cytostatik – aktualizované doporučení (2020) pro standardní péči v rámci České republiky ze spolupráce Sekce podpůrné léčby České onkologické společnosti ČLS JEP, České hematologické společnosti ČLS JEP, Onkologické sekce České asociace sester a Společnosti pro porty a permanentní katétry
- Systémová liečba hepatocelulárneho karcinómu
Kurzy
Zvyšte si kvalifikaci online z pohodlí domova
Revma Focus: Spondyloartritidy
nový kurz
Autoři: prof. MUDr. Vladimír Palička, CSc., Dr.h.c., doc. MUDr. Václav Vyskočil, Ph.D., MUDr. Petr Kasalický, CSc., MUDr. Jan Rosa, Ing. Pavel Havlík, Ing. Jan Adam, Hana Hejnová, DiS., Jana Křenková
Autoři: MUDr. Irena Krčmová, CSc.
Všechny kurzyPřihlášení#ADS_BOTTOM_SCRIPTS#Zapomenuté hesloZadejte e-mailovou adresu, se kterou jste vytvářel(a) účet, budou Vám na ni zaslány informace k nastavení nového hesla.
- Vzdělávání