-
Medical journals
- Career
Degradace proteinů ubikvitin‑proteazomovou dráhou
: J. Matějíková 1; L. Kubiczková 1,2; L. Sedlaříková 1; A. Potáčová 1; R. Hájek 1,2; S. Ševčíková 1,2
: Babákova myelomová skupina, Ústav patologické fyziologie, LF MU, Brno2 Oddělení klinické hematologie, FN Brno 1
: Klin Onkol 2013; 26(4): 251-256
: Reviews
Všechny intracelulární a některé extracelulární proteiny jsou kontinuálně degradovány a nahrazovány syntézou nových proteinů. Oba tyto děje musejí zůstat ve vzájemné rovnováze, neboť její narušení by mohlo vést ke vzniku závažných onemocnění. Buňky obsahují mnoho proteolytických systémů, které zajišťují vysoce specifickou a kontrolovanou degradaci proteinů. Jedním z nich je proteazom, složitý molekulární stroj sloužící k degradaci proteinů konjugovaných s ubikvitinem. Od první izolace proteazomu v roce 1968 bylo objeveno mnoho detailů o fungování tohoto systému. V roce 2004 byla za tyto objevy dokonce udělena Nobelova cena za chemii. V naší přehledové práci jsme se zaměřili na shrnutí dosavadních poznatků o mechanizmech vysoce selektivní degradace proteinů ubikvitin‑proteazomovou dráhou. Jednotlivé kapitoly tohoto článku pojednávají o struktuře a funkci systému ubikvitin‑proteazom, mechanizmech regulace degradace proteinů a biologických účincích inhibitorů proteazomu.
Klíčová slova:
proteazom – ubiquitin – NF-kappa B – proteazomové inhibitory – degradace proteinůÚvod
Všechny intracelulární a některé extracelulární proteiny jsou kontinuálně degradovány a nahrazovány syntézou nových proteinů. Jednotlivé proteiny v jádře a cytosolu, ale i v endoplazmatickém retikulu a mitochondriích jsou degradovány různě rychle – od několika minut pro některé regulační proteiny až do dnů a týdnů pro proteiny, jako jsou aktin a myosin [1,2]. Buňky obsahují mnoho proteolytických systémů, které provádějí degradaci tak, aby byla vysoce specifická a kontrolovaná. Ve zdravé buňce musí existovat rovnováha mezi degradací a syntézou proteinů. Poruchy v těchto systémech mohou vést ke hromadění nefunkčních nebo poškozených proteinů, což ve svém důsledku vede ke vzniku mnoha onemocnění. Většina intracelulárních proteinů je degradována ubikvitin‑proteazomovou dráhou [3]. A právě tímto systémem se práce zabývá.
Systém ubikvitin‑proteazom
Proteazom je složitý molekulární stroj sloužící k degradaci proteinů konjugovaných s ubikvitinem. Skládá se ze základny a na ni navázaných regulačních jednotek, které dále upravují proteazomovou aktivitu a specifitu [4]. Proteazom byl objeven a poprvé izolován roku 1968 a pro svůj tvar dutého válce byl původně nazván cylindrin [5]. Vyskytuje se ve velkém množství v cytosolu a v jádře s odlišnou distribucí u různých typů buněk a v různých fázích buněčného cyklu [6]. Zaujímá 0,5 % až 1 % všech buněčných proteinů [7]. Vysoce proliferující a transformované buněčné linie vykazují vyšší hladiny proteazomu a jeho aktivity než klidové a netransformované buňky [8].
Z evolučního hlediska se jedná o vysoce konzervativní strukturu. Existují dva základní evoluční typy proteazomových jednotek. Ancestrální forma popsána u archeí obsahuje stejné uspořádání podjednotek jako ta savčí, ale sestává pouze z jednoho typu α podjednotek a jednoho typu β podjednotek [4].
V roce 2004 byla udělena Nobelova cena za objev ubikvitinem zprostředkované degradace proteinů. V historickém kontextu se jednalo o zcela „nezajímavou“ oblast výzkumu. Po odhalení trojrozměrné struktury DNA se zájem soustředil spíše na syntézu proteinů než na jejich degradaci. Ačkoliv se již na konci 60. let vědělo, že proteiny jsou odbourávány vysoce selektivním nonlysozomálním způsobem, kovalentní vazba ubikvitinu na proteinové substráty byla rozpoznána až na přelomu let 80. Hlavním předpokladem pro objev byla skutečnost, že k dosažení selektivity byla zapotřebí energie v podobě ATP [9].
V rámci zachování homeostázy buněčného metabolizmu využívá buňka dvou hlavních cest recyklace proteinů. Každou cestu doprovází specifické proteázy. Kromě selektivní ATP ‑ dependentní degradace zprostředkované ubikvitin‑proteazomovým systémem (UPS) jsou proteiny odbourávány prostřednictvím membránového kompartmentu lysozomu. Lysozomální degradace probíhá mechanizmem autofagie za účasti hydrolytických enzymů katepsinů v prostředí s nízkým pH. Jejím substrátem jsou extracelulární proteiny s dlouhým biologickým poločasem či struktury větších rozměrů, jakými jsou buněčné organely [10]. Substrátem pro proteazomovou degradaci jsou oproti tomu intracelulární proteiny s krátkou životností, což jsou i špatně sbalené, poškozené či dále nepotřebné proteiny [11].
UPS je svou činností zapojen do regulace buněčného cyklu, kontroly nově syntetizovaných proteinů, regulace transkripčních faktorů, genové exprese, imunitní odpovědi i patologických procesů, jakými jsou nádorová a neurodegenerativní onemocnění, zánětlivé procesy, a v neposlední řadě hraje roli v procesu stárnutí [12].
Struktura proteazomu
Má‑li protein podstoupit degradaci, je ve většině případů označen vazbou několika ubikvitinových molekul. Polyubikvitinovaný protein je následně transportován k multikatalytickému enzymovému komplexu – proteazomu, kde dochází k vlastnímu štěpení na krátké peptidové sekvence.
Běžným proteazomovým systémem savčích buněk je 26S proteazom o molekulové hmotnosti 2,5 MDa a s 31 typy podjednotek [13]. Skládá se z hlavní části 20S proteazomu, dutého útvaru tvaru válce poskytujícího uzavřený prostor pro vlastní proteolýzu a na něj navázaných jedné či dvou 19S regulačních jednotek (PA700), které obsahují několik aktivních a ubikvitin‑vazebných míst [14].
20S proteazom se skládá ze čtyř homologních prstencových struktur. Každý prstenec je tvořen buď sedmi podjednotkami α, nebo sedmi podjednotkami β. Prstence jsou uspořádány v pořadí αββα nad sebou (obr. 1) [15].
1. Struktura proteazomu. Čtyři prstence po 7 podjednotkách v pořadí αββα. 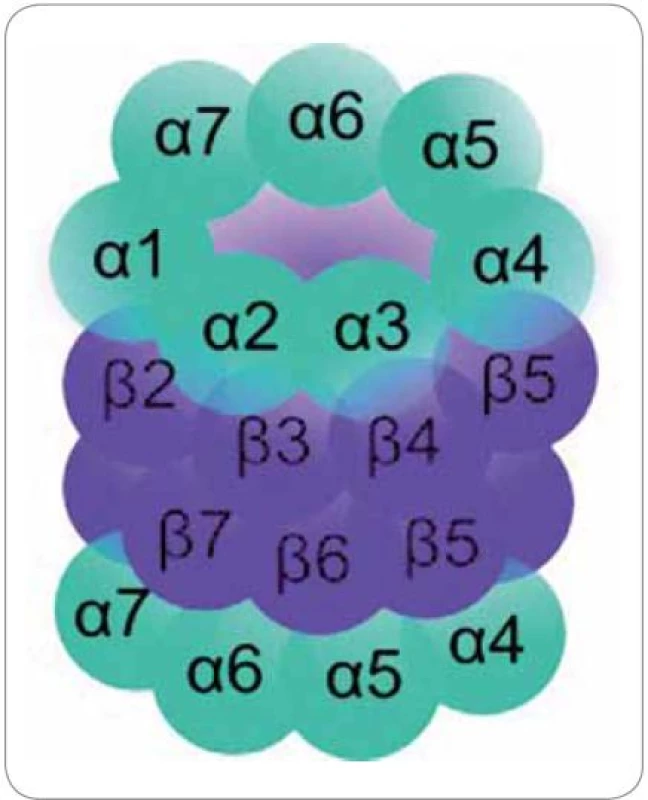
Vnější α prstence regulují přístup substrátu do vnitřního proteolytického centra. Jestliže α podjednotky rozpoznají substrát, dojde ke konformační změně, při které dochází k odstranění blokády vstupní brány N ‑ konci aminokyselinových zbytků podjednotek α ‑ 2, α ‑ 3 a α‑4 [16]. Vnitřní β prstence jsou zodpovědné za proteolytickou degradaci substrátu. Konkrétně proteolytickou aktivitu vykazují podjednotky β1, β2 a β5. Tyto podjednotky se nacházejí v proteolytickém centru, ve středu dutiny proteazomu. Každá z nich obsahuje aktivní místo na svém N ‑ konci, ve kterém se nachází zbytek aminokyseliny threoninu (Thr1). Podjednotka β1 vykazuje proteolytickou aktivitu podobnou kaspázám, které štěpí polypeptidový řetězec v místě za kyselou aminokyselinou v sekvenci. Podjednotka β2 vykazuje aktivitu podobnou trypsinu, který štěpí po přítomnosti bazických aminokyselin, a podjednotka β5 má aktivitu podobnou chymotrypsinu, který štěpí po hydrofobních aminokyselinách [17]. Oligopeptidovým produktem proteazomové degradace je minimálně 2 a maximálně 35 aminokyselin dlouhý řetězec s průměrnou délkou 8 až 12 aminokyselin [18].
Pro stabilizaci trojrozměrné struktury aktivního místa jsou důležité serinové zbytky v lokusech Ser129/ 166/ 169 a pro funkčnost proteolýzy zbytky aminokyselin Asp17 a Lys33, které obklopují Thr1. Z hlediska proteolýzy nejsou všechny aktivní β podjednotky stejně důležité, ale platí hierarchie v pořadí β5 > β2 > β1 [17].
Aby se předešlo nekontrolované degradaci buněčného materiálu, existuje v UPS několik na sobě nezávislých regulačních mechanizmů. Jedním z vysoce efektivních je přítomnost regulačních molekul. 19S regulátor je proteazomovým aktivátorem o molekulové hmotnosti 700 kDa (PA700) [19]. Je složen z 19 samostatných proteinů, přičemž 10 proteinů tvoří základnu, která se váže k α prstencům 20S proteazomu, a 9 proteinů vytváří strukturu podobnou víku, kam se váže polyubikvitinový řetězec. PA700 umožňuje přístup vyšší koncentrace označeného substrátu do proteolytického prostoru [20].
Podjednotky Rpn10 a Rpn13 19S regulátoru slouží jako ubikvitinové receptory. Rpt2, Rpt3 a Rpt5 podjednotky jsou zapojeny do otevírání přístupu k 20S proteazomu. Podjednotka Rpn11 obsahuje Zn2+ - dependentní proteolytické centrum, které odbourává polyubikvitinový řetězec a uvolňuje jej do svého okolí [20].
Mechanizmus degradace ubikvitinem
Ubikvitin (Ub) je malý protein složený ze 76 aminokyselin s celkovou molekulovou hmotností 8,5 kDa. Ubikvitinem označený protein je degradován v proteazomu a jeho aminokyseliny jsou využity k seskládání nových proteinů [9].
Proces ubikvitinace sestává ze tří kroků a vyžaduje přítomnost tří různých tříd enzymů (E1 až E3). Počátečním krokem je dvoustupňová ATP ‑ dependentní aktivace ubikvitinu enzymem E1. Nejdříve je vytvořen adenylovaný meziprodukt, který je atakován cysteinovým zbytkem aktivního centra E1 enzymu, na který se ubikvitin naváže thioesterovou vazbou. Ubikvitin‑aktivující enzym E1 následně ubikvitin přenáší k ubikvitin‑konjugujícímu enzymu E2, na jehož cysteinový zbytek se ubikvitin naváže svým C ‑ koncovým glycinem 76 transthioesterifikační reakcí. Posledním krokem je navázání ubikvitinu na substrát pomocí enzymu ubikvitin‑ligázy E3. Vzniká izopeptidová vazba mezi lyzinem cíleného proteinu a C ‑ koncovým glycinem ubikvitinu [... - CO ‑ NH2ε ‑ ...]. Substrátová ubikvitinace může probíhat dvěma různými způsoby. Za první způsob nese zodpovědnost RING doména E3 enzymů a dochází k přímému přenosu ubikvitinu na substrát. V druhém případě dochází k vytvoření E3 - ubikvitin intermediátu, jehož vznik je katalyzován HECT doménou E3 enzymů (obr. 2) [21].
2. Mechanizmus ubikvitinace cílového proteinu (CP) enzymy E1 (ubikvitin-aktivační enzym), E2 (ubikvitin-konjugační enzym) a E3 (ubikvitin-ligáza). 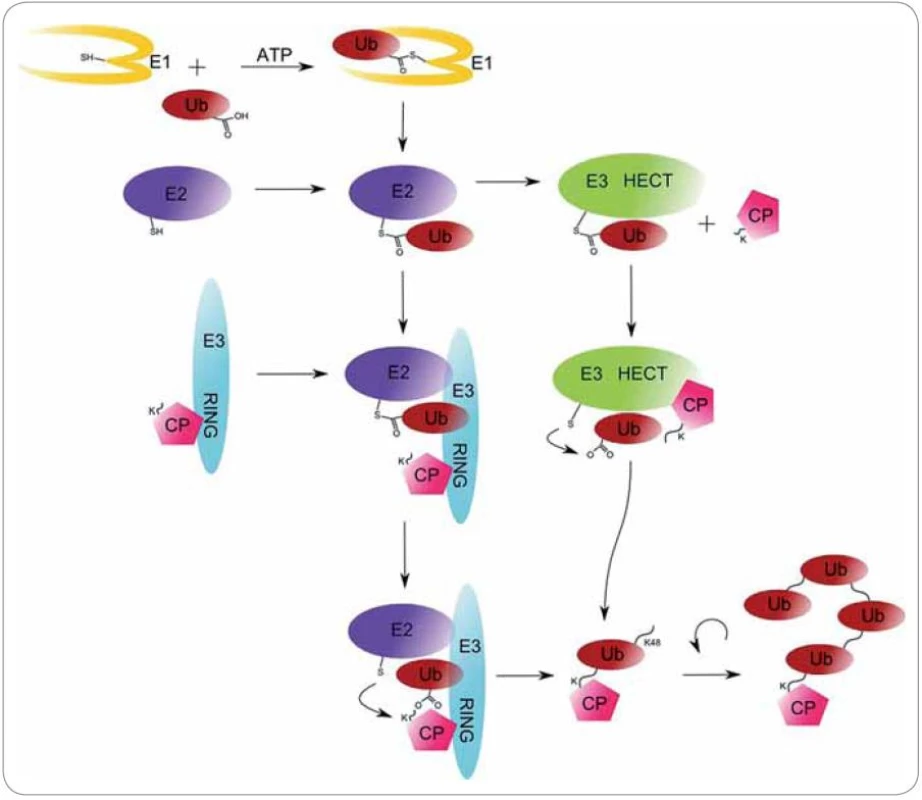
Předpokládá se, že existuje několik málo E1 enzymů, několik desítek E2 enzymů a zhruba 600 různých E3 enzymů zakódovaných v lidském genomu [22]. Substrátová specifita je dána právě velkým počtem ubikvitinových ligáz (E3), přičemž každá z nich je specifická pro limitní počet substrátových proteinů [23].
Aby mohly být substráty pro degradaci rozpoznány proteazomem, musejí být v řetězci navázány alespoň čtyři molekuly ubikvitinu [24]. Způsob vazby ubikvitinů není jednotný a určuje, zda daný protein bude opravdu degradován, nebo dojde k alternativním buněčným procesům, jakými mohou být aktivace enzymů, transkripce genů či transport membránových proteinů [25]. V ubikvitinové sekvenci aminokyselin se nachází sedm lyzinových zbytků, mezi kterými může docházet k vazbě [26].Nejčastější spojení ubikvitinů se uskutečňuje přes lyzin 48 (K48 řetězce) a vede k destrukci označeného proteinu [27].Nekanonické spojení řetězců vedoucí ke stejnému cíli bylo in vitro prokázáno také mezi lyziny 63 [28]. Katalýzy polyubikvitinace se kromě E4 enzymů účastní i poslední rodina E3 U ‑ box proteiny [29].
Polyubikvitinovaný protein je rozpoznáván ubikvitinovými receptory proteazomu jako substrát proteolýzy. Rpt2 podjednotka 19S regulátoru reguluje vstup substrátu i uvolňování produktu do/ ze základny proteazomu. Ubikvitinové molekuly jsou uvolněny do cytozolu deubikvitinázou Rpn11 ve formě polyubikvitinového řetězce [30]. Zde jsou řetězce degradovány na jednotlivé molekuly dalšími deubikvitinačními enzymy (DUBs) [31].
Regulační mechanizmus UPS v sobě zahrnuje přítomnost antagonisticky působících, ubikvitinu podobných proteinů (UBLs), mezi které patří SUMO modifikující protein p53 a substrát specifických DUBs, mezi které patří CYLD odstraňující ubikvitin ze signálních molekul NFκB dráhy a USP9X regulující ubikvitinaci Mcl ‑ 1 [32 – 34].
Vlastní degradaci proteinu předcházejí procesy jako rozbalení do primární struktury a přemístění do proteolytického centra, které jsou zprostředkované proteázami podobnými chaperonům za spotřeby energie získané hydrolýzou ATP. Tyto ATPázy (podjednotky Rpt 1 – 6) jsou součástí 19S regulační partikule [35]. Štěpení proteinu probíhá tak dlouho, dokud nejsou vytvořeny všechny oligopeptidové produkty schopné difundovat ven z proteazomu.
V buňkách hematopoetické řady je hlavním typem proteazomu jeho inducibilní izoforma – imunoproteazom, jehož výskyt koreluje s hladinou cytokinů. Je charakterizován nahrazením proteolyticky aktivních podjednotek β1, β2 a β5 jejich ekvivalenty β1i (LMP2), β2i (MELC1) a β5i (LMP7). Zároveň je 20Si základna spojena s odlišnou regulační jednotkou – 11S regulátorem. Imunoproteazom štěpí proteiny na 8 – 10 aminokyselin dlouhé oligopeptidy, které jsou optimální pro prezentaci hlavním histokompatibilním komplexem I na povrchu buňky [36].
Karcinogenní substráty proteazomové degradace a jejich funkce
Roli proteazomu a jeho inhibitorů v procesu karcinogeneze lze nejlépe demonstrovat identifikací reprezentativních substrátů proteolýzy. Substráty související s karcinogenezí lze obecně rozdělit na strukturně nefunkční proteiny a proteiny s krátkým biologickým poločasem.
Ačkoliv primární sekvence aminokyselin každého nově syntetizovaného proteinu obsahuje veškerou informaci potřebnou pro zformování jeho terciární struktury, je ve skutečnosti 30 % proteinů sbaleno chybně, bez možnosti správného znovusbalení [37]. Aby buňka předešla akumulaci nefunkčních proteinů uvězněných v nízkoenergetickém stavu a jejich aminokyseliny mohly být dále recyklovány, využívá systém chaperonů, který tyto struktury rozpoznává, a systém transmembránových proteinů, který je dále transportuje z endoplazmatického retikula, kde probíhá jejich sbalování, do cytozolu, kde se nachází proteazom. Tento proces se nazývá degradace asociovaná s endoplazmatickým retikulem (ERAD) [38]. Také cytozolové proteiny s narušenou strukturou nebo náchylné k agregaci jsou vychytávány k proteazomové degradaci pomocí dvou vysoce konzervovaných kompartmentů JUNQ a IPOD [39]. Kromě a principio špatně sbalených proteinů vstupuje do proteazomové degradace převážná většina proteinů s dlouhým biologickým poločasem poškozených reaktivními radikály kyslíku, intracelulární denaturací či jinak strukturně modifikovaných a neschopných plnit svou funkci. Hlavním substrátem UPS však zůstávají proteiny s krátkým biologickým poločasem. Zpravidla na svém N ‑ konci obsahují hydrofobní zbytky aminokyselin. Mezi takové substráty řízené proteolýzy patří regulační molekuly buněčného cyklu, transkripční faktory, onkogenní produkty, nádorové supresory, apoptotické faktory a antigeny (tab. 1) [40].
1. Substráty proteazomu důležité pro nádorové buňky. 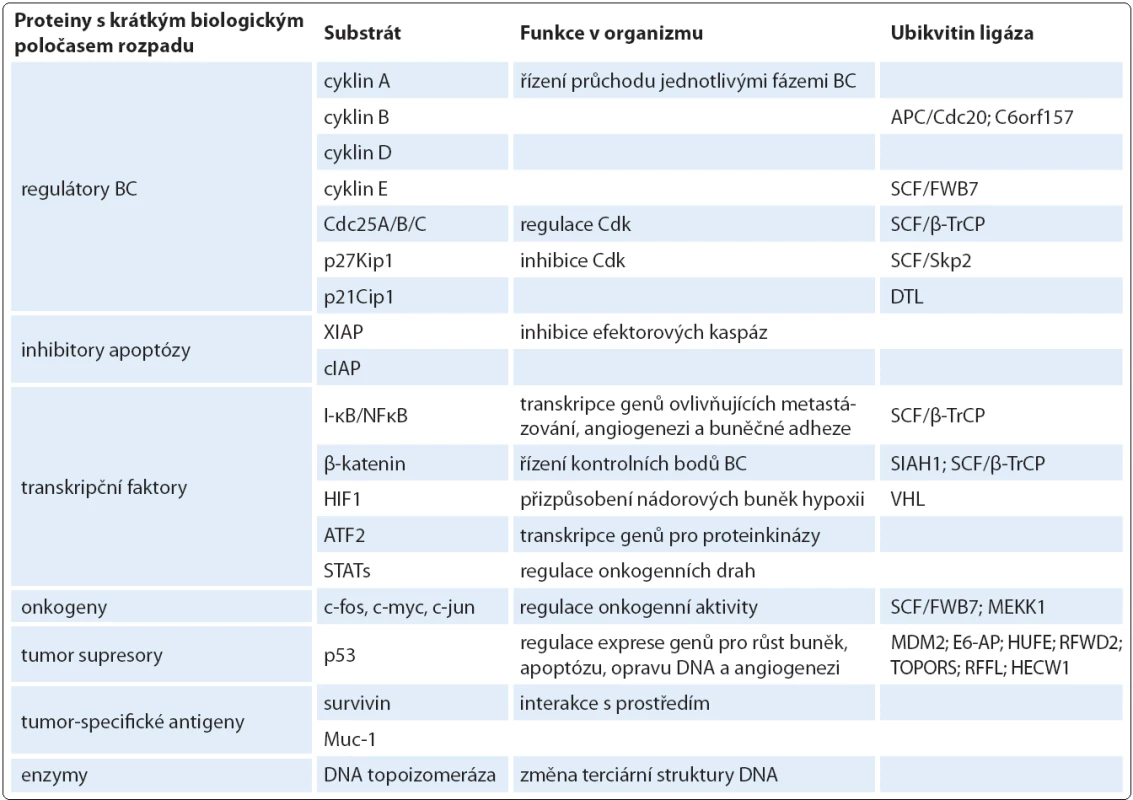
Mezi nejvýznamnější molekuly regulované proteazomem patří členové signální dráhy NFκB (obr. 3). Jaderný faktor NFκB je souhrnný název pro skupinu rychle působících primárních transkripčních faktorů patřících do rodiny Rel (reticuloendotheliosis), které ovlivňují transkripci genů pro růstové faktory, cytokiny, chemokiny, modulátory angiogeneze, adhezivní molekuly a antiapoptotické faktory [41]. Poprvé byl NFκB popsán roku 1986 jako B‑lymfocytární jaderný faktor potřebný pro transkripci κ lehkých řetězců Ig [42]. Od té doby mu byly připsány desítky dalších funkcí v rámci většiny typů buněk lidského organizmu.
3. Role proteazomu v kanonické a nekanonické cestě aktivace transkripčního faktoru NFκB. 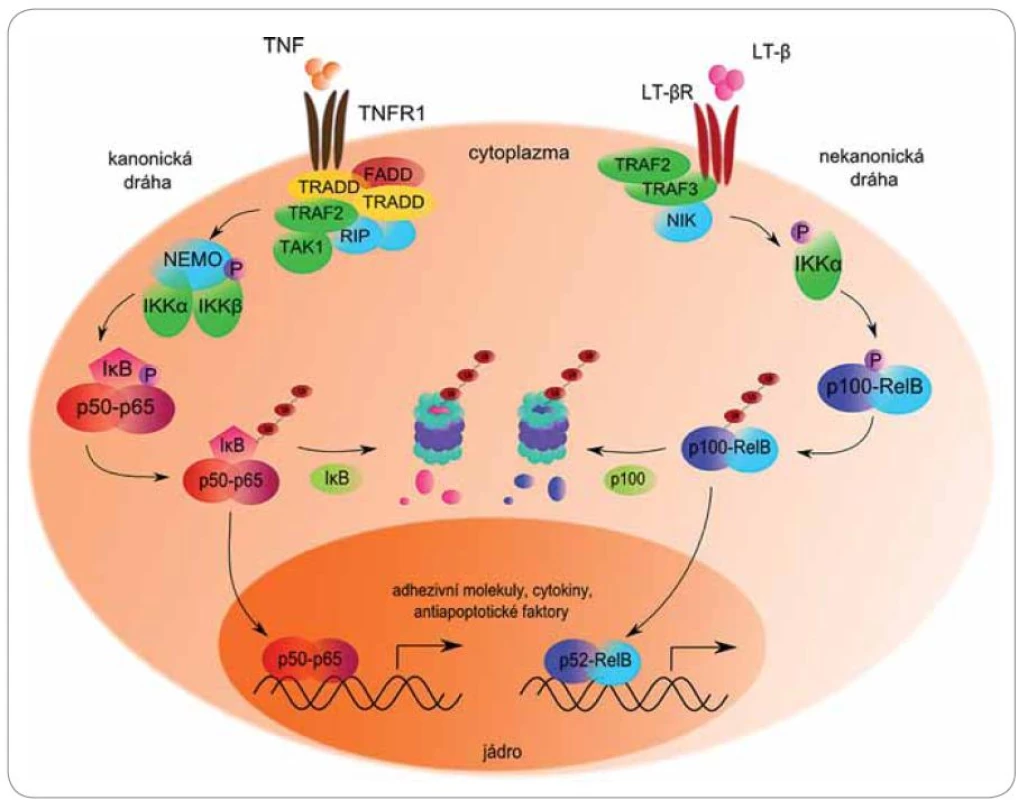
NFκB existuje v cytoplazmě v podobě inaktivních dimerických komplexů navázaných na inhibitor κB (IκB). Tento inhibitor maskuje jaderný lokalizační signál, a tím brání translokaci NFκB do jádra [43]. Aktivaci dimeru předchází fosforylace inhibitoru specifickou IęB kinázou (IKK), která se nachází v cytoplazmě a vykazuje serin‑proteinkinázovou aktivitu. IKK se skládá ze tří podjednotek – heterodimeru IKKα/ IKKβ s katalytickou aktivitou a regulační podjednotky IKKγ (NEMO) [44].
Inaktivní komplex NFκB ‑ IκB je stimulován celou řadou podnětů, jakými jsou prozánětlivé cytokiny TNFα a IL‑1, mitogeny, růstové faktory, stresory, lipopolysacharidy či volné radikály. Různé stimuly mohou vyvolat aktivaci NFκB dvěma odlišnými způsoby.
Klasickou signální cestou je IκBα fosforylován na serinu 32 a 36 podjednotkou βIKK. Fosforylovaný IκBα je rozeznáván ubikvitin‑ligázovým komplexem SCF/ β ‑ TrCP, polyubikvitinován a degradován proteazomem [45]. Odstraněním inhibitoru je NFκB schopen přenosu do jádra a následné indukce exprese více než 50 různých genů, mimo jiné pro IL‑6, IL‑1, IL‑2, TNFα, COX‑2, ICAM‑1, VCAM‑1, XIAP, cIAP a dalších molekul významných pro onkogenezi [41]. Nejznámějším zástupcem aktivovaným kanonickou dráhou je heterodimer proteinů p50/ p65, známý jako specifický NFκB.
Nekanonická alternativní dráha je založena na odlišném inhibitoru, kterým je prekurzorový protein p100. Aktivace je zahájena fosforylací NFκB ‑ inducibilní kinázy (NIK) podjednotkou α IKK homodimeru. Aktivovaná NIK dále fosforyluje protein p100 (100 kDa) na C ‑ konci, což vede k jeho zkrácení proteolytickým štěpením v proteazomu na p52 (52 kDa). Výsledkem je transkripčně aktivní dimer p52/ RelB [46,47]. Deregulace NFκB nastává při zánětlivých, autoimunitních, virových a především nádorových onemocněních [48].
Biologické účinky inhibitorů proteazomu
Vzhledem k důležitosti proteazomu v mnoha aspektech buněčného metabolizmu jsou účinky jeho inhibitorů na živé buňky komplexními procesy založenými na akumulaci, inhibici, inaktivaci, snížení anebo zvýšení hladiny jednotlivých proteinů, které spolu vzájemně interagují v rámci signálních drah.
Primárním důsledkem inhibice proteazomu je úplný pokles odbourávání proteinů, čímž nastává jejich hromadění. Velká množství proteinů konjugovaných s ubikvitinem 40 se akumulují v podobě agresomů v perinukleární oblasti buňky, kde jsou lokalizovány nefunkční proteazomy. Formování agresomů vyžaduje histonovou deacetylázu 6 (HDAC6) k vytvoření pevného spojení mezi mikrotubuly a ubikvitinovanými proteiny. Formace proteinů v agresomy nakonec vyvolá autofagické odbavení v podobě lyzozomální degradace [49]. Nahromadění nesbalených proteinů také vyvolává zvýšení hladiny chaperonů a stresových proteinů ER, které buňce zprostředkují ochranu před toxickými podmínkami [50].
I přes prvotní obrannou linii dochází po dlouhodobém vystavení působení inhibitoru k programované smrti téměř všech typů buněk. Existuje několik možných vysvětlení závislosti selektivity na stupni proliferace buňky. V prvé řadě jsou vysoce proliferující nádorové buňky odlišné od ostatních buněk organizmu tím, že mají narušené kontrolní body, a tedy jsou mnohem více závislé na proteazomem mediované degradaci pozitivních i negativních regulátorů buněčného cyklu. Za druhé, je pravděpodobné, že dysregulace buněčného cyklu tyto buňky činí citlivější k proapoptickým faktorům. A konečně, je možné, že transkripční faktory, které regulují progresi buněčného cyklu, jsou také svým dílem zodpovědné za inhibitorem vyvolanou buněčnou smrt [51].
Inhibitory proteazomu se v současnosti používají i v klinické praxi. V léčbě mnohočetného myelomu, krevního nádorového onemocnění, došlo k výraznému pokroku v přežívání pacientů právě díky používání těchto inhibitorů [52].
Závěr
Regulované odbourávání proteinů u eukaryot pomocí 26S proteazomu má zásadní význam pro degradaci regulátorů buněčného cyklu, transkripčních faktorů, onkogenních produktů a nádorových supresorů, což je nezbytně nutné pro řádnou kontrolu a řízení buněčných procesů. Zároveň slouží k odbourání poškozených bílkovin z cytozolu a endoplazmatického retikula. Protože aktivita proteazomu v nádorových buňkách je obecně zvýšena, je také klíčovým cílem pro vývoj protinádorových léčiv. Některá jsou již užívána v klinické praxi při léčbě mnohočetného myelomu.
Tato práce byla podpořena výzkumnými projekty Ministerstva školství, mládeže a tělovýchovy MSM0021622434, Grantové agentury ČR GAP304/10/1395 a Interní grantové agentury Ministerstva zdravotnictví NT12130, NT11154, NT13190 a NT14575.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do bi omedicínských časopisů.
RNDr. Sabina Ševčíková, Ph.D.
Babákova myelomová skupina
Ústav patologické fyziologie
LF MU, Brno
Kamenice 5, A3
625 00 Brno
e-mail: sevcik@med.muni.cz
Obdrženo: 16. 1. 2013
Přijato: 31. 3. 2013
Sources
1. Lecker SH, Goldberg AL, Mitch WE. Protein degradation by the ubiquitin‑proteasome pathway in normal and disease states. J Am Soc Nephrol 2006; 17(7): 1807 – 1819.
2. Mitch WE, Goldberg AL. Mechanisms of muscle wasting. The role of the ubiquitin‑proteasome pathway. N Engl J Med 1996; 335(25): 1897 – 1905.
3. Rock KL, Gramm C, Rothstein L et al. Inhibitors of the proteasome block the degradation of most cell proteins and the generation of peptides presented on MHC class I molecules. Cell 1994; 78(5): 761 – 771.
4. Löwe J, Stock D, Jap B et al. Crystal structure of the 20S proteasome from the archaeon T. acidophilum at 3.4 A resolution. Science 1995; 268(5210): 533 – 539.
5. Harris JR. The isolation and purification of a macromolecular protein component from the human erythrocyte ghost. Biochim Biophys Acta 1969; 188(1): 31 – 42.
6. Wójcik C, DeMartino GN. Intracellular localization of proteasomes. Int J Biochem Cell Biol 2003; 35(5): 579 – 589.
7. Peters JM. Proteasomes: protein degradation machines of the cell. Trends Biochem Sci 1994; 19(9): 377 – 382.
8. Kumatori A, Tanaka K, Inamura N et al. Abnormally high expression of proteasomes in human leukemic cells. Proc Natl Acad Sci U S A 1990; 87(18): 7071 – 7075.
9. Hershko A. The ubiquitin system for protein degradation and some of its roles in the control of the cell ‑ division cycle (Nobel lecture). Angew Chem Int Ed Engl 2005; 44(37): 5932 – 5943.
10. Todde V, Veenhuis M, van der Klei IJ. Autophagy: principles and significance in health and disease. Biochim Biophys Acta 2009; 1792(1): 3 – 13.
11. Lee DH, Goldberg AL. Proteasome inhibitors: valuable new tools for cell biologists. Trends Cell Biol 1998; 8(10): 397 – 403.
12. Jung T, Catalgol B, Grune T. The proteasomal system. Mol Aspects Med 2009; 30(4): 191 – 296.
13. Voges D, Zwickl P, Baumeister W. The 26S proteasome: a molecular machine designed for controlled proteolysis. Annu Rev Biochem 1999; 68 : 1015 – 1068.
14. Bedford L, Paine S, Sheppard PW et al. Assembly, structure, and function of the 26S proteasome. Trends Cell Biol 2010; 20(7): 391 – 401.
15. Unno M, Mizushima T, Morimoto Y et al. The structure of the mammalian 20S proteasome at 2.75 A resolution. Structure 2002; 10(5): 609 – 618.
16. Groll M, Bajorek M, Köhler A et al. A gated channel into the proteasome core particle. Nat Struct Biol 2000; 7(11): 1062 – 1067.
17. Jäger S, Groll M, Huber R et al. Proteasome beta‑type subunits: unequal roles of propeptides in core particle maturation and a hierarchy of active site function. J Mol Biol 1999; 291(4): 997 – 1013.
18. Luciani F, Keşmir C, Mishto M et al. A mathematical model of protein degradation by the proteasome. Biophys J 2005; 88(4): 2422 – 2432.
19. DeMartino GN, Moomaw CR, Zagnitko OP et al. PA700, an ATP ‑ dependent activator of the 20 S proteasome, is an ATPase containing multiple members of a nucleotide‑binding protein family. J Biol Chem 1994; 269(33): 20878 – 20884.
20. Sharon M, Taverner T, Ambroggio XI et al. Structural organization of the 19S proteasome lid: insights from MS of intact complexes. PLoS Biol 2006; 4(8): e267.
21. Hochstrasser M. Lingering mysteries of ubiquitin‑chain assembly. Cell 2006; 124(1): 27 – 34.
22. Li W, Bengtson MH, Ulbrich A et al. Genome ‑ wide and functional annotation of human E3 ubiquitin ligases identifies MULAN, a mitochondrial E3 that regulates the organelle‘s dynamics and signaling. PLoS One 2008; 3(1): e1487.
23. Metzger MB, Hristova VA, Weissman AM. HECT and RING finger families of E3 ubiquitin ligases at a glance. J Cel Sci 2012; 125(Pt 3): 531 – 537.
24. Thrower JS, Hoffman L, Rechsteiner M et al. Recognition of the polyubiquitin proteolytic signal. EMBO J 2000; 19(1): 94 – 102.
25. Pickart CM, Fushman D. Polyubiquitin chains: polymeric protein signals. Curr Opin Chem Biol 2004; 8(6): 610 – 616.
26. Peng J, Schwartz D, Elias JE et al. A proteomics approach to understanding protein ubiquitination. Nat Biotechnol 2003; 21(8): 921 – 926.
27. Petroski MD, Deshaies RJ. Mechanism of lysine 48‑linked ubiquitin‑chain synthesis by the cullin‑RING ubiquitin‑ligase complex SCF ‑ Cdc34. Cell 2005; 123(6): 1107 – 1120.
28. Saeki Y, Kudo T, Sone T et al. Lysine 63‑linked polyubiquitin chain may serve as a targeting signal for the 26S proteasome. EMBO J 2009; 28(4): 359 – 371.
29. Hatakeyama S, Yada M, Matsumoto M et al. U box proteins as a new family of ubiquitin‑protein ligases. J Biol Chem 2001; 276(35): 33111 – 33120.
30. Verma R, Aravind L, Oania R et al. Role of Rpn11 metalloprotease in deubiquitination and degradation by the 26S proteasome. Science 2002; 298(5593): 611 – 615.
31. Reyes ‑ Turcu FE, Ventii KH, Wilkinson KD. Regulation and cellular roles of ubiquitin‑specific deubiquitinating enzymes. Annu Rev Biochem 2009; 78 : 363 – 397.
32. Rodriguez M, Desterro J, Lain S et al. SUMO ‑ 1 modification activates the transcriptional response of p53. EMBO J 1999; 18(22): 6455 – 6461.
33. Sowa ME, Bennett EJ, Gygi SP et al. Defining the human deubiquitinating enzyme interaction landscape. Cell 2009; 138(2): 389 – 403.
34. Schwickart M, Juany X, Lill JR et al. Deubiquitinase USP9X stabilizes MCL1 and promotes tumour cell survival. Nature 2010; 463(7277): 103 – 107.
35. Striebel F, Kress W, Weber ‑ Ban E. Controlled destruction: AAA+ ATPases in protein degradation from bacteria to eukaryotes. Curr Opin Struct Biol 2009; 19(2): 209 – 217.
36. Kloetzel PM. The proteasome and MHC class I antigen processing. Biochim Biophys Acta 2004; 1695(1 – 3): 225 – 233.
37. Kaufman RJ. Orchestrating the unfolded protein response in health and disease. J Clin Incest 2002; 110(10): 1389 – 1398.
38. Brodsky JL. The protective and destructive roles played by molecular chaperones during ERAD (endoplasmic ‑ reticulum‑associated degradation). Biochem J 2007; 404(3): 353 – 363.
39. Bagola K, Sommer T. Protein quality control: on IPODs and other JUNQ. Curr Biol 2008; 18(21): R1019 – R1021.
40. Kropff M, Bisping G, Wenning D et al. Proteasome inhibition in multiple myeloma. Eur J Cancer 2006; 42(11): 1623 – 1639.
41. Pahl HL. Activators and target genes of Rel/ NF ‑ kappaB transcription factors. Oncogene 1999; 18(49): 6853 – 6866.
42. Sen R, Baltimore D. Inducibility of kappa immunoglobulin enhancer‑binding protein Nf ‑ kappa B by a posttranslational mechanism. Cell 1986; 47(6): 921 – 928.
43. Régnier CH, Song HY, Gao X et al. Identification and characterization of an IkappaB kinase. Cell 1997; 90(2): 373 – 383.
44. Makris C, Roberts JL, Karin M. The carboxyl‑terminal region of IkappaB kinase gamma (IKKgamma) is required for full IKK activation. Mol Cell Biol 2002; 22(18): 6573 – 6581.
45. Kanarek N, Ben ‑ Neriah Y. Regulation of NF ‑ κB by ubiquitination and degradation of the IκBs. Immunol Rev 2012; 246(1): 77 – 94.
46. Dejardin E. The alternative NF ‑ kappaB pathway from biochemistry to biology: pitfalls and promises for future drug development. Biochem Pharmacol 2006; 72(9): 1161 – 1179.
47. Annunziata CM, Davis RE, Demchenko Y et al. Frequent engagement of the classical and alternative NF ‑ kappaB pathways by diverse genetic abnormalities in multiple myeloma. Cancer Cell 2007; 12(2): 115 – 130.
48. Luqman S, Pezzuto JM. NFκB: a promising target for natural products in cancer chemoprevention. Phytother Res 2010; 24(7): 949 – 963.
49. Hideshima T, Bradner JE, Wong J et al. Small‑molecule inhibition of proteasome and aggresome function induces synergistic antitumor activity in multiple myeloma. Proc Natl Acad Sci U S A 2005; 102(24): 8567 – 8572.
50. Kaufman RJ. Orchestrating the unfolded protein response in health and disease. J Clin Incest 2002; 110(10): 1389 – 1398.
51. McConkey DJ, Zhu K. Mechanisms of proteasome inhibitor action and resistance in cancer. Drug Resist Updat 2008; 11(4 – 5): 164 – 179.
52. Kubiczková L, Matějíková J, Sedlaříková L et al. Inhibitory proteazomu v léčbě mnohočetného myelomu. Klin Okol 2013; 26(1): 11 – 18.
Labels
Paediatric clinical oncology Surgery Clinical oncology
Article was published inClinical Oncology

2013 Issue 4-
All articles in this issue
- Modern Imaging Techniques for Anthracycline Cytostatics – Review of the Literature
- Contemporary Trends of the Adjutant Chemotherapy in Non‑ small Cell Lung Cancer
- Degradation of Proteins by Ubiquitin‑Proteasome Pathway
- Pancreatic Cancer and Lifestyle Factors
- Chromosome Banding Analysis of Peripheral Blood Lymphocytes Stimulated with IL‑2 and CpG Oligonucleotide DSP30 in Patients with Chronic Lymphocytic Leukemia
- Registry of Neuroendocrine Tumors (NET) in Czech Republic After Three Years of Data Collection
- Is the Same Tyrosine Kinase Inhibitor Still Effective After Development of Brain Metastases? A Case Report
- Týdenní vs dvoutýdenní aplikace cetuximabu v léčbě metastatického kolorektálního karcinomu – aktuální klinická data
- Sphere of Surgical Oncology
- Synchronous Bilateral Testicular Germ Cell Tumour: Case Report and Review of the Literature
-
Oncology in Images
Giant Metastatic Testicular Tumor
- Clinical Oncology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Registry of Neuroendocrine Tumors (NET) in Czech Republic After Three Years of Data Collection
- Degradation of Proteins by Ubiquitin‑Proteasome Pathway
- Contemporary Trends of the Adjutant Chemotherapy in Non‑ small Cell Lung Cancer
- Chromosome Banding Analysis of Peripheral Blood Lymphocytes Stimulated with IL‑2 and CpG Oligonucleotide DSP30 in Patients with Chronic Lymphocytic Leukemia
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

