-
Medical journals
- Career
Implementace array vyšetření do prenatální diagnostiky v I. trimestru
Authors: M. Trková; M. Putzová; V. Bečvářová; J. Horáček; I. Soldátová; L. Krautová; M. Sekowská; J. Hodačová; L. Hnyková; E. Hlavová; D. Smetanová; D. Stejskal
Authors‘ workplace: Gennet, Centrum lékařské genetiky a reprodukční medicíny, Praha, vedoucí lékař MUDr. D. Stejskal
Published in: Ceska Gynekol 2015; 80(3): 176-180
Overview
Cíl studie:
Analýza klinického přínosu array vyšetření choriové biopsie (CVS) a návrh efektivnějšího postupu genetického vyšetření v I. trimestru.Typ studie:
Retrospektivní studie.Název a sídlo pracoviště:
Gennet, Centrum lékařské genetiky a reprodukční medicíny, Praha.Materiál a metodika:
V rámci prenatální diagnostiky v I. trimestru bylo u 913 vzorků CVS provedeno QF-PCR (screening aneuploidií chromozomů 13,18, 21, X, Y) a stanovení karyotypu. Paralelně s těmito metodami bylo u 179 vzorků s normálním výsledkem z obou metod provedeno vyšetření SNP-array (Illumina HumanCytoSNP12 v2.1).Výsledky:
Metodou QF-PCR bylo zachyceno 229 chromozomálních aneuploidií z 911 úspěšně provedených vyšetření (25 %). Konvenčními cytogenetickými metodami byly zachyceny nebalancované chromozomální aberace u 239 z 897 úspěšně vyšetřených plodů (27 %), v 95 % šlo o potvrzení výsledku QF-PCR (227/239), 10 nebalancovaných chromozomálních aberací nezahrnovalo chromozomy sledované metodou QF-PCR. Metodou array bylo u plodů s normálním výsledkem z obou výše uvedených metod odhaleno dalších 13 klinicky relevantních chromozomálních aberací (7,5 %).Závěr:
Na základě analýzy našich dat a publikovaných studií jsme v laboratořích Gennetu navrhli nový algoritmus pro vyšetření choriových klků v I. trimestru. Hlavní změnou je nahrazení karyotypu metodou array u všech plodů, kde je normální výsledek z QF-PCR. Výsledkem bude efektivnější záchyt patologických klinicky relevantních chromozomálních aberací u vyšetřovaných plodů.Klíčová slova:
CVS, prenatální diagnostika, QF-PCR, karyotyp, arrayÚVOD
Molekulárně cytogenetická metoda array postupně nahrazuje konvenční cytogenetickou metodu (karyotyp) jako test první volby u postnatálních vyšetření pacientů s vývojovými odchylkami. V současné době je metoda array všeobecně přijímána jako součást standardního prenatálního vyšetření u gravidit s abnormalitami u plodu [1, 9]. Její hlavní výhodou ve srovnání se standardním karyotypováním je rozlišení: 0,1–0,5 Mb array vs 5–10 Mb karyotyp; u plodů s ultrazvukovými abnormalitami je vyšetřením array zachyceno dalších 7 % chromozomálních aberací [5]. Na základě zkušeností získaných paralelním používáním obou cytogenetických metod (karyotyp a array) v letech 2010–2014 jsme pro vyšetření plodů, kde byla provedena biopsiechoriových klků (CVS), navrhli nové vyšetřovací schéma.
SOUBOR A METODIKA
Od 1. 1. 2010 do 31. 12. 2014 bylo v cytogenetické laboratoři přijato 913 vzorků CVS (tab. 1). Indikace pro odběr CVS byly: pozitivní screening v I. trimestru (NT – nuchální translucence; PAPP-A – pregnancy-associated plasma protein A; fβhCG –freeβ-human chorionic gonadotropin), věk matky, abnormální ultrazvukový nález, chromozomová aberace v anamnéze. Standardní schéma vyšetření všech přijatých vzorků CVS zahrnovalo rychlý screening základních aneuploidií metodou QF-PCR (kvantitativní fluorescenční PCR) a vyšetření karyotypu z dlouhodobé kultivace dvou nezávislých kultur buněk choria. Část tkáně byla po očištění předána do molekulárně genetické laboratoře, kde byl do druhého pracovního dne proveden screening aneuploidií chromozomů 13, 18, 21, X, Y (OmniPlex) [7]. Část tkáně byla nasazena na dlouhodobou kultivaci ve dvou nezávislých kulturách a po sklizení buněk byla provedena karyotypizace (doba trvání vyšetření průměrně dva týdny). U 179 vzorků byla ze záložního vzorku nebo kultivovaných buněk CVS izolována DNA a paralelně s karyotypem bylo provedeno vyšetření array na chipu Illumina HumanCytoSNP-12v2.1 (Illumina Inc., San Diego, CA). Indikace pro vyšetření array jsou shrnuty v tabulce 2. Postup metody a hodnocení SNP array byl prováděn podle instrukcí výrobce a stručně byl popsán v předchozí práci [3]. Skenování bylo provedeno na přístroji iScan (Illumina Inc., San Diego, CA). Data byla analyzována softwarem firmy Illumina GenomeStudio v2011.1 a KaryoStudio v1.4.
Table 1. Přehled výsledků vyšetření CVS za období 2010–2014 (paralelní vyšetření QF-PCR a karyotypizace) 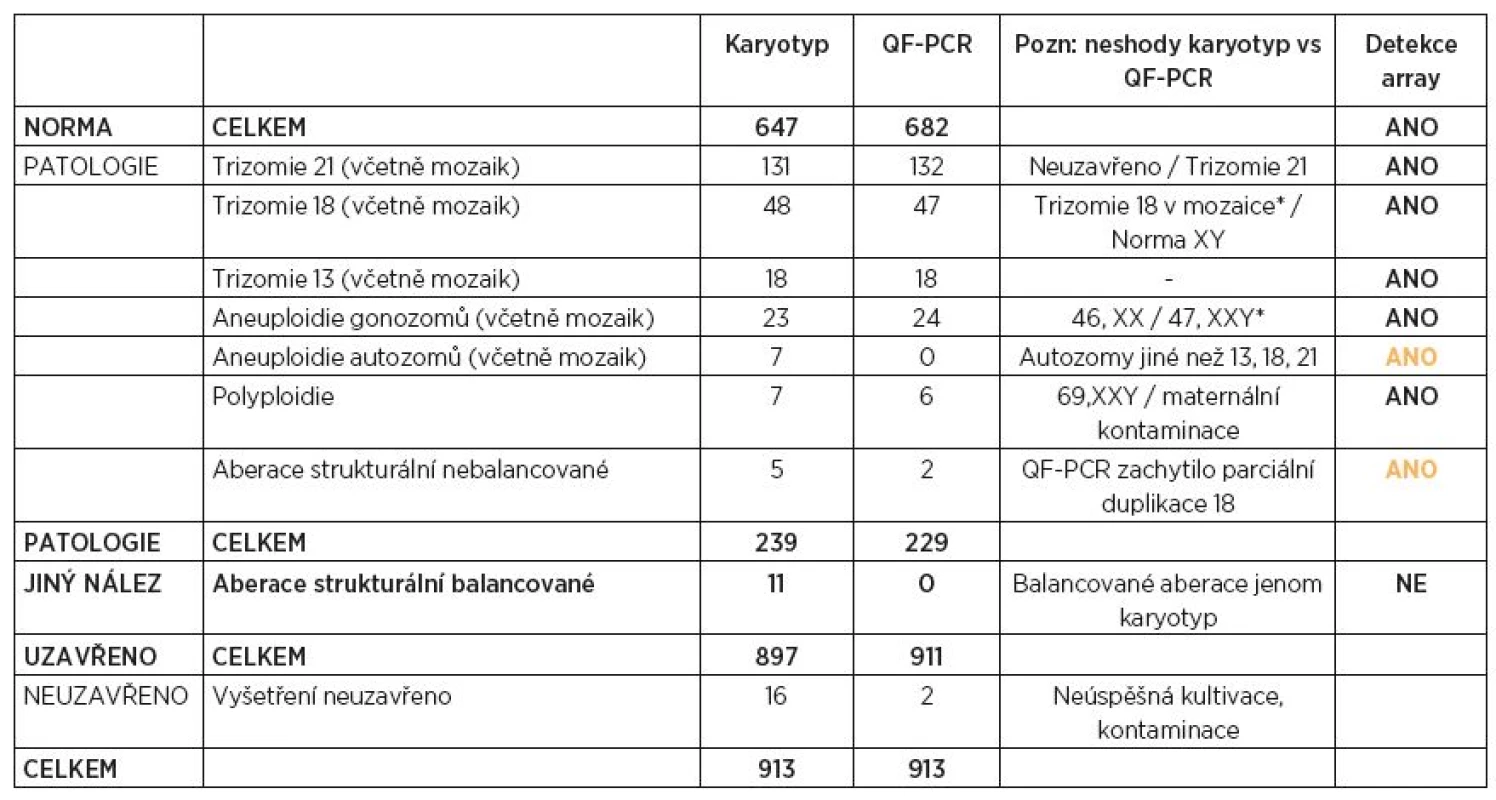
*pravděpodobně fetoplacentární mozaicismus, viz text VÝSLEDKY
V průběhu 4 let bylo paralelně metodami QF-PCR a karyotypizace vyšetřeno 913 vzorků choriových klků. Kompletní výsledky jsou uvedeny v tabulce 1. Metoda QF-PCR byla úspěšná v 911 případech, výsledek nebyl uzavřen pouze ve dvou případech z důvodu masivní maternální kontaminace vzorku (0,2 %). Aneuploidie chromozomů byla zjištěna u 229 CVS (25 %). Cytogenetická laboratoř vydala celkem 897 kompletních výsledků, v 16 případech (1,8 %) nebylo možné karyotyp stanovit (neúspěšná dlouhodobá kultivace). U 239 (27 %) plodů byla nalezena patologická nebalancovaná chromozomální aberace; ve 227 případech karyotyp potvrzoval patologický nález z QF-PCR (95 %). V porovnání s QF-PCR karyotyp odhalil navíc 10 patologických aneuploidií a strukturálních přestaveb autozomů nezahrnutých ve sreeningu. Pouze jedno vyšetření CVS nebylo uzavřeno ani jednou metodou (masivní kontaminace tkání matky).
Byly zjištěny tři neshody (0,3 %) mezi výsledky obou laboratoří: 1. v karyotypu zachycená mozaika trizomie chromozomu 18 nebyla potvrzena ani metodou QF-PCR, ani následným cytogenetickým vyšetřením plodové vody;2. v karyotypu stanovená trizomie chromozomu 21 nebyla v prvním dodaném vzorku CVS metodou QF-PCR prokázána, při opakované izolaci DNA jiného vzorku tkáně byla trizomie 21 potvrzena; 3. gonozomální aneuploidie XXY nalezená metodou QF-PCR nebyla v karyotypu plodu potvrzena (46,XY). Všechny tři případy odpovídají popisovanému jevu placentárního mozaicismu, jehož incidence se uvádí mezi 1 a 2 % [6].
U 179 vzorků choriových klků bylo paralelně s QF-PCR a karyotypem provedeno vyšetření array. Nejčastější indikací k array vyšetření byl ultrazvukový nález (150 ze 174; 84 %). U pěti případů šlo o upřesnění patologického karyotypu (marker chromozomy, derivované chromozomy). Celkem 174 plody vyšetřené metodou array měly normální karyotyp a nebyly u nich detekovány aneuploidie metodou QF-PCR (u pěti případů byla z QF-PCR suspektní mikrodelece nebo mikroduplikace na chromozomech 13 a 18).
U 14 ze 174 (8 %) jsme našli klinicky relevantní mikroskopicky nedetekovatelnou chromozomální aberaci; v osmi případech šlo o mikrodeleci, ve třech případech o mikroduplikaci, u dvou případů byla kombinace mikrodelece a mikroduplikace současně (tab. 2). Žádná z těchto aberací nebyla vzhledem k velikosti (0,5 Mb až 7 Mb) zachycena při hodnocení karyotypu (standardní rozlišení mitóz z choriových klků bylo 500 pruhů).
Table 2. Přehled indikací k vyšetření a výsledky array u 179 vzorků CVS za období let 2010–2014 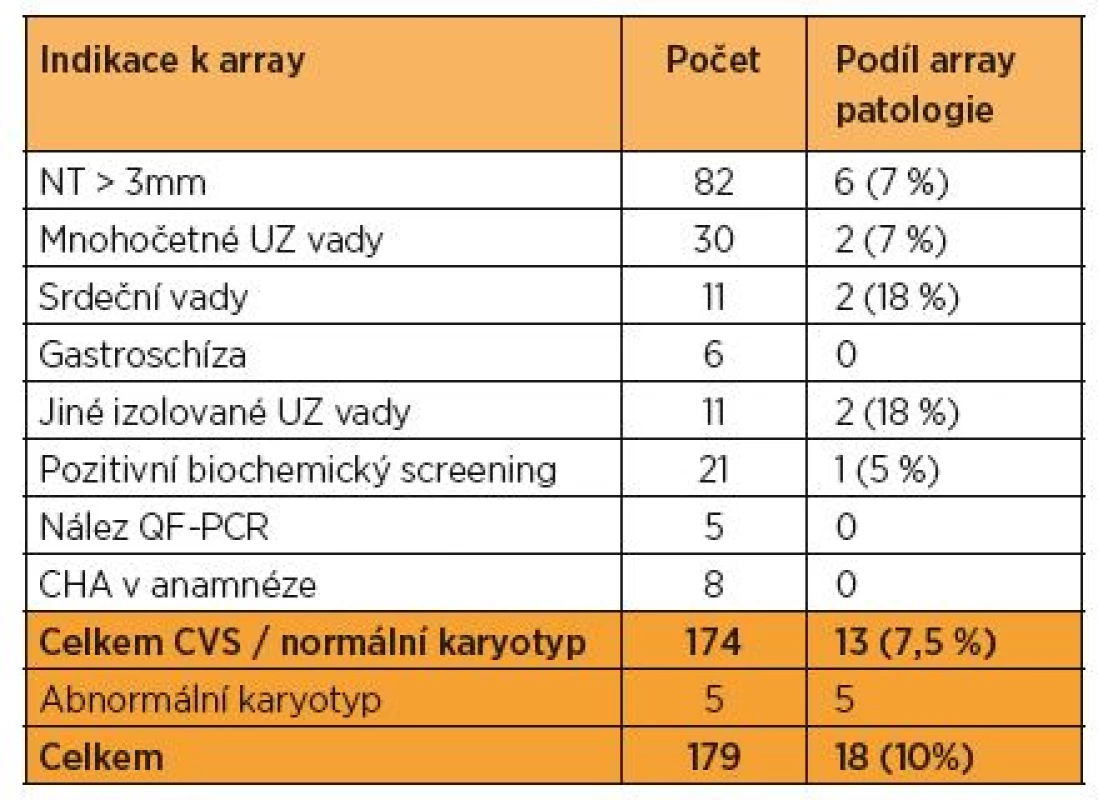
NOVÝ POSTUP PRO PRENATÁLNÍ VYŠETŘENÍ ZE VZORKU CHORIOVÝCH KLKŮ
Na základě našich i publikovaných výsledků [11, 12] jsme se rozhodli upravit vyšetřovací schéma u prenatálních vyšetření ze vzorku choriových klků (obr. 1) s platností od 1. 1. 2015. Zásadní změnou je ukončení plošného stanovování karyotypu u všech vzorků CVS s normálním výsledkem QF-PCR a jeho nahrazení metodou array. Hlavním argumentem pro takové rozhodnutí je fakt, že všechny nebalancované aberace zachycené v karyotypu jsou zachytitelné i metodou array. Karyotyp se bude ze záložní dlouhodobé kultivace CVS stanovovat pro ověření volné respektive translokační formy trizomie 13 a 21, dále pak v kombinaci s metodou FISH pro upřesnění chromozomální aberace detekované metodou array (v některých případech). Analýzou velikosti klinicky relevantních aberací a ve shodě s publikovanými údaji jsme se rozhodli stanovit standardní hranici rozlišení metody array 0,5 Mb [10].
Image 1. Nové schéma vyšetření vzorků choriových klků (CVS) v laboratořích Gennet 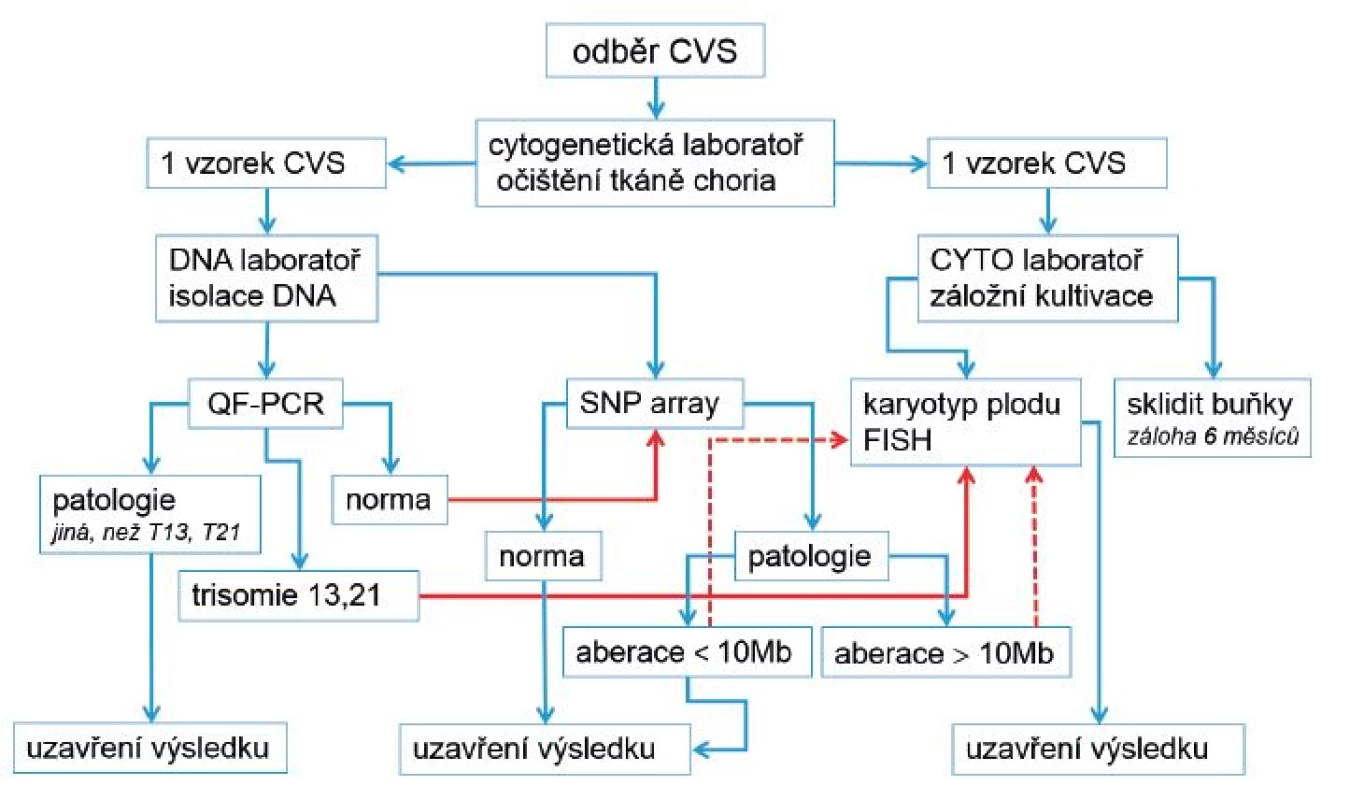
DISKUSE
QF-PCR pro odhalení základních aneuploidií a konvenční cytogenetická metoda stanovení karyotypu jsou standardní součástí prenatálního vyšetření choriových klků. V současné době se v rámci prenatální diagnostiky používá i metoda array, v některých laboratořích pak metoda array zcela nahrazuje konvenční cytogenetické metody [11, 12]. V laboratořích Gennetu se v letech 2010 až 2014 metoda array indikovala jako doplňková metoda k metodám výše uvedeným u 20 % CVS (179 z 913). Největší výhodou metody array ve srovnání s karyotypem je její rozlišení. Můžeme zachytit aberace menší než 1 Mb, zatímco v karyotypu vidíme aberace o minimální velikosti 5–10 Mb. V našem souboru 174 plodů s normálním výsledkem QF-PCR a normálním karyotypem jsme nalezli u dalších 13 (7,5 %) klinicky relevantních submikroskopických chromozomálních aberací (tab. 2). Tento záchyt je ve shodě s publikovanými údaji jiných pracovišť [4, 5, 8, 13].
Nejčastější indikací (150/179, 84 %) k doplnění array u vyšetřovaných plodů byly abnormální ultrazvukové nálezy (tab. 2). U 12 % (21 ze 179) bylo array vyšetření pouze u plodů s pozitivním biochemickým screeningem, přičemž ve třech případech jsme prováděli array ze záložního vzorku CVS až na základě abnormálního ultrazvukového nálezu ve 20. týdnu těhotenství. U dvou plodů (1 %) s normálním ultrazvukovým obrazem ve 12. týdnu jsme našli klinicky významnou chromozomální mikrodeleci.
Analýzou všech aberací nalezených v karyotypu jsme zjistili, že všechny nebalancované přestavby, aneuploidie autozomů a jejich mozaiky jsou zachytitelné metodou array.
V našem souboru bylo v karyotypu nalezeno 11 balancovaných aberací (inverze, translokace): pět aberací vzniklých de novo a šest maternálního nebo paternálního původu. Je všeobecně přijímáno, že balancovaná chromozomální aberace děděná od zdravého rodiče nepředstavuje pro potomka zvýšené riziko. V literatuře uváděné 6% riziko fenotypových odchylek u nosičů de novo vzniklé balancované aberace souvisí zejména s přítomností submikroskopických delecí a duplikací v místě chromozomálních zlomů [2] a takové změny jsou metodou array detekovatelné.
Porovnáním výsledků QF-PCR a karyotypu jsme nalezli několik neshod (tab. 1). Pravděpodobným důvodem je v literatuře popisovaný jev fetoplacentárního mozaicismu, jehož incidence se uvádí mezi 1 a 2 % [6]. Falešně negativní výsledek QF-PCR lze odhalit nezávislým vyšetřením array, nejasný výsledek z metody QF-PCR, array nebo obou může být vždy doplněn karyotypem.
Izolaci DNA k vyšetření je možné provádět jak z kultivované tkáně, tak i z tkáně nekultivované, eliminuje se tak riziko neúspěšné kultivace (u 2 % vzorků jsme nevydali karyotyp právě z důvodu neúspěšné kultivace). Kontaminace maternální tkání bude jako dosud kontrolována vyšetřením DNA izolované z periferní krve matky.
Hlavním úskalím metody array je nalézání mikrodelecí a mikroduplikací nejasného významu nebo aberací spojených s neočekávanou diagnózou (např. aberace související s nádorovou predispozicí nebo nemocemi s pozdním nástupem). Na základě vlastní analýzy klinicky relevantních aberací a publikovaných studií [10] jsme zvolili jako hranici rozlišení ≥ 0,5 Mb, čímž bychom měli eliminovat výše popisované riziko.
ZÁVĚR
Na základě všech uvedených skutečností jsme se rozhodli od 1. 1. 2015 změnit strategii prenatálního vyšetření u vzorků CVS a použít jako vyšetření první volby u plodů s normálním výsledkem QF-PCR metodu array s vhodně nastaveným rozlišením (≥ 0,5 Mb). Výsledkem tohoto nového algoritmu by měl být efektivnější záchyt patologických, klinicky relevantních chromozomálních aberací, které mohou být klíčové pro rozhodování o osudu gravidity.
RNDr. Marie Trková, Ph.D.
Gennet
Centrum lékařské genetiky a medicíny
Kostelní 292/9
170 00 Praha 7
e-mail: marie.trkova@gennet.cz
Sources
1. ACOG Committee Opinion No. 581: The use of chromosomal microarray analysis in prenatal diagnosis. 2013, Obstet Gynecol, 122(6), p. 1374–1377.
2. Baptista, J., Mercer, C., Prigmore, E., Gribble, SM., et al. Breakpoint mapping and array CGH in translocations: comparison of a phenotypically normal and abnormal cohort. 2008, Am J Hum Genet, 82, p. 927–936.
3. Bečvářová, V., Hynek, M., Putzová, M., et al. [Application of SNP array method in prenatal diagnosis]. Ces Gynek, 2011, 76(4), p. 261–217.
4. Breman, A., Pursley, AN., Hixson, P., Ward, P., et al. Prenatal chromosomal microarray analysis in a diagnostic laboratory; experience with >1000 cases and review of the literature. Prenat Diagn, 2012, 32(4), p. 351–361.
5. De Wit, MC., Srebniak, MI., Govaerts, LC., et al. Additional value of prenatal genomic array testing in fetuses with isolated structural ultrasound abnormalities and a normal karyotype: a systematic review of the literature. Am J Ultrasound Obstet Gynecol, 2014, 43(2), p. 139–146.
6. Hahnemann, JM., Vejerslev, LO. European collaborative research on mosaicism in CVS (EUCROMIC) – fetal and extrafetal cell lineages in 192 gestations with CVS mosaicism involving single autosomal trisomy. Med Genet, 1997, 70(2), p. 179–187.
7. Putzová, M., Pecnová, L., Dvořáková, L., et al. OmniPlex--a new QF-PCR assay for prenatal diagnosis of common aneuploidies based on evaluation of the heterozygosity of short tandem repeat loci in the Czech population. Prenat Diagn, 2008, 28(13), p. 1214–1220.
8. Shaffer, LG., Rosenfeld, JA., Dabell, MP., Coppinger, J., et al. Detection rates of clinically significant genomic alterations by microarray analysis for specific anomalies detected by ultrasound. Prenat Diagn, 2012, 32(10), p. 986–995.
9. Srebniak, MI., Boter, M., Oudesluijs, GO., et al. Genomic SNP array as a gold standard for prenatal diagnosis of foetal ultrasound abnormalities. Mol Cytogenet, 2012, 5(1), p. 14.
10. Srebniak, MI., Mout, L., Van Opstal, D., Galjaard, RJ. 0.5 Mb array as a first-line prenatal cytogenetic test in cases without ultrasound abnormalities and its implementation in clinical practice. Hum Mutat, 2013, 34(9), p. 1298–1303.
11. Vanakker, O., Vilain, C., Janssens, K., Van der Aa N., et al. Implementation of genomic arrays in prenatal diagnosis: the Belgian approach to meet the challenges. Eur J Med Genet, 2014, 57(4), p. 151–156.
12. Van Opstal, D., de Vries, F., Govaerts, L., Boter, M., et al. Benefits and burdens of using a SNP array in pregnancies at increased risk for the common aneuploidies. Hum Mutat, 2014, doi: 10.1002/humu.22742. [Epub ahead of print].
13. Vestergaard, EM., Christensen, R., Petersen, OB., Vogel, I. Prenatal diagnosis: array comparative genomic hybridization in fetuses with abnormal sonographic findings. Acta Obstet Gynecol Scand, 2013, 92(7), p. 762–768.
Labels
Paediatric gynaecology Gynaecology and obstetrics Reproduction medicine
Article was published inCzech Gynaecology

2015 Issue 3-
All articles in this issue
- Tubulo-skvamózní polyp vaginy
- Implementace array vyšetření do prenatální diagnostiky v I. trimestru
- Vedomosti respondentov na Slovensku a v Čechách o karcinóme cervixu – projekt Aurora
- Specifické placentární komplikace u monochoriálních biamniálních dvojčat porozených po 24. týdnu gravidity – retrospektivní analýza
- Porovnání kvality života pacientek léčených pro stresovou inkontinenci moči operačními přístupy AJUST a TVT-O – tříměsíční výsledky randomizované studie
- Význam uroflowmetrie v diagnostice poruch dolních močových cest
- Torze omenta – překvapivá příčina náhlé příhody břišní v graviditě – kazuistika
- Ektopie srdce – kazuistika
- Agresivní malobuněčný karcinom ovaria hyperkalcemického typu, chirurgická a onkologická léčba: kazuistika
-
Problematika niektorých infekčných ochorení tehotných žien v každodennej praxi
Časť I. Bakteriálne a parazitárne ochorenia - Prevence preeklampsie – přehledový článek
- Význam vyšetření protiláteka krevní skupiny v těhotenství
- Czech Gynaecology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Prevence preeklampsie – přehledový článek
- Význam vyšetření protiláteka krevní skupiny v těhotenství
- Specifické placentární komplikace u monochoriálních biamniálních dvojčat porozených po 24. týdnu gravidity – retrospektivní analýza
- Ektopie srdce – kazuistika
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

