-
Medical journals
- Career
Potenciál dlouhých nekódujících molekul RNA v diagnostice nádorových onemocnění
: B. Gromesová; V. Kubaczkova; B. Bollova; L. Sedlaříková; S. Ševčíková
: Babákova myelomová skupina, Ústav patologické fyziologie, LF MU, Brno
: Klin Onkol 2016; 29(1): 20-28
: Review
prolekare.web.journal.doi_sk: https://doi.org/10.14735/amko201620Dlouhé nekódující molekuly RNA (long non-coding RNA – lncRNA) jsou definovány jako molekuly o délce více než 200 nukleotidů, které jsou lokalizovány v jádře a cytoplazmě buněk. Přestože u většiny lncRNA jejich konkrétní funkce nejsou dosud známé, je evidentní, že se podílejí na celé řadě biologických procesů. LncRNA hrají klíčové role jak v transkripčních, tak v post‑transkripčních regulačních drahách a podílejí se na významných buněčných procesech, jako je proliferace, diferenciace, apoptóza a v neposlední řadě i na patogenezi různých nemocí. Svou deregulací se významně podílejí také na procesech nádorové transformace. V tomto přehledovém článku jsou popsány vlastnosti, funkce a molekulární podstata lncRNA a také jejich diagnostický potenciál. Pozornost je věnována zejména jejich využití u nejčastěji diagnostikovaných nádorových onemocnění v české populaci, a to u kolorektálního karcinomu, karcinomu prsu a prostaty.
Klíčová slova:
dlouhé nekódující molekuly RNA – nádorový marker – deregulace lncRNA – solidní nádoryÚvod
Je známo, že velikost genomu eukaryotických organizmů neodpovídá jejich genetické komplexitě (tzv. paradox hodnoty C). Lidský genom je tvořen 20 000 – 25 000 geny kódujícími proteiny z celkového množství 3 000 Mbp [1]. To odpovídá přibližně 1,5 % genomu, což je mnohem méně, než se původně předpokládalo [2 – 4]. Ačkoli sekvence kódující proteiny tvoří pouze minimální část genomu, nejméně 90 % z něj je transkripčně aktivní. To naznačuje, že většinu lidského genomu představují DNA sekvence, které nekódují proteiny. Výsledkem transkripce takovýchto sekvencí je velké množství nekódujících molekul RNA (non‑coding RNA – ncRNA). Původně byly tyto molekuly považovány za transkripty evolučně nahromaděného genetického odpadu, tzv. junk DNA, která vzhledem ke své nízké úrovni exprese není funkční [5 – 9]. Nedávné studie však prokázaly, že transkripty těchto sekvencí se účastní významných fyziologických, ale i patologických buněčných procesů [4,7].
Celkový počet ncRNA v lidském genomu je stále neznámý, ale prostřednictvím dostupných technologií se odhaduje existence několika desítek tisíc těchto molekul [10]. NcRNA jsou děleny do dvou hlavních skupin na základě jejich velikosti [5,6]:
- malé nekódující RNA (short non‑coding RNA – sncRNA), které jsou kratší než 200 nukleotidů (nt) a jsou mezidruhově vysoce konzervované. Patří zde např. mikroRNA (miRNA, 19 – 24 nt), malé interferující RNA (small interfering RNA – siRNA, 20 – 25 nt), RNA spojené s Piwi proteinem (Piwi ‑ interacting RNA – piRNA, 24 – 32 nt) a další;
- dlouhé nekódující RNA (long non‑coding RNA – lncRNA), které představují transkripty nekódující proteiny o délce větší než 200 nt. Jedná se o rozmanitou a mezi druhy málo konzervovanou skupinu. Molekuly lncRNA představují nejméně prostudovanou a pravděpodobně nejrozsáhlejší rodinu nekódujících molekul RNA [10,11].
Mezi nejznámější molekuly RNA nekódující proteiny patří transferové (transfer RNA – tRNA) a ribozomální (ribosomal RNA – rRNA) RNA [11,12]. Tyto molekuly byly identifikovány před více než 50 lety a svou funkcí se významně podílejí na syntéze proteinů [13]. Studie těchto molekul ukázaly, že ncRNA jsou nedílnou součástí mnoha biologických procesů v živých organizmech, vč. člověka.
Vlastnosti lncRNA
Tyto molekuly jsou definovány jako transkripty delší než 200 nt s mediánem okolo 600 nt a obvykle dosahují kratších délek než protein‑kódující mediátorové RNA (messenger RNA – mRNA) [14], a to i přesto, že jejich exony i introny bývají delší než u molekul mRNA – většina lncRNA obsahuje pouze dva exony [15].
Jak již bylo zmíněno, sekvence kódující lncRNA pokrývají větší část lidského genomu než sekvence genů kódujících proteiny. Geny pro lncRNA lze nalézt v různých oblastech na všech chromozomech (obr. 1) [11,15]. Nacházejí‑li se tyto geny v intergenových oblastech v určité vzdálenosti od kódujících genů, jsou pak tyto molekuly označovány jako dlouhé intergenové nekódující RNA (long non‑coding RNA – lincRNA). Pokud se jejich geny nacházejí v intronech různých genů, jde o intronové lncRNA [6,7,11,15 – 17]. Jsou‑li přepisovány ze stejného řetězce jako geny kódující proteiny, hovoříme o „sense“ lncRNA, jsou‑li transkribovány z opačného řetězce, jedná se o „antisense“ lncRNA. Také se mohou vyskytovat jako divergentní ‑ obousměrné sekvence, které sdílejí promotor s jiným transkriptem v opačném řetězci [12,18]. Sekvence pro lncRNA se mohou překrývat se sekvencemi jiných transkriptů. Přesto lze jejich lokalizaci v genomu využít jako jeden z přístupů klasifikace.
1. Typy molekul lncRNA podle lokalizace genů v genomu. 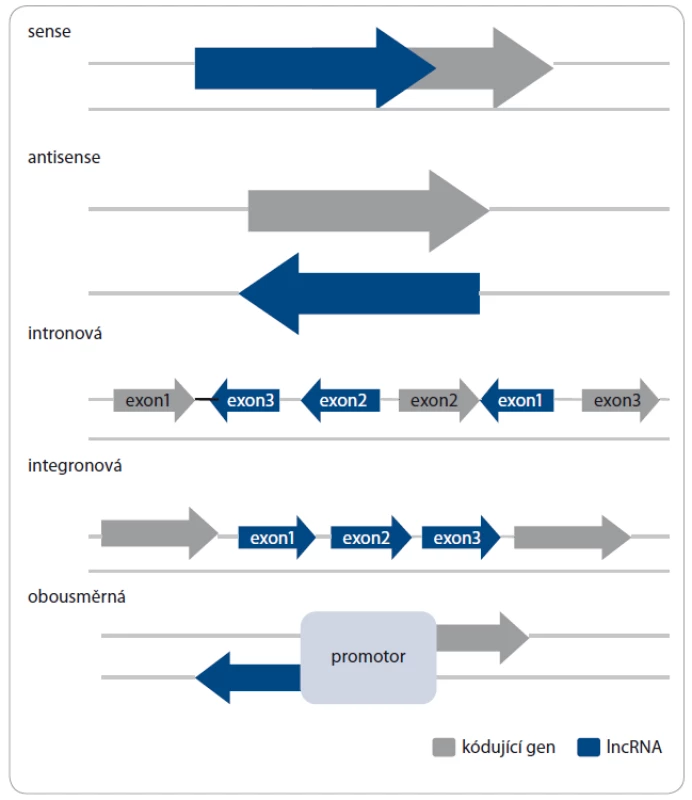
Ohromné množství a neúplné informace o těchto molekulách činí klasifikaci lncRNA velmi obtížnou. Další komplikací je skutečnost, že stejné vlastnosti a funkce mohou být sdíleny více molekulami. LncRNA lze dělit např. podle výše zmíněné genomové lokalizace, vlivu na DNA sekvence, mechanizmu funkcí [6] nebo pomocí specifických charakteristik (tab. 1) [18].
1. Příklady klasifikace lncRNA [6,13,16,18]. ![Příklady klasifikace lncRNA [6,13,16,18].](https://www.prolekare.cz/media/cache/resolve/media_object_image_small/media/image/f6eea1606d0e68c46eb77c3785e7a447.png)
O molekulárních a biologických vlastnostech lncRNA je doposud známo jen omezené množství informací. V několika ohledech jsou však podobné genům kódujícím proteiny [11,14,15,18 – 20]. Molekuly lncRNA jsou přepisovány RNA polymerázou II nebo III podle struktury jejich promotorových sekvencí [5,10,11,19]. Přepis RNA polymerázou III byl prokázán u molekul lncRNA, které mají funkční úlohu v transkripčním mechanizmu RNA polymerázy II [10]. Taktéž mohou být molekuly lncRNA upraveny post‑transkripčními modifikacemi (5’ čepička, 3’ polyadenylace), jako je tomu u mRNA. Přítomnost 3’ poly (A) konce je u těchto molekul zodpovědná za poměrně vysokou stabilitu [11,14,15,20]. Není‑li molekula polyadenylována, může se na jejím 3’ konci nacházet trojšroubovice, která taktéž slouží ke stabilizaci molekuly [21].
Geny pro lncRNA podléhají epigenetickým regulacím – modifikacím histonů, které se však mohou odlišovat od modifikací histonů genů kódujících mRNA. U standardních genů kódujících proteiny probíhají např. modifikace H3K4me3 a H3K3me6, u molekul lncRNA dochází k modifikaci H3K4me2/ 3, H3K9ac a H3K27ac [11,14].
Obecně jsou lncRNA méně stabilní než mRNA a zdá se, že jejich stabilita závisí i na jejich různých vlastnostech, např. na genomové lokalizaci (intergenové lncRNA jsou stabilnější než intronové), na sestřihu (molekuly podléhající sestřihu jsou stabilnější) nebo na buněčné lokalizaci (jaderné se mohou rozpadat dříve než lncRNA lokalizované v cytoplazmě) [11]. Po post‑transkripčních úpravách přechází mRNA do cytoplazmy, kde dochází k translaci do proteinů. U lncRNA je translace znemožněna nepřítomností otevřených čtecích rámců (open reading frame – ORF) nebo jejich nedostatečnou délkou (ORF jsou kratší než 100 aminokyselin) [5,7,12,15]. Nižší kódující potenciál může vysvětlovat častější lokalizaci lncRNA v jádře než v cytoplazmě, což bylo prokázáno fluorescenční in situ hybridizací (FISH) [13 – 15,18,19,22].
LncRNA se vyznačují nízkou expresí ve srovnání s hladinou exprese genů kódujících proteiny, avšak jejich exprese je tkáňově specifická a je rozdílná v různých vývojových stadiích člověka, za fyziologických i patologických podmínek. Z toho vyplývá, že lncRNA se účastní důležitých biologických procesů, jako je alternativní sestřih, epigenetické regulace, transkripce, translace, regulace buněčného cyklu, proteinové lokalizace, ale také diferenciace buněk, vč. hematopoézy, proliferace, apoptózy a např. i procesů souvisejících s maligní transformací [11,15,19].
Konzervace lncRNA
LncRNA se nacházejí u různých živočišných druhů, jejich přítomnost byla popsána také u rostlin, kvasinek, prokaryot, a dokonce i virů [6]. Přestože se vyskytují u široké škály organizmů, jejich obrovská rozmanitost vypovídá o špatné konzervaci mezi druhy, na rozdíl od sncRNA, které jsou mezidruhově významně konzervovány [4,8,23]. Celkově jsou lncRNA konzervovány méně než protein kódující geny, ale naopak více než introny nebo náhodné intergenové oblasti [8,15,20,24]. Existují čtyři možné varianty konzervace lncRNA [12,17,25]:
- Konzervace nukleotidové sekvence – je podobná konzervaci genů kódujících protein. Obvykle bývají zachovány pouze krátké úseky sekvence, což vysvětluje, proč se lncRNA tak rychle vyvíjejí a často postrádají ortology u jiných druhů.
- Konzervace struktury za předpokladu, že molekuly lncRNA jsou skládány do sekundárních či terciárních struktur. Tyto struktury jsou důležitější pro zachování jejich funkcí, a proto jsou pravděpodobně přednostně konzervovány před sekvencemi nukleotidů.
- Konzervace jejich funkce – vzhledem k nedostatečným znalostem o funkcích lncRNA se frekvence tohoto typu zachování nedá odhadnout.
- Konzervace transkripce genového lokusu pro lncRNA – transkripce ze stejného lokusu ovlivňuje i genovou expresi sousedních genů.
Všechny tyto čtyři způsoby konzervace se mohou vzájemně kombinovat. Přestože jsou lncRNA špatně konzervovány, jejich promotorové oblasti jsou ve srovnání s exony mnohem více zachovány. Stupeň konzervace je téměř stejný jako u promotorů mnoha genů kódujících proteiny [20]. Překvapivým objevem se staly lncRNA, které jsou transkribovány z tzv. ultrakonzervovaných oblastí (UCR) [16,18]. Tyto oblasti jsou delší než 200 párů bází a jsou zcela konzervovány u různých živočišných druhů, vč. člověka. Transkripty těchto oblastí se označují jako T ‑ UCR a jejich vlastnosti nejsou ještě zcela známy.
Funkce lncRNA
Vzhledem k nízkým hladinám exprese a špatné konzervaci primární sekvence těchto molekul se v minulosti předpokládalo, že lncRNA nehrají významnou roli v buněčných funkcích. Neustále se však ukazuje, že tato představa byla mylná. Ačkoli u nově objevených transkriptů je jako indikátor funkčního významu široce používána evoluční konzervace [25], u lncRNA je více než konzervace sekvence nukleotidů pro zachování funkce důležitější konzervace jejich sekundární či terciární struktury [26].
Přestože u většiny lncRNA jejich konkrétní funkce nejsou dosud známé, je evidentní, že se podílejí na celé řadě biologických procesů [15,27]. O tom, na jakých procesech se lncRNA budou podílet, rozhoduje mimo jiné jejich lokalizace v buňce. Na rozdíl od mRNA většina transkriptů lncRNA zůstává v jádře, kde se podílejí na imprintingu, modifikaci chromatinu a především na regulaci genové exprese [4,15,28 – 30]. LncRNA, které jsou transportovány do cytoplazmy, se zde účastní sestřihu mRNA, regulace translace nebo mohou sloužit jako prekurzory pro sncRNA [15]. Vzhledem k množství procesů, kterých se molekuly lncRNA účastní, není nepřekvapivé, že jejich deregulace se může podílet na vzniku mnoha lidských onemocnění, vč. těch nádorových.
Jednou z hlavních funkcí lncRNA je ovlivňování DNA, mohou regulovat genovou expresi, a to buď in cis (např. lncRNA Xist, Kcnq1ot1), nebo in trans (HOTAIR) [29,31]. Cis působící lncRNA vedou epigenetické regulátory k jejich místu transkripce, zatímco jsou samy transkribovány. Tímto způsobem mohou pozitivně či negativně regulovat expresi sousedních genů [24]. Zatímco trans působící lncRNA regulují expresi genů na větší vzdálenosti. Mohou tak regulovat i expresi genů umístěných na jiných chromozomech, než na kterých jsou samy geny pro lncRNA lokalizovány [30 – 32]. LncRNA mohou vytvářet různé interakce s DNA, např. RNA:DNA heteroduplex nebo RNA:DNA:DNA triplex [31].
Molekuly lncRNA své funkce zprostředkovávají většinou spojením s různými proteinovými komplexy, např. chromatin modifikujícími komplexy, které epigeneticky modifikují DNA, či s transkripčními faktory, které ovlivňují transkripci cílených genů [20,24,33 – 35].
Molekulární podstata funkce lncRNA
Existují čtyři molekulární mechanizmy, tzv. archetypy (obr. 2), prostřednictvím kterých molekuly lncRNA vykonávají své předpokládané funkce [4,6]:
2. Typy molekulárních mechanizmů lncRNA – tzv. funkční „archetypy“ – signál, návnada, průvodce a lešení. 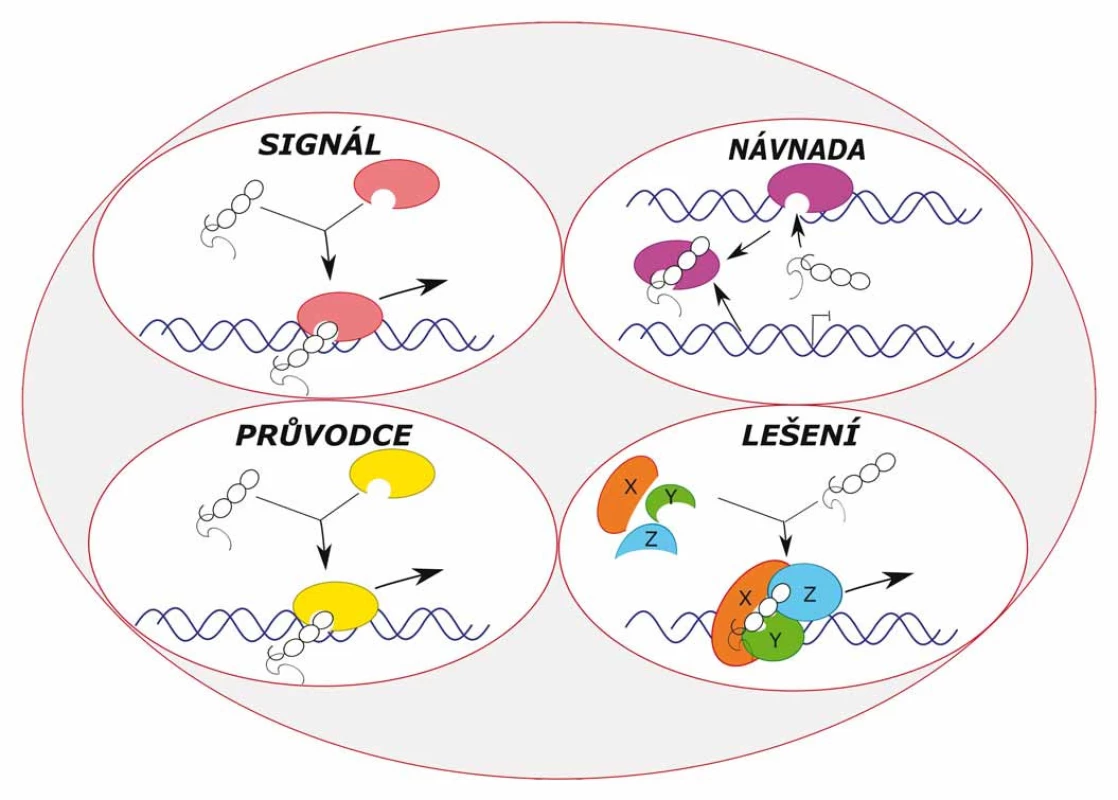
1. Signál – Signal
LncRNA mohou působit jako signály, protože jejich transkripce nastává v konkrétním čase a na konkrétním místě a jejich exprese je specifická pro určitý buněčný typ. Některé tyto lncRNA mají regulační funkci, jiné jsou pouze jako vedlejší produkty transkripce. Jako příklad může sloužit molekula COLDAIR (cold assisted intronic noncoding RNA) nebo XIST (X inactive ‑ specific transcript). Molekula XIST je signálem pro lncRNA TSIX (XIST antisense RNA), která molekulu XIST potlačuje.
2. Návnada – Decoy
Transkripty lncRNA se vážou na cílové proteiny a brání tak jejich navázání na DNA. Vyvazují RNA vazebné proteiny, které jsou samy sobě transkripčními faktory, chromatinové modifikátory nebo jiné regulační faktory. Příkladem jsou molekuly PANDA (P21 associated ncRNA DNA damage activated) a TERRA (telomeric repeat ‑ containing RNAs).
3. Průvodce – Guide
Tyto lncRNA vážou proteiny a přímo lokalizují ribonukleoproteinové komplexy ke konkrétním cílovým místům. Příkladem jsou molekuly XIST a HOTAIR (hox transcript antisense intergenic RNA). Molekula XIST váže polycomb represivní komplex 2 (PRC2), který propaguje epigenetické umlčení individuálního chromozomu X [36]. Molekula HOTAIR je transkribována z lokusu HOXC, váže se na PRC2, poté dochází k trimetylaci H3K27 a transkripčnímu umlčení lokusu HOXD [30].
4. Lešení – Scaffold
LncRNA zde slouží jako centrální vzor pro shromáždění více proteinů pro tvorbu ribonukleoproteinových komplexů, které stabilizují jaderné struktury, nebo působí jako modifikátory chromatinu a histonů. Příkladem je molekula HOTAIR, která může vázat dva různé komplexy modifikující histony (např. PRC2 a LSD1/ CoREST/ REST komplexy) a slouží jako molekulární lešení pro jejich propojení [35].
Každá z molekul lncRNA nepracuje prostřednictvím pouze jednoho molekulárního mechanizmu, ale k dosažení jejich biologické funkce využívá různé kombinace těchto čtyř archetypů [4]. Například molekula HOTAIR využívá archetyp 3 a 4.
LncRNA u nádorových onemocnění
Kancerogeneze je vícestupňový a velmi složitý proces, při kterém dochází k nahromadění genetických i epigenetických změn. V průmyslově vyspělých zemích je pravděpodobnost vzniku nádoru během života jedince poměrně vysoká. Přestože dochází k výrazným pokrokům v léčebných postupech, nádorová onemocnění stále patří mezi nejčastější příčiny úmrtí u všech věkových skupin v populaci. U nádorových onemocnění jsou vysoce sledovanými jevy vznik a šíření metastáz a také relaps nemoci. Záměrem vědců je tedy nalézt vhodné diagnostické, prognostické a prediktivní biomarkery, jež by napomohly v diagnostice nádorových onemocnění. Molekuly lncRNA by také mohly sloužit jako potenciální terapeutické cíle.
U mnoha nádorových onemocnění dochází k deregulaci lncRNA, které se podílejí na procesech nádorové transformace. LncRNA se u různých nádorových onemocnění vyznačují změnami jejich exprese [7,28,30,37,38], které jsou pravděpodobně vývojově a tkáňově specifické [37].
LncRNA se účastní vývoje nádorových onemocnění ve třech různých drahách [39]. Za prvé mohou lncRNA působit jako onkogeny nebo onkogenní regulátory. Příkladem může být působení molekuly H19 (imprinted maternally expressed transcript) u karcinomu tlustého střeva [40]. Jako onkogen působí rovněž molekula HOTAIR, jejíž exprese je nízká v normálním epitelu prsní tkáně, ale vysoká u primárního karcinomu prsu a také v případě metastatických lézí [41]. Vyšší hladiny exprese molekuly HOTAIR byly patrné také v nádorové tkáni pacientů ve IV. stadiu kolorektálního karcinomu (colorectal cancer – CRC) oproti zdravým kontrolám [42]. Dalším příkladem je molekula MALAT1 (metastasis‑associated lung adenocarcinoma transcript 1), která je široce exprimována ve zdravé lidské tkáni [43,44], ale k její zvýšené expresi taktéž dochází u šesti různých typů nádorů [38,39,45,46]. Za druhé mohou být lncRNA spojovány se vznikem metastáz a prognózou onemocnění, což dokládá studie, ve které použitím kvantitativní PCR (polymerázová řetězová reakce) byla zjištěna zvýšená exprese molekuly HOTAIR u pacientů s karcinomem prsu. Tato molekula se jevila jako významný prediktor nadcházejících metastází a úmrtí pacientů [41]. A nakonec lncRNA mohou sloužit také jako tumor supresorové geny podporující tumor supresorové dráhy. Příkladem je molekula MEG3 (maternally expressed 3), která může stimulovat transaktivaci zprostředkovanou p53 a také potlačení syntézy DNA [26]. Dalším příkladem je molekula GAS5 (growth arrest ‑ specific 5), která byla prokázána u několika druhů nádorů (např. u melanomu, karcinomu prsu a prostaty) [47,48], ačkoli její funkce nebyla dosud plně objasněna.
Přestože je počet molekul lncRNA vysoký, pravděpodobně pouze relativně malé množství z nich je zahrnuto do patogeneze nádorových onemocnění [49]. V tomto přehledovém článku jsou popsány molekuly lncRNA, které hrají významnou roli u třech nejčastěji diagnostikovaných zhoubných nádorových onemocnění v české populaci, a to CRC, karcinomu prsu a prostaty.
Karcinom prsu
Prsní žláza je orgán, který po celý život podstupuje cykly vývoje a regrese [50]. Je zde tedy vysoká pravděpodobnost maligní transformace, a proto není zcela překvapivé, že karcinom prsu patří spolu s karcinomem kůže mezi nejčastější nádorová onemocnění žen. Karcinom prsu se může vyvinout i u mužů, ale tvoří pouze asi 1 % všech případů. Každý rok je v ČR toto onemocnění nově diagnostikováno u více než 6 000 žen. Ačkoli je léčba karcinomu prsu jednou z nejúspěšnějších, každoročně tomuto onemocnění podlehne přibližně 2 000 žen [51].
Díky úsilí mnoha vědeckých skupin bylo objeveno množství lncRNA molekul, které se jeví jako vhodné biomarkery,jsou jimi např. molekuly HOTAIR a ZFAS1 (ZNFX1 antisense RNA 1) [30,41,52].
Molekula HOTAIR je exprimována z HOXC lokusu, který se nachází v oblasti 12q13.13 [30]. Spolu s PRC2 komplexem trimetyluje lysin histonu H3 v pozici 27 na lokusu HOXD, čímž inhibuje jeho genovou expresi a mění jeho cíl [30,41]. Molekula HOTAIR se také podílí na vzniku metastáz karcinomu prsu tím, že umlčuje několik tumor supresorových genů nádorových buněk (např. HOXD 10, PGR, protokadherinová genová rodina). Gupta a jeho výzkumný tým ve své studii uvedli, že v normálním epitelu prsu je exprese molekuly HOTAIR nízká, ale u primárního karcinomu prsu a u metastatických lézí dochází k jejímu zvýšení. Pacienti s vysokou expresí molekuly HOTAIR měli také horší prognózu pro celkové přežití a pro vznik metastáz oproti pacientům s nízkou expresí této molekuly [41].
Molekula ZFAS1 je antisense transkript k 5’ konci protein kódujícího genu ZNFX1 [52], jenž se může podílet na reparaci DNA. Tato molekula, exprimovaná z lokusu v oblasti 20q13.13, pravděpodobně působí jako represor proliferace a diferenciace. Jejím vyřazením u buněčné linie prsního epitelu došlo ke zvýšení buněčné proliferace a diferenciace, což poukazuje na její funkci nádorového supresoru. U karcinomu prsu je hladina molekuly ZFAS1 snížena oproti normální prsní tkáni. Snížená exprese ZFAS1 by tedy mohla být markerem pro diagnostiku karcinomu prsu.
Detailnější informace ohledně studií jednotlivých molekul lncRNA jsou zahrnuty v tab. 2.
2. Shrnutí studií jednotlivých lncRNA k daným karcinomům. 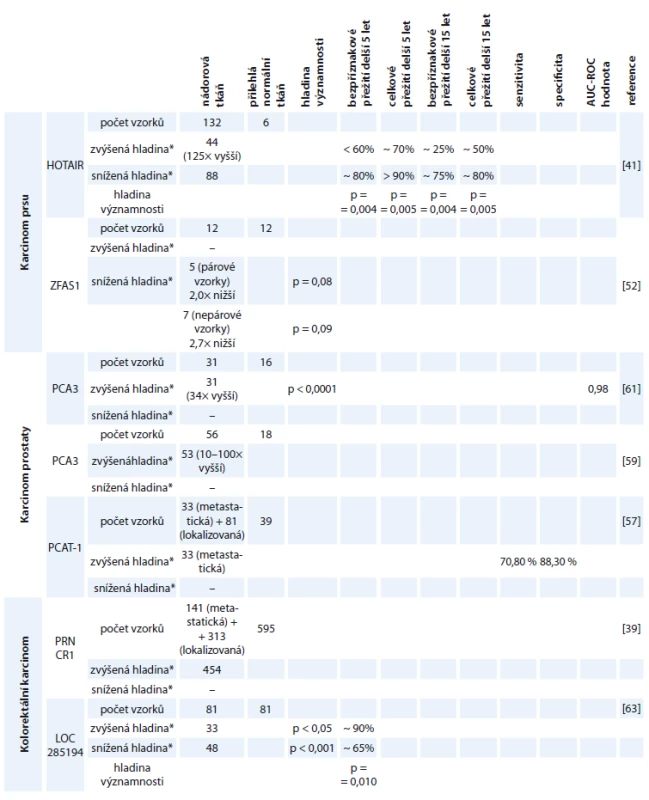
*Hladina jednotlivých lncRNA u daných karcinomů porovnána s hladinou jednotlivých lncRNA v odpovídající přilehlé tkáni. Karcinom prostaty
Karcinom prostaty patří mezi nejčastěji diagnostikovaná nádorová onemocnění u mužů. V ČR každoročně onemocní karcinomem prostaty přibližně 7 000 mužů, z nichž asi 1 500 na toto onemocnění umírá [51]. V počátečních fázích se u většiny pacientů neobjevují žádné příznaky, čímž dochází k záchytu nemoci až v pozdějších stadiích [53]. V současnosti se karcinom prostaty primárně testuje pohmatem skrze rektum a pomocí krevního testu na přítomnost prostatického specifického antigenu, který je vylučován specifickými buňkami prostaty. Problémem tohoto testu je, že i když je pozitivní, nemusí se jednat výhradně o karcinom prostaty. Pozitivní výsledek se může objevit i u jiných zdravotních obtíží prostaty, jako je např. benigní hyperplazie nebo zánět. Proto je snaha najít nové biomarkery, které by pomohly odlišit pacienty s karcinomem prostaty od pacientů s jiným nemaligním onemocněním tohoto orgánu.
Mezi studované molekuly patří např. PCAT ‑ 1 (prostate cancer-associated transcript 1) a PCA3 (prostate cancer antigen 3) [54,55].
Molekula PCAT ‑ 1 patří do skupiny 121 neanotovaných transkriptů PCAT, které jsou schopny od sebe odlišit vzorky benigní, vzorky lokalizovaných maligních nádorů a vzorky metastatické [54]. Gen pro tuto ncRNA je lokalizován na chromozomu 8q24, kde se nachází jednonukleotidový polymorfizmus (single nucleotide polymorphisms – SNP) asociovaný s karcinomem prostaty a onkogen c ‑ MYC. Tento lokus je u nádorových onemocnění často amplifikován, což naznačuje, že může být spojen s dalšími aspekty nádorové biologie. Molekula PCAT ‑ 1 může korelovat s expresí onkoproteinu EZH2, který vykazuje zvýšenou expresi u solidních nádorů, a společně se tak podílejí na vzniku metastází [56,57]. PCAT ‑ 1 může působit dvěma způsoby: prvním, ve kterém PRC2 reprimuje PCAT ‑ 1, a druhým, kde PRC2 nereprimuje PCAT ‑ 1, ta zůstává aktivní a podporuje buněčnou proliferaci. Molekuly PCAT ‑ 1 a PRC2 mohou proto charakterizovat rozdílné podskupiny karcinomu prostaty [54]. Molekula PCAT ‑ 1 byla také testována u buněčných linií karcinomu prsu a plic a u 19 normálních tkáňových typů. Výsledky však ukázaly, že její zvýšená exprese je specifická pouze pro karcinom prostaty, a to především u metastatických a high‑risk lokalizovaných nádorů.
Molekula PCA3 je lokalizována v oblasti 9q21-22 [55]. Její přesná funkce v jádře není dosud známa, ačkoli existuje hypotéza, že má regulační funkci v rámci exprese nebo dělení genů [58]. U většiny pacientů s karcinomem prostaty je PCA3 vysoce exprimována, zatímco u zdravých jedinců a pacientů s nemaligními onemocněními prostaty jsou hodnoty PCA3 nízké [55,59]. V případě jiných typů nádorů byla analýza exprese PCA3 negativní. Dalo by se tedy říci, že vysoká hladina PCA3 je specifická pouze pro karcinom prostaty, díky čemuž mohl být vyvinut neinvazivní specifický test na přítomnost této molekuly v moči. První verze tohoto testu byla vyvinuta společností DiagnoCure a od roku 2006 je v Evropě schválen Progensa test od společnosti Gen‑Probe.
Detailnější informace ohledně studií jednotlivých molekul lncRNA jsou zahrnuty v tab. 2.
Kolorektální karcinom
CRC je nejčastější maligní nádorové onemocnění tlustého střeva a konečníku nejen v ČR, ale i ve světě. Každý rok je CRC nově diagnostikován u 8 000 lidí, kdy až polovina z nich na toto onemocnění umírá [51]. I přes vysoký výskyt tohoto nemocnění je stále velmi obtížné určit, které faktory jsou pro vývoj CRC nejpodstatnější [42].
Většina případů CRC se objevuje sporadicky a vyvíjí se pomalu z prekancerózních benigních polypů na maligní adenokarcinom a v nejhorším případě vede až ke vzniku vzdálených metastáz, které jsou fatální až pro 20 % pacientů s CRC. Pro přesnou diagnostiku a adekvátní léčbu je důležitá identifikace molekul odpovědných za nádorovou progresi [60]. Pro predikci metastáz bylo určeno a ověřeno jen několik málo genů, které by mohly představovat vhodné biomarkery [61]. Z těchto důvodů je nezbytné pokračovat v hledání nových diagnostických, prognostických a prediktivních markerů.
Do popředí zájmu vědců se dostávají molekuly LOC285194 a PRNCR1 (prostate cancer non‑coding RNA 1) [39,60,62].
Gen pro LOC285194 je lokalizován v oblasti 3q13.31 [60]. Hladina jeho exprese je významně nižší u nádorové tkáně ve srovnání s přilehlou normální tkání, což naznačuje roli nádorového supresoru. Analýzy ukázaly, že nízká exprese LOC285194 koreluje s některými klinicko‑patologickými faktory, např. s větší velikostí nádoru, s vyšším stadiem nemoci a s přítomností vzdálených metastáz. Pacienti s nízkou hladinou LOC285194 vykazovali kratší dobu přežití. Hladina exprese LOC289154, histologický stupeň, hloubka nádoru, vznik metastáz, cévní a nervová invaze a stupeň progrese nádoru se ukázaly jako statisticky významné prognostické faktory. Nicméně vztah mezi expresí LOC289154 a vývojem a/ nebo progresí CRC zatím nebyl zcela objasněn.
Molekula PRNCR1 je lncRNA, která je spojována především s karcinomem prostaty [39]. Tato molekula je lokalizována v oblasti 8q2 [63], kde se nachází několik SNP, které mohou mít vliv na proces sestřihu a stabilitu konformace mRNA. To má za následek modifikaci jejich interagujících partnerů [62] a také možnou souvislost se vznikem rizikového stadia CRC. SNP rs13252298 a rs1456315 jsou spojeny s významně sníženým rizikem CRC, avšak polymorfizmus rs1456315 spolu s rs7007694 a také rs16901946 souvisí s velikostí a mírou diferenciace nádoru [39].
Detailnější informace ohledně studií jednotlivých molekul lncRNA jsou zahrnuty v tab. 2.
Závěr
Přestože dosud byla popsána funkce pouze jen u omezeného množství lncRNA, význam těchto molekul je výrazně patrný. Pozornost vědců se obrací na využití těchto molekul jako potenciálních biologických markerů různých patologických stavů, u kterých dochází k jejich deregulaci. V případě nádorových onemocnění jsou změny exprese lncRNA spojovány se vznikem, progresí, tvorbou metastáz a recidivou onemocnění. V současnosti se do popředí zájmu dostávají cirkulující formy lncRNA nacházejících se v tělních tekutinách člověka (periferní krev, moč).
Doposud nejlépe popsanou lncRNA je molekula PCA3, jež je tkáňově specifická pro prostatickou tkáň a k její zvýšené expresi dochází u nádorově transformovaných buněk prostaty. Na základě získaných dat byl vyvinut komerční test Progensa sloužící k detekci karcinomu prostaty. Právě tento konkrétní příklad poukazuje na to, jak velký diagnostický potenciál tyto molekuly skýtají. Stále je však nezbytné provedení dalších studií vedoucích k odhalení jejich funkcí a tím klinického využití těchto molekul.
Tato práce byla podpořena grantem Ministerstva zdravotnictví AZV 15-29508A.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do biomedicínských časopisů.
RNDr. Sabina Ševčíková, Ph.D.
Babákova myelomová skupina
Ústav patologické fyziologie
LF MU
Kamenice 5, A3
625 00 Brno
e-mail: sevcik@med.muni.cz
Obdrženo: 23. 10. 2015
Přijato: 2. 12. 2015
Sources
1. International Human Genome Sequencing Consortium. Finishing the euchromatic semence of the human genome. Nature 2004; 431(21): 931 – 945.
2. Bertone P, Stolc V, Royce TE et al. Global identification of human transcribed sequences with genome tiling arrays. Science 2004; 306(24): 2242 – 2246.
3. Kapranov P, Cheng J, Dike S et al. RNA maps reveal new RNA classes and a possible fiction for pervasive transcription. Science 2007; 316(8): 1484 – 1488.
4. Wang KC, Chang HY. Molecular mechanisms of long noncoding RNAs. Mol Cell 2011; 43(16): 904 – 914. doi: 10.1016/ j.molcel.2011.08.018.
5. Calore F, Lovat F, Garofalo M. Non ‑ coding RNAs and cancer. Int J Mol Sci 2013; 14(8): 17085 – 17110. doi: 10.3390/ ijms140817085.
6. Ma L, Bajic VB, Zhang Z. On the classification of long non‑coding RNAs. RNA Biol 2013; 10(6): 924 – 933. doi: 10.4161/ rna.24604.
7. Maruyama R, Suzuki H. Long noncoding RNA involvement in cancer. BMB Rep 2012; 45(11): 604 – 611.
8. Ponjavic J, Ponting CP, Lunter G. Functionality or transcriptional noise? Evidence for selection within long noncoding RNAs. Genome Res 2007; 17(5): 556 – 565.
9. Šána J, Faltejsková P, Svoboda M et al. Dlouhé nekódující RNA a jejich význam u nádorových onemocnění. Klin Onkol 2012; 25(4): 246 – 254.
10. Ayers D. Long non‑coding RNAs: novel emergent biomarkers for cancer diagnostics. J Cancer Res Treat 2013; 1(2): 31 – 35.
11. Morceau F, Chateauvieux S, Gaigneaux A et al. Long and short non‑coding RNAs as regulators of hematopoietic differentiation. Int J Mol Sci 2013; 14(7): 14744 – 14770. doi: 10.3390/ ijms140714744.
12. Knauss JL, Sun T. Regulatory mechanisms of long noncoding RNAs in vertebrate central nervous system development and function. Neuroscience 2013; 235(3): 200 – 214. doi: 10.1016/ j.neuroscience.2013.01.022.
13. Ernst C, Morton CC. Identification and function of long non‑coding RNA. Front Cell Neurosci 2013; 7 : 168. doi: 10.3389/ fncel.2013.00168.
14. Li CH, Chen Y. Targeting long non‑coding RNAs in cancer: progress and prospects. Int J Biochem Cell Biol 2013; 45(8): 1895 – 1910. doi: 10.1016/ j.biocel.2013.05.030.
15. Derrien T, Johnson R, Bussotti G et al. The GENCODE v7 catalog of human long noncoding RNAs: analysis of their gene structure, evolution, and expression. Genome Res 2012; 22(9): 1775 – 1789. doi: 10.1101/ gr.132159.111.
16. Esteller M. Non ‑ coding RNAs in human disease. Nature 2011; 12(12): 861 – 874. doi: 10.1038/ nrg3074.
17. Han BW, Chen YQ. Potencial pathological and functional links between long noncoding RNAs and hematopoiesis. Sci Signal 2013; 6(289): re5. doi: 10.1126/ scisignal.2004099.
18. Garitano ‑ Trajola A, Agirre X, Prosper F et al. Long non‑coding RNA in haematological malignancies. Int J Mol Sci 2013; 14(8): 15386 – 1522. doi: 10.3390/ ijms140815386.
19. Alvarez ‑ Dominguez JR, Hu W, Gromatzky AA et al. Long noncoding RNAs during normal and malignit hematopoiesis. Int J Hematol 201; 99(5): 531 – 541. doi: 10.1007/ s12185 ‑ 014 ‑ 1552 ‑ 8.
20. Guttman M, Amit I, Garber M et al. Chromatin signature reveals over a thousand highly conserved large non‑coding RNAs in mammals. Nature 2009; 458(7235): 223 – 227.
21. Wilusz JE, JnBaptiste CK, Lu LY et al. A triple helix stabilizes the 3’ ends of long noncoding RNAs that lack poly(A) tails. Genes Dev 2012; 26(21): 2392 – 2407.
22. Deng G, Sui G. Noncoding RNA in oncogenesis: a new era of identifying key players. Int J Mol Sci 2013; 14(9): 18319 – 18349. doi: 10.3390/ ijms140918319.
23. Wang H, Wang L, Erdjument ‑ Bromage H et al. Role of histone H2A ubiquitination in Polycomb silencing. Nature 2004; 431(7010): 873 – 878.
24. Ørom UA, Derrien T, Beringer M et al. Long noncoding RNAs with enhancer‑like function in human cells. Cell 2010; 143(1): 46 – 58. doi: 10.1016/ j.cell.2010.09.001.
25. Diederichs S. The four dimensions of nocoding RNA conservation. Trends Genet 2014; 30(4): 121 – 123. doi: 10.1016/ j.tig.2014.01.004.
26. Zhang X, Rice K, Wang Y et al. Maternally expressed gene 3 (MEG3) noncoding ribonucleic acid: isoform structure, expression and functions. Endocrinology 2010; 151(3): 939 – 947. doi: 10.1210/ en.2009 ‑ 0657.
27. Dinger ME, Amaral PP, Mercer TR et al. Long noncoding RNAs in mouse embryonic stem cell pluripotency and differentiation. Genome Res 2008; 18(9): 1433 – 1445. doi: 10.1101/ gr.078378.108.
28. Mercer TR, Dinger ME, Mattick JS. Long non‑coding RNAs: insights into functions. Nature 2009; 10(3): 155 – 159. doi: 10.1038/ nrg2521.
29. Pandey RR, Mondal T, Mohammad F et al. Kcnq1ot1 antisense noncoding RNA mediates lineage ‑ specific transcriptional silencing through chromatin‑level regulation. Mol Cell 2008; 32(2): 232 – 246. doi: 10.1016/ j.molcel.2008.08.022.
30. Rinn JL, Kertesz M, Wang JK et al. Functional demarcation of aktive and silent chromatin domains in human HOX loci by noncoding RNAs. Cell 2007; 129(7): 1311 – 1323.
31. Hung T, Chang HY. Long noncoding RNA in genome regulation: prospects and mechanisms. RNA Biol 2010; 7(5): 582 – 585.
32. Huarte M, Guttman M, Feldser D et al. A large intergenic noncoding RNA induced by p53 mediates global gene repression in the p53 response. Cell 2010; 142(3): 409 – 419. doi: 10.1016/ j.cell.2010.06.040.
33. Guttman M, Donaghey J, Carey BW et al. LincRNAs act in the circuitry controlling pluripotency and differentiation. Nature 2011; 477(7364): 295 – 300. doi: 10.1038/ nature10398.
34. Khalil AM, Guttman M, Huarte M et al. Many human large intergenic noncoding RNAs associate with chromatin‑modifying complexes and affect gene expression. Proc Natl Acad Sci U S A 2009; 106(28): 11667 – 11672. doi: 10.1073/ pnas.0904715106.
35. Tsai MC, Manor O, Wan Y et al. Long noncoding RNA as modular scaffold of histone modification complexes. Science 2010; 329(5992): 689 – 693. doi: 10.1126/ science.1192002.
36. Zhao J, Sun BK, Erwin J Aet al. Polycomb proteins targeted by a short repeat RNA to the mouse X chromosome. Science 2008; 322(5902): 750 – 756. doi: 10.1126/ science.1163045.
37. Gibb EA, Vucic EA, Enfield KS et al. Human cancer long non‑coding RNA transcriptomes. PLoS One 2011; 6(10): e25915. doi: 10.1371/ journal.pone.0025915.
38. Guffanti A, Iacono M, Pelucchi P et al. A transcriptional sketch of a primary human breast cancer by 454 deep sequencing. BMC Genomics 2009; 10 : 163. doi: 10.1186/ 1471 ‑ 2164 ‑ 10 ‑ 163.
39. Li L, Sun R, Liang Y et al. Association between polymorphisms in long non‑coding RNA PRNCR1 in 8q24 and risk of colorectal cancer. J Exp Clin Cancer Res 2013; 32 : 104. doi: 10.1186/ 1756 ‑ 9966 ‑ 32 ‑ 104.
40. Barsyte ‑ Lovejoy D, Lau SK, Boutros PC et al. The c ‑ Myc onkogene directly induces the H19 noncoding RNA by allele ‑ specific binding to potentiate tumorigenesis. Cancer Res 2006; 66(10): 5330 – 5337.
41. Gupta RA, Shah N, Wang KC et al. Long non‑coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature 2010; 464(7291): 1071 – 1076. doi: 10.1038/ nature08975.
42. Kogo R, Shimamura T, Mimori K et al. Long noncoding RNA HOTAIR regulates polycomb ‑ dependent chromatin modification and is associated with poor prognosis in colorectal cancers. Cancer Res 2011; 71(20): 6320 – 6326. doi: 10.1158/ 0008 ‑ 5472.CAN ‑ 11 ‑ 1021.
43. Ji P, Diederichs S, Wang W et al. MALAT ‑ 1, a novel noncoding RNA, and thymosin β4 predict metastasis and survival in early‑stage non‑small cell lung cancer. Oncogene 2003; 22(39): 8031 – 8041.
44. Hutchinson JN, Ensminger AW, Clemson CM et al. A screen for nuclear transcript identifies two linked noncoding RNAs associated with SC35 splicing domains. BMC Genomics 2007; 8 : 39.
45. Lin R, Maeda S, Liu C et al. A large noncoding RNA is a marker for murine hepatocellular carcinomas and a spectrum of human carcinomas. Oncogene 2007; 26(6): 851–858.
46. Luo JH, Ren B, Keryanov S et al. Transcriptomic and genomic analysis of human hepatocellular carcinomas and hepatoblastomas. Hepatology 2006; 44(4): 1012 – 1024.
47. Morrison LE, Jewell SS, Usha L et al. Effects of ERBB2 amplicon size and genomic alternations of chromosomes 1, 3, and 10 on patient response to transtuzumab in metastatic breast cancer. Genes Chromosomes Cancer 2007; 46(4): 397 – 405.
48. Smedley D, Sidhar S, Birdsall S et al. Characterization of chromosome 1 abnormalities in malignant melanomas. Genes Chromosomes Cancer 2000; 28(1): 121 – 125.
49. Huarte M, Rinn JL. Large non‑coding RNAs: missing links in cancer? Hum Mol Genet 2010; 19(2): 152 – 161. doi: 10.1093/ hmg/ ddq353.
50. Májek O, Daneš J, Skovajsová M et al (eds). Mamo.cz – Program mamografického screeningu v České republice [online]. Masarykova univerzita, Brno; 2015. [citováno 26. září 2015]. Dostupné z: http://www.mamo.cz/.
51. Dušek L, Mužík J, Kubásek M et al (eds). Epidemiologie zhoubných nádorů v České republice [online]. Masarykova univerzita, Brno; 2005. [citováno 23. září 2015]. Dostupné z: http://www.svod.cz.
52. Askarian ‑ Amiri ME, Crawford J, French JD et al. SNORD ‑ host RNA Zfas1 is a regulator of mammary develop-ment and a potential marker for breast cancer. RNA 2011; 17(5): 878 – 891. doi: 10.1261/ rna.2528811.
53. Cz.movember.com [internetová stránka]. The movember foundation – celosvětová charitativní organizace, Česká republika; [aktualizováno 2015; citováno 26. září 2015]. Dostupné z: http://cz.movember.com/.
54. Prensner JR, Iyer MK, Balbin OA et al. Transcriptome sequencing across a prostate cancer kohort identifies PCAT ‑ 1, an unannotated lincRNA implicated in disease progression. Nat Biotechnol 2011; 29(8): 742 – 749. doi: 10.1038/ nbt.1914.
55. Bussemakers MJ, van Bokhoven A, Verhaegh GW et al. DD3: a new prostate ‑ specific gene, highly overexpressed in prostate cancer. Cancer Res 1999; 59(23): 5975 – 5979.
56. Kleer CG, Cao Q, Varambally S. EZH2 is a marker of aggressive breast cancer and promotes neoplastic transformation of breast epithelial cells. Proc Natl Acad Sci U S A 2003; 100(20): 11606 – 11611.
57. Varambally S, Dhanasekaran SM, Zhou M et al. The polycomb group protein EZH2 is involved in progression of prostate cancer. Nature 2002; 419(6907): 624 – 629.
58. Meng FJ, Shan A, Jin L et al. The expression of a variant prostate ‑ specific antigen in human prostate. Cancer Epidemiol Biomarkers Prev 2002; 11(3): 305 – 309.
59. De Kok JB, Verhaegh GW, Roelofs RW et al. DD3PCA3, a very sensitive and specific marker to detect prostate tumors. Cancer Res 2002; 62(9): 2695 – 2698.
60. Qi P, Xu M, Ni S et al. Low expression of LOC285194 is associated with poor prognosis in colorectal cancer. J Transl Med 2013; 11 : 122. doi: 10.1186/ 1479 ‑ 5876 ‑ 11 ‑ 122.
61. Ge X, Chen Y, Liao X et al. Overexpression of long noncoding RNA PCAT ‑ 1 is a novel biomarker of poor prognosis in patients with colorectal cancer. Med Oncol 2013; 30(32): 588. doi: 10.1007/ s12032 ‑ 013 ‑ 0588 ‑ 6.
62. Chung S, Nakagawa H, Uemura M et al. Association of a novel long non‑coding RNA in 8q24 with prostate cancer susceptibility. Cancer Sci 2011; 102(1): 245 – 252. doi: 10.1111/ j.1349 ‑ 7006.2010.01737.x.
63. Pal P, Xi H, Guha S et al. Common variants in 8q24 are associated with risk for prostate cancer and tumor aggressiveness in men of European ancestry. Prostate 2009; 69(14): 1548 – 1556. doi: 10.1002/pros.20999.
Labels
Paediatric clinical oncology Surgery Clinical oncology
Article was published inClinical Oncology

2016 Issue 1-
All articles in this issue
- Genomic Tests as Predictors of Breast Cancer Patients’ Prognosis
- Potential of Long Non- coding RNA Molecules in Diagnosis of Tumors
- Survival Analysis Three-year Follow-up of Pacients with Head and Neck Cancer
- Successful Therapy of Czech Patients with ROS1 Translocation by Crizotinib
- The Role of Heat Shock Proteins in Leukemia
- Predicting Vitality Change in Older Breast Cancer Survivors after Primary Treatment – an Approach Based on Using Time-related Difference of Pro-inflammatory Marker C-reactive Protein
- Successful Associating Liver Partition and Portal Vein Ligation after Unsuccessful Double TACE Procedure Complicated with Sepsis and Pancreatitis
- Clinical Oncology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Survival Analysis Three-year Follow-up of Pacients with Head and Neck Cancer
- Genomic Tests as Predictors of Breast Cancer Patients’ Prognosis
- The Role of Heat Shock Proteins in Leukemia
- Potential of Long Non- coding RNA Molecules in Diagnosis of Tumors
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

