-
Medical journals
- Career
Retrospektivní NGS studie u vysoce rizikových pacientů s hereditární predispozicí k nádorovému onemocnění v Masarykově onkologickém ústavu
Authors: E. Macháčková; J. Hazova; E. Sťahlová Hrabincová; P. Vašíčková; M. Navratilova; M. Svoboda; L. Foretová
Authors‘ workplace: Oddělení epidemiologie a genetiky nádorů, Masarykův onkologický ústav, Brno
Published in: Klin Onkol 2016; 29(Supplementum 1): 35-45
Category: Original Articles
doi: https://doi.org/10.14735/amko2016S35Overview
Východiska:
V současné době bylo popsáno již více než 200 nádorových syndromů. Ve většině populací jsou však dostupné pouze informace o mutačním spektru ve vysoce rizikových genech limitované počtem vyšetřených jedinců.Metody:
V rámci retrospektivní NGS studie v Masarykově onkologickém ústavu bylo provedeno vyšetření TruSight Cancer panelem zahrnujícím 94 genů u 50 vysoce rizikových jedinců se závažnou osobní i rodinnou anamnézou onkologického onemocnění bez detekované kauzální mutace v genech BRCA1, BRCA2, MLH1, MSH2, MSH6, TP53 nebo APC dle indikace. Všechny patogenní nebo pravděpodobně patogenní mutace detekované pomocí NGS technologie byly potvrzeny Sangerovým sekvenováním.Výsledky:
Ztrátové (frame-shift, nonsense) mutace byly detekovány v genech ATM, BAP1, FANCC, FANCI, PMS2, SBDS, ERCC2, RECQL4. Několik patogenních nebo pravděpodobně patogenních mutací (missense, predikované sestřihové mutace, in-frame delece/ inzerce) bylo zachyceno v genech ATM, BRIP1, CDH1, CHEK2, ERCC2, ERCC3, ERCC4, FANCA, MC1R, MEN1, MRE11A, MUTYH, PALB2, RAD51C, RET, SDHB, STK11. Nacházejí se ve vysoce konzervovaných funkčních doménách proteinů a některé z nich již byly prokázány jako patogenní mutace pomocí funkčních testů nebo u závažných autozomálně recesivních syndromů (Ataxia telangiectasia, Fanconiho anémie). Většina z detekovaných missense variant v řadě dalších genů je nejasného klinického významu a determinace jejich významnosti zůstává otevřená do budoucna.Závěr:
Detekce variant se střední nebo nízkou penetrancí má pouze limitovanou klinickou využitelnost. Panelové testování u vysoce rizikových osob s nádorovým onemocněním může poskytnout důležitou informaci o příčině nádorové predispozice a může pomoci s výběrem optimální léčby a v preventivní personalizované onkologii.Klíčová slova:
hereditární nádorové syndromy – hereditární nádor prsu a ovaria – hereditární nepolypózní kolorektální karcinom – sekvenování nové generace – TruSight cancer panel – MiSeqÚvod
Během posledních 20 let došlo k výraznému rozvoji v oblasti vyšetření genetické predispozice k nádorovým onemocněním. Na základě genetických vyšetření byla do klinické praxe zavedena léčebná a preventivní doporučení (guidelines) vedoucí ke snížení rizika a časnému záchytu onkologického onemocnění u osob s výraznou genetickou predispozicí s detekovanou mutací ve vysoce penetrantním genu: např. Li-Fraumeni syndrom (mutace v genu TP53), Lynchův syndrom (mutace v některém z „mismatch“ reparačních genů –MLH1, MSH2, MSH6, PMS2 – u dědičného nepolypózního kolorektálního karcinomu), dědičná forma nádoru prsu a ovaria (mutace v genech BRCA1, BRCA2).
V současné době již bylo popsáno více než 200 různých nádorových syndromů (OMIM databáze), ale molekulárně genetické vyšetření bylo do současné doby dostupné pouze pro nejznámější vysoce rizikové geny.
Pokroky v sekvenační technologii – tzv. sekvenování nové generace (next-generation sequencing – NGS) – umožnily masivní testování sekvence desítek až stovek genů současně, což umožňuje testování známých i potenciálně rizikových genů, které jsou součástí signálních a reparačních drah významných pro nádorovou predispozici. Výrazné rozšíření spektra analyzovaných genů vede nejen k identifikaci kauzálních mutací ve vysoce rizikových genech, ale také k detekci celé řady mutací s dosud nedeterminovanou mírou rizika nebo k detekci středně rizikových variant, které ovšem mohou ve vzájemných kombinacích výrazně navyšovat relativní riziko nádorové predispozice v postižených rodinách.
V současné době jsou nabízeny k vyšetření různé multigenové komerční panely cílené na predikci rizika ke vzniku nádorového onemocnění. K těm rozsáhlejším patří TruSight Cancer Target Genes panel (Illumina), který je jedním z nejčastěji používaných komerčních panelů v laboratořích západní Evropy. TruSight Cancer panel byl vyvinut ve spolupráci Illuminy s týmem profesorky Nazneen Rahman (Institute of Cancer Research ICR, London) v roce 2012 (http://www.illumina.com/products/trusight_cancer.html). Panel je cílen na sekvence 94 genů a 284 SNPs (singe nucleotide polymorphims), které byly v předchozích letech asociovány s hereditární predispozicí k nádorovému onemocnění.
Součástí TruSight Cancer panelu (http:/ / www.illumina.com/ products/ trusight_cancer.html) jsou proby pro analýzu těchto genů: AIP, ALK, APC, ATM, BAP1, BLM, BMPR1A, BRCA1, BRCA2, BRIP1, BUB1B, CDC73, CDH1, CDK4, CDKN1C, CDKN2A, CEBPA, CEP57, CHEK2, CYLD, DDB2, DICER1, DIS3L2, EGFR, EPCAM, ERCC2, ERCC3, ERCC4, ERCC5, EXT1, EXT2, EZH2, FANCA, FANCB, FANCC, FANCD2, FANCE, FANCF, FANCG, FANCI, FANCL, FANCM, FH, FLCN, GATA2, GPC3, HNF1A, HRAS, KIT, MAX, MEN1, MET, MLH1, MSH2, MSH6, MUTYH, NBN, NF1, NF2, NSD1, PALB2, PHOX2B, PMS1, PMS2, PRF1, PRKAR1A, PTCH1, PTEN, RAD51C, RAD51D, RB1, RECQL4, RET, RHBDF2, RUNX1, SBDS, SDHAF2, SDHB, SDHC, SDHD, SLX4, SMAD4, SMARCB1, STK11, SUFU, TMEM127, TP53, TSC1, TSC2, VHL, WRN, WT1, XPA, XPC.
V naší retrospektivní studii jsme se zaměřili na otestování komerčně dostupného TruSight Cancer panelu a možnost jeho využití pro odhalení příčiny nádorové predispozice u dříve neobjasněných retrospektivních případů.
Soubor pacientů
Pilotní retrospektivní studie byla provedena u 50 vzorků genomové DNA izolované z periferní krve. DNA byla získána od pacientů s vysoce rizikovou osobní a rodinnou anamnézou výskytu onkologického onemocnění s různými druhy onkologických diagnóz, u kterých nebyla nalezena riziková mutace v rutinně analyzovaných vysoce rizikových genech. Většina z těchto vybraných pacientů byla již dříve vyšetřena na přítomnost mutací v genech BRCA1, BRCA2 (hereditární syndrom nádoru prsu a ovaria – 28 pacientů), MLH1, MSH2, MSH6 nebo APC (hereditární syndrom nepolypózního nebo polypózního kolorektálního karcinomu – 10 pacientů) nebo TP53 (Li-Fraumeni syndrom), případně na jiné vzácné, s vysokou pravděpodobností hereditární nádorové syndromy bez odhalené příčiny nádorové predispozice (nádory ledvin, mozkové nádory, maligní melanom atd.).
Všichni pacienti zařazení do retrospektivní studie souhlasili s vyšetřením genů, které mohou způsobit predispozici k onkologickému onemocnění, podepsali informovaný souhlas s vyšetřením a souhlasili také s anonymním využitím DNA k lékařskému výzkumu.
Metody
Vyšetření bylo provedeno pomocí Tru-Sight Cancer Target Genes panelu. Principem postupu je enzymatická fragmentace (tagmentace) 50 ng genomové DNA → indexování a pool knihovny → enrichment/ hybridizační (TruSight Rapid Capture protocol) postup s ≈ 4 000 prób cílených na → 1 700 exonů 94 nádorově predispozičních genů (255 Kb cílových sekvencí). Sekvenační běh připravené knihovny (s MiSeq Reagent kit V2) na MiSeq systému (Illumina) → analýza pomocí MiSeq reporter software → anotace VCF souborů pomocí Illumina VariantStudio (www.illumina.com/content/dam/illumina-marketing/documents/products/datasheets/datasheet_trusight_cancer.pdf).
Výběr patogenních a potenciálně patogenních variant (predikce významnosti dle Align-GVGD, PolyPhen-2, SIFT programů u missense variant; predikce ovlivnění sestřihu dle GeneSplicer, NNSPLICE; BDGP Splice Site Prediction) → kontrola správnosti HGVS nomenklatury (www.hgvs.org) u in/del variant → kontrola a případně převedení nálezu na referenční sekvenci doporučenou v příslušných mezinárodních mutačních databázích (HGMD, LOVD, IARC atd.).
Navolení primerů k analýze sledovaných variant pomocí LightScanner Primer Design Software (Idaho Technology Inc.), optimalizace PCR (polymerase chain reaction) podmínek a PCR amplifikace sledované oblasti. Potvrzení přítomnosti významných (patogenních a potenciálně patogenních vhodných k segregační analýze) nálezů pomocí Sangerova sekvenování na 3130 Genetic Analyser (Applied Biosystems) a jeho ověření u nezávisle izolovaného vzorku DNA.
Segregační analýza u rodin zatížených patogenní nebo potenciálně patogenní variantou.
V případě potvrzování nálezů u genů s přítomností pseudogenů byla provedena dvoukolová „nested“ PCR vycházející z publikovaných primerů, kde autoři deklarovali amplifikaci specifických produktů bez přítomnosti pseudogenů. Pro analýzu genu PMS2 byly použity primery pro Long-Range PCR (s použitím Expand Long Template PCR system, Roche) a vnitřní primery dle Vaughna et al [1]. Pro analýzu exonu 2. genu SBDS byly použity vnější primery dle Kawakamiho et al [2] a vnitřní primery byly navoleny softwarem LightScanner Primer Design (Idaho Technology Inc.).
Výsledky a vybrané kazuistiky
Téměř u poloviny z vyšetřených vzorků DNA byly detekovány patogenní nebo možné/ pravděpodobně patogenní (IARC klasifikace UV-4: „likely pathogenic“) mutace v některém z 94 analyzovaných genů.
Ve skupině 28 pacientů s podezřením na dědičnou predispozici nádoru prsu a/nebo ovaria (dříve indikováni k analýze v genech BRCA1, BRCA2 nebo v některých případech také k analýze genu TP53 bez detekované kauzální mutace) byly nejčastěji detekovány mutace v dráhách reparace dvouřetězcových zlomů (geny ATM, CHEK2) a ICL (interstrand cross-links) reparace křížových vazeb v DNA (Fanconi anemia geny – FANC geny) spřažené s homologní rekombinací (geny BRIP1, RAD51C, PALB2). Další dráhou s opakovanými záchyty pravděpodobně významných nálezů byla dráha nukleotidové excizní reparace (NER – geny ERCC2, ERCC3) a geny z rodiny RecQ helikáz (RECQL4, WRN), které se účastní DNA reparačních procesů. Souhrnné výsledky nálezů u skupiny s podezřením na dědičnou predispozici nádoru prsu a/ nebo ovaria jsou uvedeny v tab. 1. Ztrátové (nonsense, frame-shift) mutace byly u této skupiny vyšetřených detekovány v genech: ATM, FANCI, FANCC, RECQL4 a SBDS.
Table 1. Nálezy u skupiny vyšetřených s převážně nádory prsu, ovaria, dělohy, štítné žlázy a nádory prostaty (28 vyšetřených jedinců). 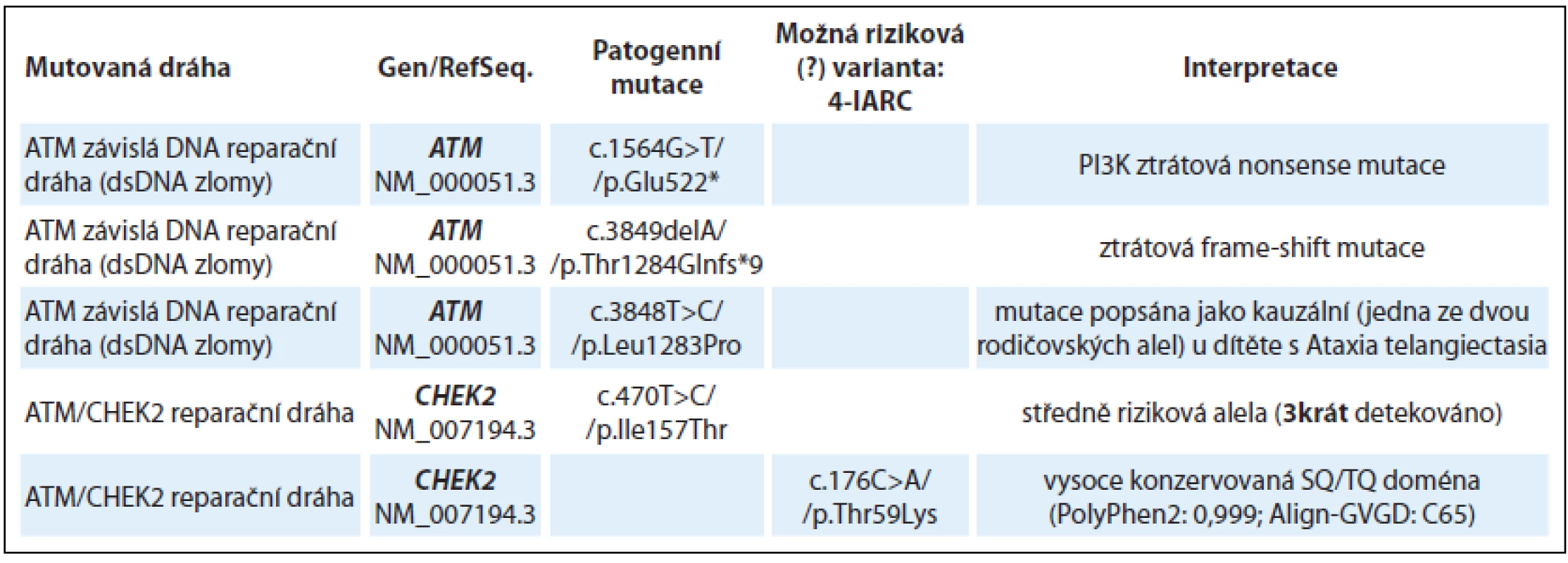
Tab. 1 – pokračování. Nálezy u skupiny vyšetřených s převážně nádory prsu, ovaria, dělohy, štítné žlázy a nádory prostaty (28 vyšetřených jedinců). 
AA – aminokyselina Patogenní missense mutace byly detekovány v genech FANCA, BRIP1 a dále bylo zachyceno několik středně rizikových (≈ 2násobné zvýšení rizika) missense mutací v CHEK2 genu a MC1R receptoru.
Nově detekovaným nálezem je missense mutace v genu BRIP1 (synonymum FANCJ, BACH1) p.Cys350Tyr u ženy s karcinomem prsu (kazuistika 1). BRIP1 missense mutace p.Cys350Tyr je lokalizována v jednom ze čtyř vazebných cysteinů Fe-S domény a je naprosto ztrátová pro helikázovou funkci BRIP1 [3]. BRIP1 helikáza interaguje s komplexem BRCA1/ BRCA2/ RAD51/ PALB2 a je nezbytná pro reparaci dsDNA zlomů a homologní rekombinaci. BRIP1 mutace p.Cys350Tyr zatím nebyla popsána germinálně, ale ztrátové mutace v genu BRIP1 jsou asociovány s predispozicí k nádorům prsu [4].
Kazuistika 1. Kazuistika pacientky s <em>BRIP1</em> mutací. 
U některých jedinců bylo nalezeno několik patogenních a potenciálně patogenních variant, zatímco u jiných byly přítomny pouze běžné (nízkorizikové) nálezy.
U pacientky (ročník 1977) s nádorem ovaria ve 29 letech a pozdější diagnózou nádoru štítné žlázy byla detekována ztrátová mutace v genu ATM c.3849delA, přičemž nádory vaječníků nepatří do typického spektra nádorů u ztrátových ATM mutací. U této pacientky však byla zároveň zachycena missense mutace v genu RAD51C p.Thr287Ala, která byla popsána jako riziková mutace na základě funkčního testu [5,6]. RAD51C je protein funkční v procesech homologní rekombinace a DNA reparace spřažené s homologní rekombinací. Patogenní mutace v genu RAD51C/ FANCO jsou popisovány především v souvislosti s HBOC fenotypem v postižených rodinách a v případě homozygotní nebo bialelické mutace s projevy recesivního syndromu Fanconiho anémie (FA) [7]. A právě mutace v genu RAD51C jsou častěji přítomny u ovariálních nádorů a mohlo by se v tomto případě jednat o významný modifikační faktor. Analýza probandek s karcinomem ovaria z HOC a HBOC rodin v ČR prokázala přítomnost patogenních mutací v genech RAD51C a RAD51D u 3 % vyšetřovaných [8].
Jiným příkladem vícečetných nálezů je žena (ročník 1970) s nádorem prsu v 32 letech s missense mutací ve vysoce konzervované doméně genu ATM p.Leu1283Pro, která byla již dříve zachycena jako jedna z rodičovských alel u dítěte s recesivním syndromem Ataxia Telangiectasia (Ataxia-Telangiectasia databáze). Jako další riziková mutace byla u této ženy missense mutace v genu FANCA p.Arg517Trp, která se opět nachází ve vysoce konzervované doméně a byla zachycena germinálně jako druhá alelická mutace v německé populaci u ženy s relativně pozdními projevy FA (AML po 40. roce věku) a s nádorem prsu ve 37 letech [9].
U pacientky s nádorem slinivky (ročník 1992) s rodinnou anamnézou karcinomu prsu a karcinomu slinivky byla zachycena nonsense mutace v genu FANCI p.Arg1285* (kazuistika 2). Autozomálně recesivní syndrom FA je způsoben bi-alelickými mutacemi v FA genech. Relativní riziko u heterozygotních nosičů mutace v některém z FANC genů je značně variabilní [10]. Mutace v genu FANCI jsou celosvětově velmi vzácné, riziko solidních nádorů u heterozygotních nosičů ve FANCI genu není známo (LOVD Fanconi anemia database [11]).
Kazuistika 2. Kazuistika pacientky s <em>FANCI</em> mutací. 
Pacienti s podezřením na HNPCC nebo FAP
Ve skupině 10 pacientů s podezřením na dědičnou predispozici nádoru k nádoru kolorekta bez detekované kauzální mutace v genech MLH1, MSH2, MSH6 (nepolypózní kolorektální karcinom) nebo APC (familiární polypózní syndrom) byly detekovány patogenní mutace v reparační dráze bázové excizní reparace BER (gen MUTYH způsobující autozomálně recesivní formu polypózy), středně rizikové mutace v genu CHEK2 a „in-frame“ delece v genu MEN1 c.809_817del, která již byla popsána u jedinců s MEN1 syndromem [12] a v naší studii byla zachycena u mladého muže (ročník 1986) s rodinnou anamnézou nádoru kolorekta, gastrinomu a nádoru žaludku. Souhrnné výsledky nálezů u skupiny s podezřením na dědičnou predispozici k nádorům GIT jsou uvedeny v tab. 2. Je zde zařazena také žena (ročník 1946) s triplicitou: duplicitní nádor prsu (52, 62 let) s adenokarcinomem tlustého střeva a maligním melanomem, která byla jako jediná zařazena v obou výše zmíněných skupinách. U pacientky byla detekována frame-shift mutace v genu FANCC c.455dupA (tab. 1) spolu s pravděpodobně patogenní in/ del mutací v genu STK11 (synonymum LKB1) c.740_743delinsTCAC/p.Asn247_Ile248delinsIleThr nacházející se ve vysoce konzervované katalytické doméně STK11 kinázy (tab. 2). V tomto případě můžeme spekulovat, že FANCC mutace je pravděpodobným rizikovým faktorem predispozice k nádoru prsu a STK11 mutace ke vzniku adenokarcinomu.
Table 2. Nálezy u skupiny vyšetřených s převážně kolorektálními nádory polypózními i nepolypózními a nádory GIT (10 vyšetřených jedinců). 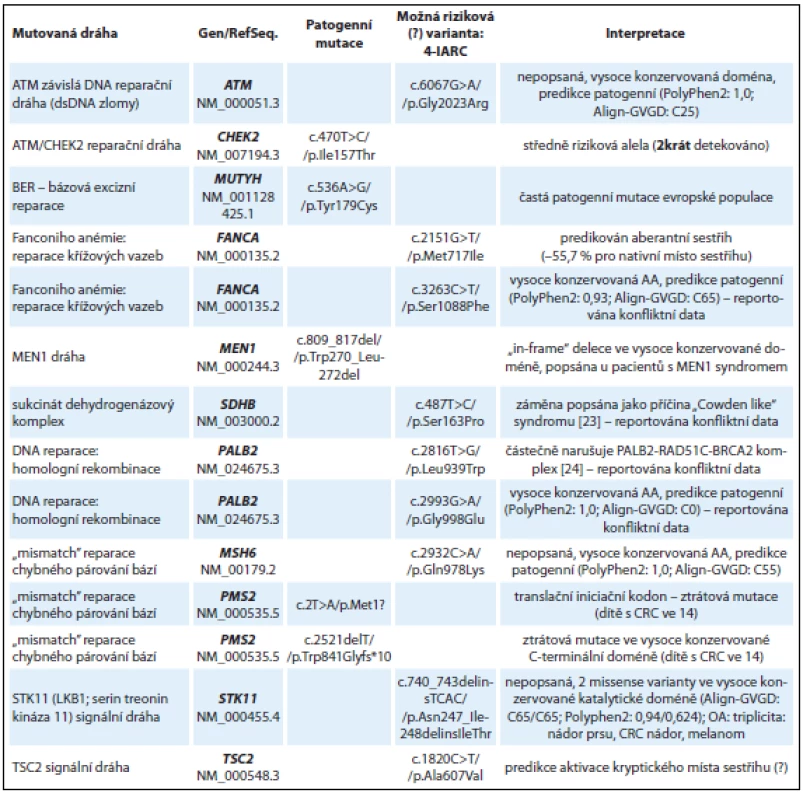
AA – aminokyselina U dítěte (chlapec, ročník 1999) diagnostikovaného s kolorektálním karcinomem ve 14 letech byly detekovány dvě patogenní mutace v genu PMS2 (kazuistika 3). Inaktivující PMS2 mutace c.2T>A v iniciačním kodonu pro metionin p.Met1? je patogenní mutací znemožňující syntézu proteinu. Druhou detekovanou PMS2 mutací je c.2521delT, která způsobuje posun čtecího rámce (frame-shift) a předčasnou terminaci translace v C-terminální dimerizační doméně PMS2 proteinu, která je klíčová pro vazbu s MLH1 proteinem [13]. Bialelické mutace v PMS2 genu způsobují závažnou MMR deficienci s manifestací intestinálních nádorů v dětském věku [14].
Kazuistika 3. Kazuistika pacienta s bialelickou <em>PMS2</em> mutací. 
Pacienti se sarkomy nebo nádory mozku
V tab. 3 je uveden souhrn nálezů u skupiny pěti pacientů s diagnózami sarkomu nebo nádory mozku bez detekované mutace v genu TP53. Bezpochyby významným nálezem je missense mutace v genu ATM p.Asn2875Ser, kde Asn2875 je katalytickou aminokyselinou v aktivním místě PI3K domény, a bylo experimentálně prokázáno, že substituce za jinou aminokyselinu (Asn > Lys, Asn > Ser) inaktivuje kinázovou aktivitu ATM [15,16]. ATM mutace p.Asn2875Ser zatím nebyla germinálně popsána (kazuistika 4).
Table 3. Nálezy u skupiny vyšetřených s převážně sarkomy nebo nádory mozku (5 vyšetřených jedinců). 
AA – aminokyselina Kazuistika 4. Kazuistika pacienta s <em>ATM</em> mutací. 
Pacienti s různými typy nádorů
V tab. 4 jsou uvedeny souhrnné výsledky u osmi pacientů s různými druhy nádorů s pravděpodobnou genetickou predispozicí, kteří již byli dříve vyšetřeni na mutace v různých genech (dle diagnózy TP53, MLH1, MSH2, MSH6, FLCN, VHL, RB1) bez nálezu kauzální patogenní mutace.
Table 4. Nálezy u skupiny vyšetřených s různými druhy nádorů s pravděpodobnou dědičnou predispozicí (8 vyšetřených jedinců). 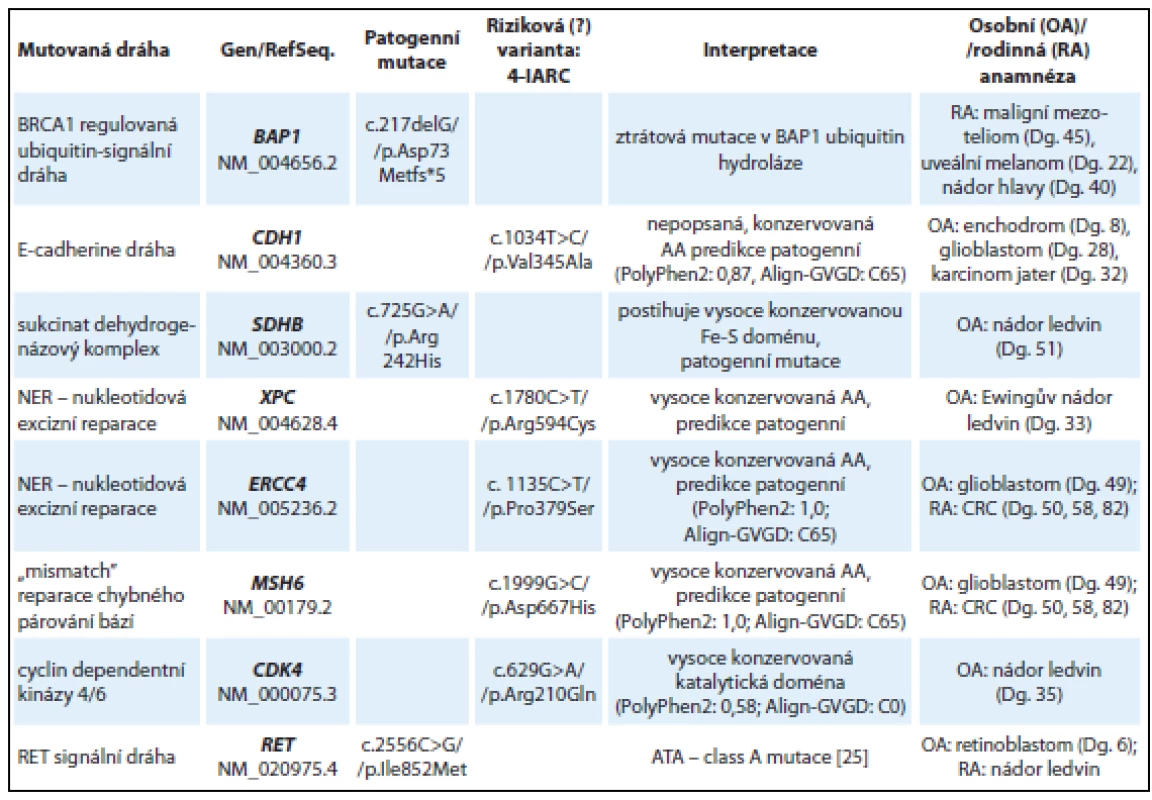
AA – aminokyselina U mladé ženy s rodinnou anamnézou maligního mezoteliomu a uveálního melanomu byla detekována frame-shift mutace v genu BAP1: c.217delG (kazuistika 5). Gen BAP1 kóduje ubikvitin-karboxyterminální hydrolázu, která se nachází v komplexu s BRCA1 proteinem a účastní se DNA reparačních procesů, regulace transkripce a buněčné proliferace. Mutace v genu BAP1 byly popsány u autozomálně dominantního nádorového syndromu, který je asociován především s výskytem maligního mezoteliomu a uveálního melanomu [17].
Kazuistika 5. Kazuistika pacientky s <em>BAP1</em> mutací. 
U pacienta s karcinomem ledviny v 51 letech byla detekována patogenní missense mutace v genu SDHB p.Arg242His, která se nachází ve vysoce konzervované Fe-S doméně SDHB proteinu. Jedná se o již dříve popsanou mutaci u pacientů s diagnózou feochromocytomu, paragangliomu a GIT stromálních tumorů (LOVD databáze, [18]).
Při analýze 94 genů je u každého jedince detekováno také velké množství variant nejasného významu (IARC klasifikace UV-3, nejasného významu – neklasifikované [19]) a frekvenčně častých nízko rizikových polymorfních variant. Tyto varianty však mají minimální klinickou využitelnost [20] a v tomto stručném sdělení nebudou rozebírány.
Diskuze
Technologie NGS v současné době přechází z výzkumu do diagnostické praxe. Rutinní klinické využití vyžaduje vysokou senzitivitu, vysokou specificitu, pokud možno jednoduchost přípravy sekvenačních knihoven, cenovou přijatelnost a uživatelsky zvládnutelnou analýzu dat.
Genetické testy by měly být hodnoceny na základě analytické validity, klinické validity a klinické využitelnosti [20].
Analytická validita vyjadřuje stupeň citlivosti a specificity testu, zda správně vyhodnotí přítomnost nebo nepřítomnost mutace (varianty) ve vyšetřovaném genu. Nezbytné je dostatečné pokrytí (min. 50krát v případě germinálních mutací) kódujících oblastí analyzovaných genů včetně sestřihových míst v intronech (min. ± 20 bp). S klesajícím pokrytím narůstá množství falešně pozitivních záchytů.
Při detekci patogenní germinální mutace v případě, že tyto výsledky mají být použity ke klinickým účelům, je nezbytný průkaz její přítomnosti Sangerovým sekvenováním a potvrzení z nezávisle izolovaného vzorku DNA (nejlépe z nového odběru). To zahrnuje navolení primerů specifických pro analyzovanou oblast, optimalizaci podmínek amplifikace a sekvenování „klasickou“ Sangerovou metodou. Tyto primery jsou pak dále využity pro segregační analýzu v postižené rodině.
V případě detekce missense variant s „pouze“ predikovaným patogenním efektem jsou zapotřebí další analýzy, jako je segregační analýza, funkční testy zaměřené na funkční domény s přítomností detekované varianty, analýza sestřihu na mRNA úrovni u predikovaných sestřihových mutací.
U GC bohatých oblastí často dochází k chybnému alignmentu (mapování na referenční sekvenci) až k úplnému výpadku analyzovaných sekvencí (V TruSight Cancer panelu je to markantní např. u genů PHOX2B nebo CDKN1C). Je proto nezbytné kontrolovat nejen detekované varianty, ale je třeba brát v úvahu také nepokryté úseky analyzovaných genů a v případě indikace vyšetření těchto genů, doplnit vyšetření chybějících oblastí alternativní metodou.
Z našich zkušeností jsou NGS technologie méně citlivé na rozsáhlejší delece/ duplikace přesahující cca 10 párů bází. Obecně ale nelze definovat pouze rozsah, protože záleží na sekvenci analyzovaného úseků a lokalizaci delece/inzerce vzhledem k pozici oligonukleotidů hybridizujících na cílené sekvence (u enrichment/ hybridizačního postupu). Se zvyšujícím se rozsahem delece/ duplikace klesá podíl zastoupení alely s delecí v celkovém pokrytí. Enrichment/hybridizační postup je výrazně specifičtější než amplifikační příprava knihovny a omezuje zanášení chyb v průběhu amplifikace [21].
Další problematickou oblastí jsou geny s pseudogeny. V analyzovaném souboru jsme řešili průkaz detekované přítomnosti mutace v genech PMS2 a SBDS. V těchto případech není možná přímá amplifikace analyzovaných úseků z důvodů vysoké homologie mezi genem a jeho pseudogeny. V případě genu PMS2 jsme při potvrzení obou mutací mohli vyjít z publikovaného zdroje a optimalizovat „nested“ PCR s publikovanými primery [1]. V případě nonsense mutace v genu SBDS c.184A>T/p.Lys62* byla situace složitější. Homologie mezi SBDS genem a pseudogenem SBDSP1 je 97 % a navíc v pozici 184 pseudogenu je T jako standardní nukleotid (SBDSP1: NR_024110.1: n.647T; g.72301327T). Již při prvotní přímé amplifikaci exonu 2 bylo ze srovnání vzorku pacientky a několika kontrolních vzorků patrné, že zastoupení c.184A>T odpovídá 50 % u kontrolních vzorků a cca 75 % u vzorku pacientky a zároveň byly ve všech vzorcích přítomny čtyři další varianty odpovídající sekvenci SBDSP1 pseudogenu. Ke specifické amplifikaci exonu 2 jsme použili publikované vnější primery [2] a vnitřní primery pro „nested“ PCR již byly navoleny softwarem LightScanner Primer Design (Idaho Technology Inc.). Tímto způsobem bylo možné získat produkt specifický pouze pro exon 2 SBDS genu bez sekvence pseudogenu.
Klinická validita zahrnuje hodnocení významnosti detekované varianty a odhad míry rizika pro nosiče této varianty. Odhad míry rizika genetických nálezů je klíčový pro klinickou využitelnost panelového testování, které by mělo vést k individuálnímu doporučení pro plán preventivního sledování, cílené medikaci nebo chirurgickým profylaktickým zákrokům, které mohou riziko nádorového onemocnění snížit.
Abychom mohli stanovit klinickou validitu, musíme znát odpověď na tyto základní otázky [20]:
- Jsou mutace v konkrétním genu asociovány s nádorovou predispozicí?
- Které varianty nebo skupiny variant jsou rizikové mutace a pro které typy nádorů?
- Jaká je výše nádorové predispozice vzhledem k incidenci onemocnění?
V oblasti klinického hodnocení je nutné vycházet z výsledků biologického hodnocení nalezené varianty, ze segregačních studií a z dosavadních klinických znalostí o uvedeném genu. U nosičů vysoce rizikových patogenních mutací v genech velkého účinku (vyšší než čtyřnásobné riziko vzhledem k běžné populaci: např. mutace v genech TP53, BRCA1, BRCA2, CDH1, MLH1, PTEN, APC atd.) lze navrhnout dispenzarizaci nosičů dle platných doporučení. V případě neznámé klinické validity není vhodné nabízet prediktivní testování zdravých příbuzných, ale odhadnout riziko a navrhnout prevenci dle rodinné anamnézy.
Pro řadu genů z TruSight panelu ale zatím nebyla doporučení stanovena a míra relativního rizika pro heterozygotní nosiče mutací je nejasná.
Dispenzarizace u středně rizikových genů většinou zatím nebyla navržena ani publikována. Incidence středního rizika je uváděna jako cca 2–4násobná v porovnání s rizikem průměrné populace [22]. Kumulací získaných dat (v rámci multicentrických studií) o středně rizikových genech je možné postupně odhalit předpokládanou výši a spektrum nádorových rizik a navrhnout rozsah preventivní péče. Zatím je možné se opřít o publikované informace jednotlivých studií, a především o empirická rizika v testované rodině.
Odhad rizika založený na analýzách v přísně selektované vysoce rizikové skupině rodinných případů nemusí vždy odrážet správnou hodnotu průměrného rizika u nosičů v běžné populaci [20]. V současné době chybí pro naši populaci srovnávací výsledky analýz v dostatečně velké kontrolní skupině – zdravé věkově starší populaci bez nádorové predispozice v osobní nebo rodinné anamnéze.
Detekce nízkorizikových mutací a častých polymorfních variant, u kterých je uváděna spíše modifikace rizika, nemají z klinického hlediska uplatnění. Na velkých genových panelech jsou jich detekovány desítky a jsou s podobnou frekvencí detekovány také u kontrolních vzorků.
U nosičů patogenních mutací v genech pro známé recesivní syndromy (FA, AT, XP, aj.) je vhodné zvážit podání informace o mutaci z důvodů možného výskytu tohoto syndromu v dalších generacích. Frekvence nosičství mutací v těchto genech je však velmi vzácná a pravděpodobnost autozomálně recesiního syndromu v dalších generacích je nízká. Proto dosud není jasný přístup k této problematice a je vhodné postupovat individuálně.
Závěr
Téměř u poloviny z vyšetřených jedinců byly detekovány patogenní nebo pravděpodobně patogenní (IARC-4) mutace v některém z analyzovaných genů. Ztrátové (nonsense, frame-shift) mutace byly detekovány v genech: ATM, BAP1, FANCI, FANCC, PMS2, RECQL4, SBDS. Dále bylo nalezeno několik s vysokou pravděpodobností patogenních missense variant nebo „in-frame inzercí/delecí v genech: ATM, BRIP1, CDH1, CHEK2, ERCC2, ERCC3, ERCC4, FANCA, MC1R, MEN1, MRE11A, MUTYH, PALB2, RAD51C, RET, SDHB, STK11, které se nacházejí ve vysoce konzervovaných funkčních doménách proteinu a některé z nich již byly prokázány jako patogenní mutace pomocí funkčních testů nebo u závažných autozomálně recesivních syndromů (Ataxia telangiectasia, FA).
Pro klinické využití je nezbytné stanovení relativního rizika, což je v řadě případů ve fázi výzkumných studií. Detekce variant s nízkou penetrancí má pouze limitovanou klinickou využitelnost. Rozšíření spektra analyzovaných genů představuje výzvu pro zajištění odpovídajícího preventivního sledování a léčebných postupů u osob v riziku. Je zapotřebí úzká spolupráce onkologů, klinických genetiků, molekulárních biologů a bioinformatiků, aby bylo možné plně využít potenciál NGS technologií v běžné diagnostické praxi a preventivní personalizované onkologii.
V oblasti klinického hodnocení je nutné vycházet z výsledků biologického hodnocení nalezené varianty, ze segregační studie a z dosavadních klinických znalostí o uvedeném genu. V případě neznámé klinické validity není vhodné nabízet prediktivní testování zdravých příbuzných, ale odhadnout riziko a navrhnout prevenci dle rodinné anamnézy.
Využití NGS technologie a vyšetření panelů mnoha desítek genů pro hereditární nádorová onemocnění je významným posunem nejen v možnostech prevence nádorových onemocnění u vysoce rizikových osob.
Práce byla podpořena MZ ČR – RVO (MOÚ, 00209805) a Evropským fondem pro regionální rozvoj a státním rozpočtem České republiky (OP VaVpI – RECAMO CZ.1.05/2.1.00/03.0101).
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do biomedicínských časopisů.
RNDr. Eva Macháčková, Ph.D.
Oddělení epidemiologie a genetiky nádorů
Masarykův onkologický ústav
Žlutý kopec 7
656 53 Brno
e-mail: emachack@mou.cz
Obdrženo: 20. 8. 2015
Přijato: 22. 9. 2015
Sources
1. Vaughn CP, Robles J, Swensen JJ et al. Clinical analysis of PMS2: mutation detection and avoidance of pseudogenes. Hum Mutat 2010; 31(5): 588–593. doi: 10.1002/ humu.21230.
2. Kawakami T, Mitsui T, Kanai M et al. Genetic analysis of Shwachman-diamond syndrome: phenotypic heterogeneity in patients carrying identical SBDS mutations. Tohoku J Exp Med 2005; 206(3): 253–259.
3. Wu Y, Brosh RM. DNA helicase and helicase-nuclease enzymes with a conserved iron-sulfur cluster. Nucleic Acids Res 2012; 40(10): 4247–4260. doi: 10.1093/ nar/ gks039.
4. De Nicolo A, Tancredi M, Lombardi G et al. A novel breast cancer – associated BRIP1 (FANCJ/ BACH1) germ-line mutation impairs protein stability and function. Clin Cancer Res 2008; 14(14): 4672–4680. doi: 10.1158/ 1078-0432.CCR-08-0087.
5. Vaz F, Hanenberg H, Schuster B et al. Mutation of the RAD51C gene in a Fanconi anemia-like disorder. Nat Genet 2010, 42(5): 406–409.
6. Meindl A, Hellebrand H, Wiek C et al. Germline mutations in breast and ovarian cancer pedigrees establish RAD51C as a human cancer susceptibility gene. Nat Genet 2010; 42(5): 410–414. doi: 10.1038/ ng.569.
7. Clague J, Wilhoite G, Adamson A et al. RAD51C germ-line mutations in breast and ovarian cancer cases from high-risk families. PLoS One 2011; 6(9): e25632. doi: 10.1371/ journal.pone.0025632.
8. Janatova M, Soukupova J, Stribrna J et al. Mutation analysis of the RAD51C and RAD51D genes in high-risk ovarian cancer patients and families from the Czech Republic. PLoS One 2015; 10(6): e0127711. doi: 10.1371/ journal.pone.0127711.
9. Huck K, Hanenberg H, Gudowius S et al. Delayed diagnosis and complications of Fanconi anaemia at advanced age – a paradigm. Br J Haematol 2006; 133(2): 188–197.
10. Berwick M, Satagopan JM, Ben-Porat L et al. Genetic heterogeneity among Fanconi anemia heterozygotes and risk of cancer. Cancer Res 2007; 67(19): 9591–9596.
11. Kennedy RD, D’Andrea AD. DNA repair pathways in clinical practice: lessons from pediatric cancer susceptibility syndromes. J Clin Oncol 2006; 24(23): 3799–3808.
12. Lemos MC, Thakker RV. Multiple endocrine neoplasia type 1 (MEN1): analysis of 1336 mutations reported in the first decade following identification of the gene. Hum Mutat 2008; 29(1): 22–32.
13. Wu X, Platt JL, Cascalho M. Dimerization of MLH1 and PMS2 limits nuclear localization of MutLalpha. Mol Cell Biol 2003; 23(9): 3320–3328.
14. Herkert J, Niessen R, Olderode-Berends M et al. Paediatric intestinal cancer and polyposis due to bi-allelic PMS2 mutations: case series, review and follow-up guidelines. Eur J Cancer 2011; 47 : 965–982. doi: 10.1016/ j.ejca.2011.01.013.
15. Rahal EA, Henricksen LA, Li Y et al. ATM regulates Mre11-dependent DNA end-degradation and microhomology – mediated end joining. Cell Cycle 2010; 9(14): 2866–2877.
16. Edvadsen H, Tefre T, Jansen L et al. Linkage disequilibrium pattern of the ATM gene in breast cancer patients and controls; association of SNPs and haplotypes to radio-sensitivity and post-lumpectomy local recurrence. Radiat Oncol 2007; 2 : 25–33.
17. Testa JR, Cheung M, Pei J et al. Germline BAP1 mutations predispose to malignant mesothelioma. Nat Genet 2011; 43(10): 1022–1025. doi: 10.1038/ ng.912.
18. Janeway KA, Kim SY, Lodish M et al. Defects in succinate dehydrogenase in gastrointestinal stromal tumors lacking KIT and PDGFRA mutations. Proc Natl Acad Sci U S A 2011; 108(1): 314–318. doi: 10.1073/ pnas.1009199108.
19. Plon SE, Eccles D, Easton D et al. Sequence variant classification and reporting: recommendations for improving the interpretation of cancer susceptibility genetic test results. Hum Mutat 2008; 29(11): 1282–1291. doi: 10.1002/ humu.20880.
20. Easton DF, Pharoah PD, Antoniou AC et al. Gene-Panel sequencing and the prediction of breast cancer risk. N Engl J Med 2015; 372 : 2243–2257. doi: 10.1056/ NEJMsr1501341.
21. Desai AN, Jere A. Next-generation sequencing: redy for the clinics? Clin Gene 2012; 81(6): 503–510. doi: 10.1111/ j.1399-0004.2012.01865.x.
22. Hollestelle A, Wasielewski M, Martens JW et al. Discovering moderate-risk breast cancer susceptibility genes. Curr Opin Genet Dev 2010; 20(3): 268–276. doi: 10.1016/ j.gde.2010.02.009.
23. Ni Y, Zbuk KM, Sadler T et al. Germline mutations and variants in the succinate dehydrogenase genes in Cowden and Cowden-like syndromes. Am J Hum Genet 2008; 83(2): 261–268. doi: 10.1016/ j.ajhg.2008.07.011.
24. Park JY, Singh TR, Nassar N et al. Breast cancer-associated missense mutants of the PALB2 WD40 domain, which directly binds RAD51C, RAD51 and BRCA2, disrupt DNA repair. Oncogene 2014; 33(40): 4803–4812. doi: 10.1038/ onc.2013.421.
25. Machens A, Spitschak A, Lorenz K et al. Germline RET sequence variation I852M and occult medullary thyroid cancer: harmless polymorphism or causative mutation? Clin Endocrinol 2011; 75(6): 801–805. doi: 10.1111/ j.1365-2265.2011.04158.x.
Labels
Paediatric clinical oncology Surgery Clinical oncology
Article was published inClinical Oncology

2016 Issue Supplementum 1-
All articles in this issue
- Editorial
- Doporučení rozšíření indikačních kriterií ke genetickému testování mutací v genech BRCA1 a BRCA2 u hereditárního syndromu nádorů prsu a ovarií
- Syndrom hereditárního karcinomu prsu a ovarií
- Gynekologická prevence a gynekologické aspekty péče u nosiček mutací genů BRCA1 a BRCA2
- PALB2 jako další kandidátní gen pro genetické testování u pacientů s hereditárním karcinomem prsu v České republice
- Retrospektivní NGS studie u vysoce rizikových pacientů s hereditární predispozicí k nádorovému onemocnění v Masarykově onkologickém ústavu
- CZECANCA: CZEch CAncer paNel for Clinical Application – návrh a příprava cíleného sekvenačního panelu pro identifikaci nádorové predispozice u rizikových osob v České republice
- Genetika tumorigenézy nádorov kolorekta (možnosti testovania a screeningovej predikcie dedičnej formy ochorenia – Lynchovho syndrómu)
- Syndromy predisponující k nádorům v dětském věku – zkušenosti Kliniky dětské onkologie FN Brno
- Genetické syndromy predisponující k dětským nádorům centrálního nervového systému
- Hepatoblastom, etiologie, kazuistiky
- Identifikace rodiny s nosičstvím germinální delece genu SUFU na podkladě diagnózy desmoplastického meduloblastomu u batolete
- Fanconiho anémie, komplementační skupina D1 v důsledku bialelické mutace genu BRCA2 – kazuistika
- Clinical Oncology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- PALB2 jako další kandidátní gen pro genetické testování u pacientů s hereditárním karcinomem prsu v České republice
- Hepatoblastom, etiologie, kazuistiky
- Genetika tumorigenézy nádorov kolorekta (možnosti testovania a screeningovej predikcie dedičnej formy ochorenia – Lynchovho syndrómu)
- Fanconiho anémie, komplementační skupina D1 v důsledku bialelické mutace genu BRCA2 – kazuistika
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

